Нуклеотидный BLAST
Задание 1. Таксономия и функция гена
Для определения таксономии и функции данной последовательности была использована часть программного пакета BLAST - а именно алгоритм BLASTN.
По данному алгоритму были выровнены нуклеотидные последовательности. Далее представлены изображения, иллюстрирующие результаты поиска, по которым
можно сделать различные выводы.

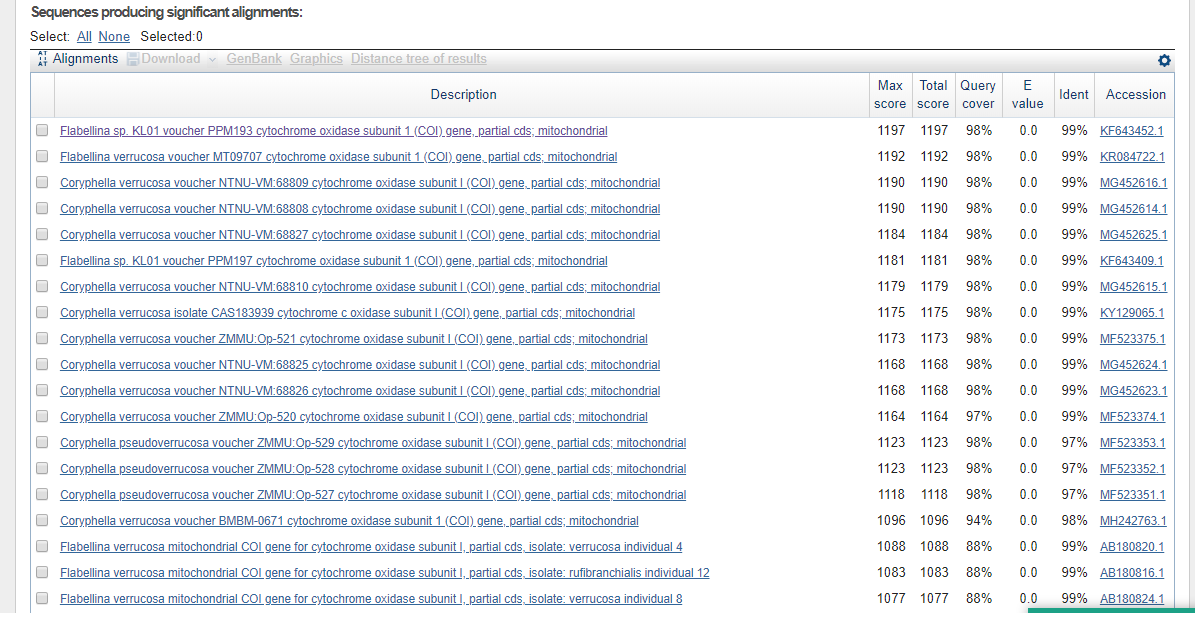
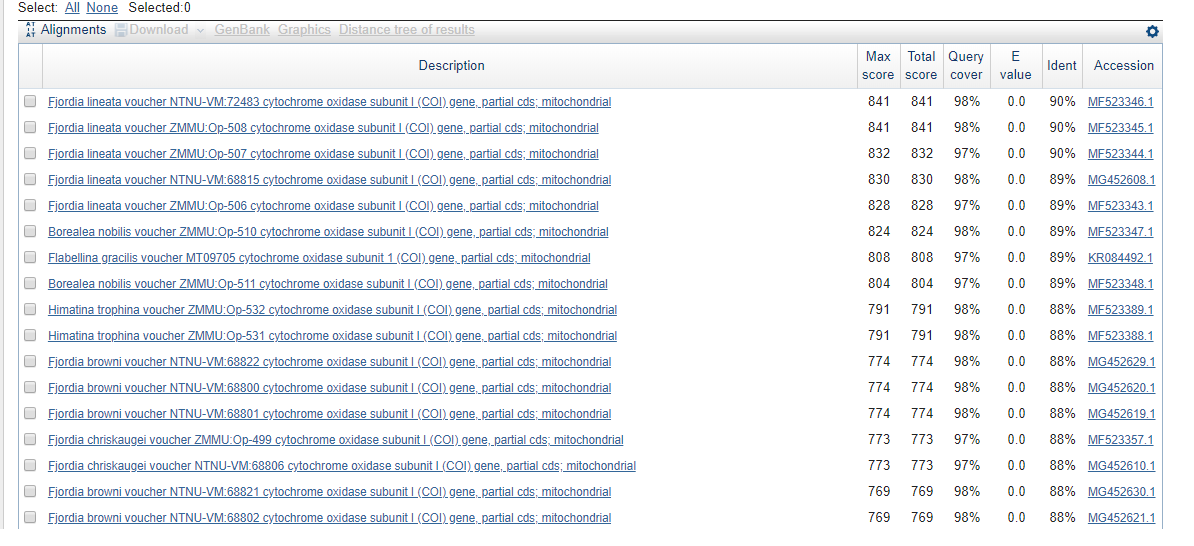
По данным результатам можно сказать, что данный организм, из которого была получена последовательность гена,
относится к виду Flabellina sp. Таксономия была выбрана по следующим признакам: лучший максимальный вес (1034),
покрытие 99%, минимальное E-value - 0.0, идентичность - 99%. Данная последовательность с большой вероятностью кодирует ген субъединицы цитохромоксидазы 1
с идентификатором KF643452.1. Данная субъединица является одной из ключевых в сборке цитохромоксидазы с - одного из важненйших ферментов
элктронтранспортной цепи митохондрий.
Таксономия
Таксон |
Название |
Тип |
Mollusca |
Класс |
Gastropoda |
Клада |
Heterobranchia |
Суперсемейство |
Flabellinoidea |
Семейство |
Flabellinidae |
Род |
Flabellina |
Задание 2. Сравнение списка находок с тремя разными вариантами BLAST
Производился поиск той же последовательностью с использованием трёх алгоритмов: blastn, discontiguous megablast, megablast. Параметры поиска:
поиск производился внутри семейств Flabellinidae (taxid:71483) и Coryphellidae (taxid:2059233) с исключением родов
Flabellina (taxid:71484) и Coryphella (taxid:2059234), использовалось ограничение по E-value: 1e-150, Длина слова варьировалась в зависимости от алгоритма поиска
(см. таблицу)
Результат поиска blastn

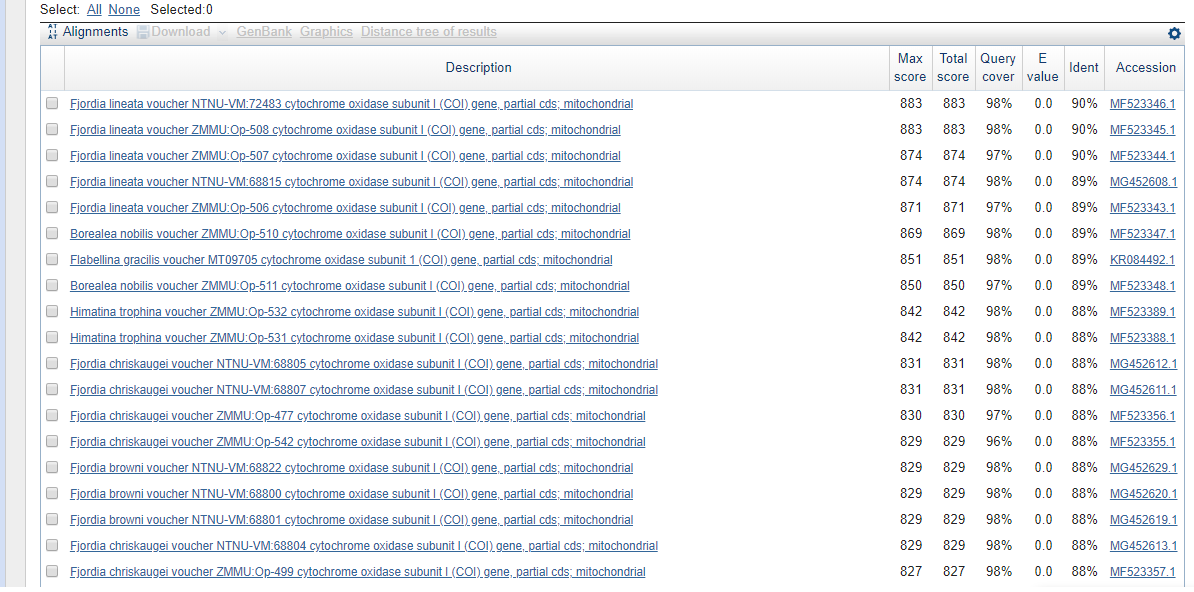
Результат поиска discontiguous megablast

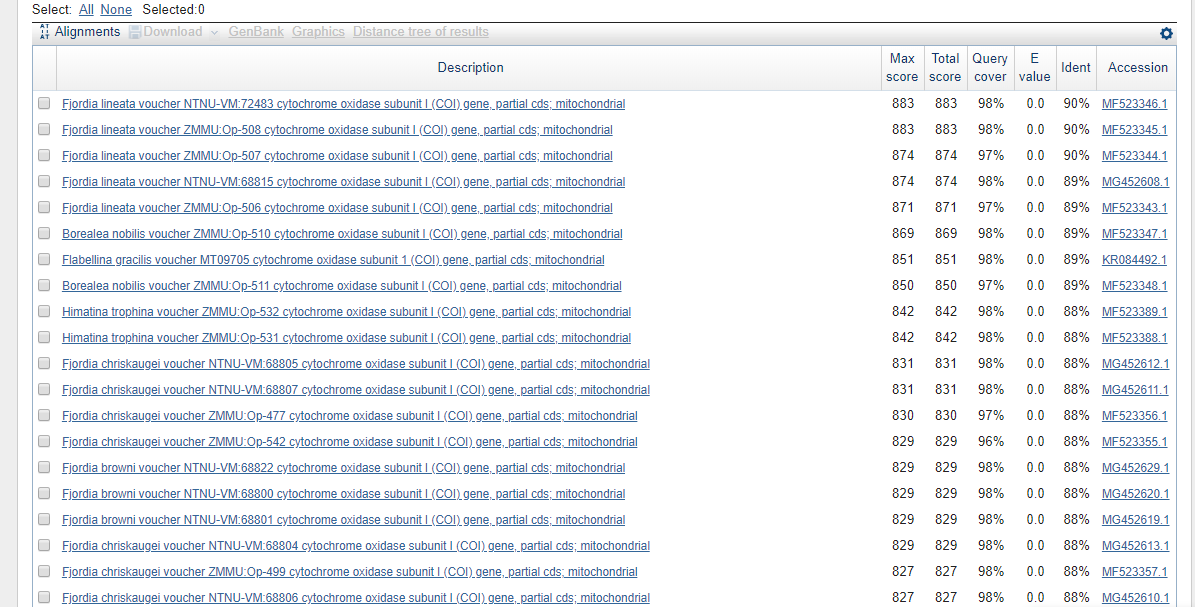
Результат поиска megablast
Ограничение длины слов и находки
Алгоритм |
Длина слова |
Число находок |
blastn |
11 |
86 |
discontiguous megablast |
11 |
86 |
megablast |
28 |
48 |
По результатам поисков с использованием трёх разных путей программа blastn, как и discontiguous megablast, дала максимальное число находок (86),
тогда как программа megablast нашла 46 последовательностей. Данное различие можно объяснить неодинаковыми стратегиями поиска, как то: megablast ищет длинные, по 28 нуклеотидов в длинну, последовательности, тогда как blastn и discontiguous megablast осуществляют поиск по подстрокам длины 11. Discontiguous megablast ищет сходство между последовательностями с низкой идентичностью и ожидалось, что результат поиска данной программы будет самым разнообразным.
Задание 3. Наличие гомологов трех белков в неаннотированном геноме
Для выполнения задания была взята сборка генома Amoeboaphelidium protococcarum - представителя таксона Афелд, родственного грибам из группы опистоконт. Афелды являются внутриклеточными паразитами или паразитоидами планктонных водорослей.
С помощью локальной версии blast - blast 2.2.31+ был произведён поиск против локально созданной из сборки базы данных. Для поиска была выбрана программа tblastn, обратно транслирующая последовательность белка в последовательность нуклеотидов ДНК и производящая поиск схожих последовательностей по этим данным.
Для выполнения задания из базы данных были скачаны белки:
HSP71_YEAST, шаперон HSP70, белок теплового шока
PRPC_EMENI, митохондриальная цитратсинтаза
TBB_NEUCR, тубулин, белок, участвующий в образовании микротрубочек
Пример команды запуска: tblastn -query hsp71_yeast.fasta -db X5.fasta -outfmt 7 > hsp71_yeast.out
Пример выдачи:
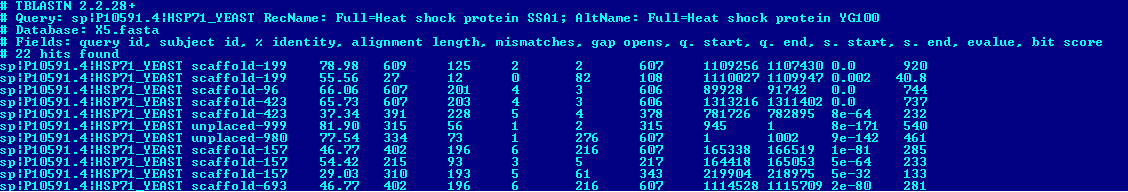
Теперь подробнее по каждому белку.
- HSP71_YEAST
В данном поиске было обнаружено немалое количество находок, лучшей из которых был scaffold-199 со значением score=920 и E-value 0.0.
Это даёт основания предполагать гомологию находки.
- PRPC_EMENI
Здесь находок было начительно меньше (всего 6), лучшим был scaffold-693 со зачением score=393 и E-value 6е-121. Это даёт основания предположить
условную гомологию находки.
- TBB_NEUCR
Среднее количество находок, у двух вообще E-value 0.0, у остальных относительно неплохой; лучшая находка - unplaced-665 со значением score=742 и E-value 0.0. Это даёт возможность
предположить о частичной гомологии белка или гомологии отдельных его доменов.
Задание 4. Ген в одном из контигов Amoeboaphelidium protococcarum
Из сборки генома Amoeboaphelidium protococcarum был извлечён скэффолд scaffold-223 длиной 76170 пар оснований. Поиск производился с использовнаием
программы blastx. По результатам проверки (приведены ниже) лучшей находкой оказалась субъеденица RPC2 фермента
ДНК-зависимая РНК-полимераза III. Выбран именно этот ген, так как для него характерен наивысший максимальный вес, E-value 0 и сходство 61%, хотя и покрытие не очень большое

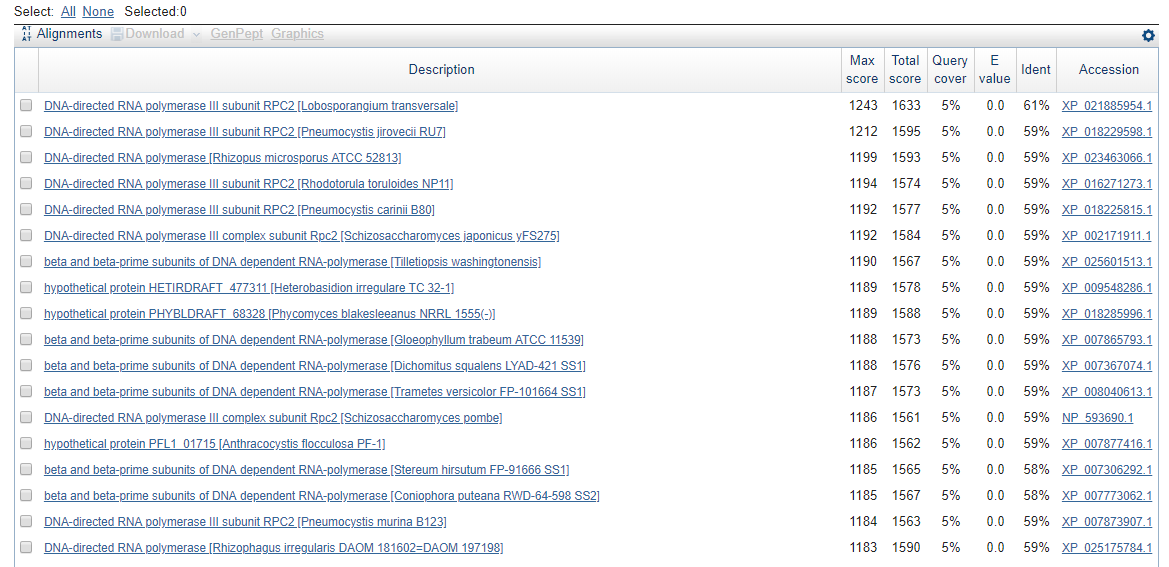
Задание 5. Карта локального сходства геномов двух бактерий
Для задания были взяты геномы двух бактерий из рода Bacillus - Bacillus cereus и Bacillus thuringiensis. Геномы двух бактерий
были сравнены путём прогонки через BLAST, а именно по алгоритму megablast. В результате получилась такая карта локального сходства:
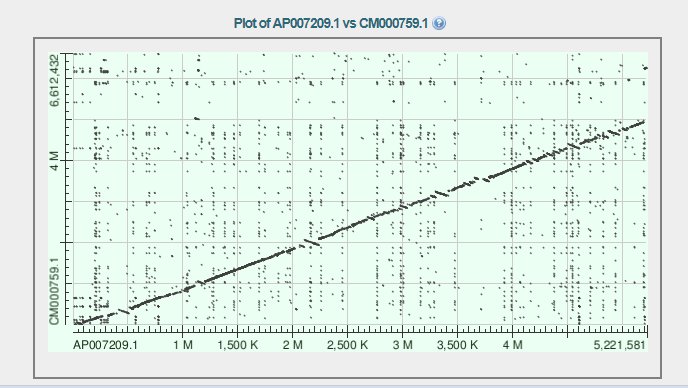
По изображению можно предположить, что в некотороых местах (где на карте короткие линии расположены под углом) могли произойти инверсии.
© Иззи Антон,2018












