De novo
Сборка генома de novo
1. Подготовка чтений
С помощью программы trimmomatic были удалены воможные участки адаптеров. Для начала нужно было распаковать загруженный файл. Команда: gunzip SRR4240388.fastq.gz.
Чтения были подготовлены, команда: fastqc SRR4240388.fastq. Далее была произведена чистка. Команда: java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240388.fastq new.fastq ILLUMINACLIP:adapters.fasta:2:7:7 TRAILING:20 MINLEN:30. И проявлена:
fastqc new.fastq. Далее представлены результаты.
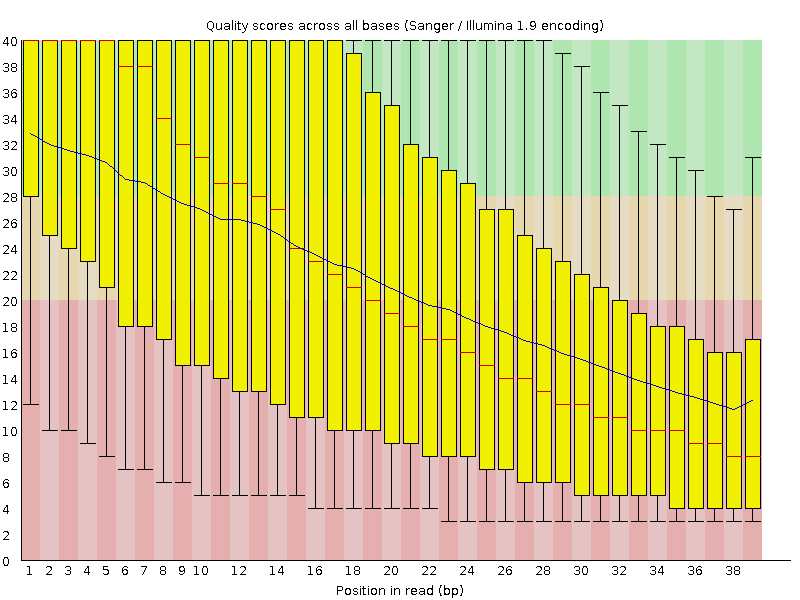
_ Изображение до чистки
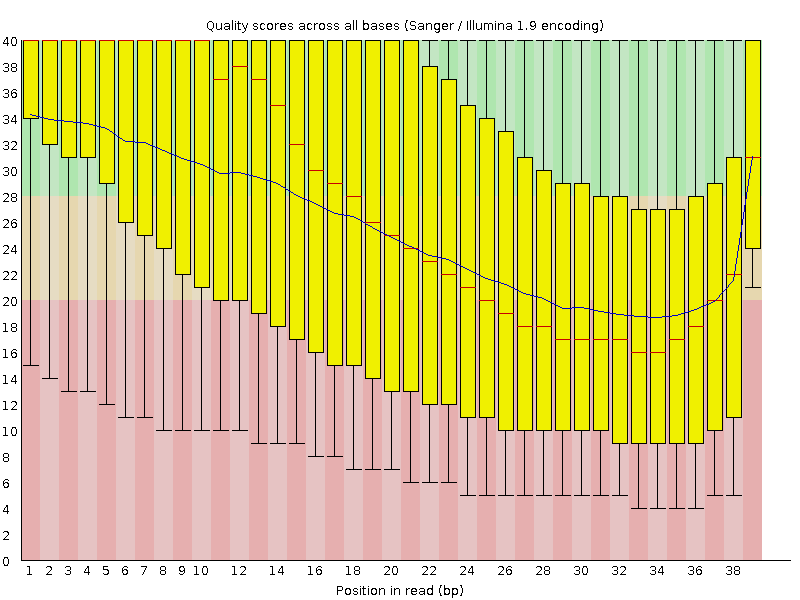
_ Изображение после чистки
_ Число чтений до чистки - 10833163, после чистки - 7336973
_ Длина чтений до чистки - 39, после чистки - 30-39
_ В итоге после чистки длиной менее 30 пар оснований было удалено 3496190 чтений
2. Создание k-меров и построение генома
Были сипользованы команды:
1_ velveth kmers 29 -fastq -short new.fastq
2_ velvetg
После завершения построения генома программа velvetg выдала N50 = 16590
Максимально длинные контиги: ID 34, длина - 16590, покрытие - 42,65; ID 41, длина - 16373, покрытие - 49,33; ID 26, длина - 14691, 55,27.
Аномальные контиги: контиг с ID 724 имеет самое высокое покрытие 1141,94 при длине 50; контиг с ID 781 имеет самое низкое покрытие 1,41 при длине 29.
3. Анализ megablast
ID 34
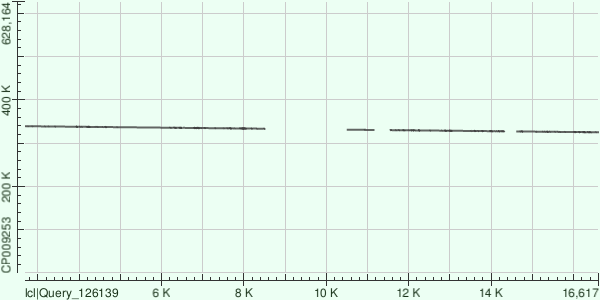
Характеристика всего выравнивания
Total score = 5762, Query cover = 67%, E-value = 0, Ident = 76%
Контиг соответствует учатску хромосомы с координатами [324944;339010]
Всего было получено 4 выравнивания со следующими координатами:
[324944;326950]
[327227;330003]
[330333;331006]
[333222;339010]
Все эти выравнивания имеют направленность (+/-) и Expect 0 (только предпоследний - 1e-170)
Процент гэпо варируется от 0 до 3, Identities - от 75 до 83
В целом можно сказать, что контиг практически полностью лёг на геном в обратном направлении, хотя в некоторых участках есть отличия
ID 41
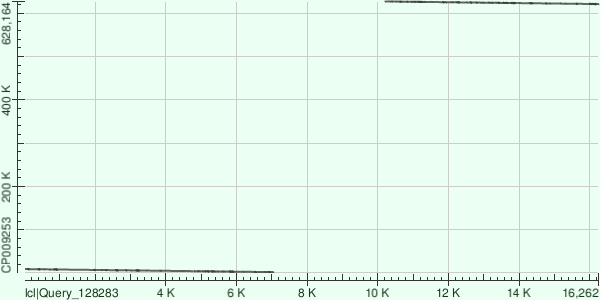
Характеристика всего выравнивания
Total score = 7344, Query cover = 79%,E-value = 0 ,Ident = 78%
Контиг соответствует учатску хромосомы с координатами [2004;627104]
Всего было получено 2 выравнивания со следующими координатами:
[2004;9041]
[621055;627104]
Все эти выравнивания имеют направленность (+/-) и Expect 0
Пороцент гэпов варируется от 2 до 3, Identities - от 76 до 78
В целом можно сказать, что контиг имеет высокое сходство с участками, только эти участки сильно удалены друг от друга
ID 26
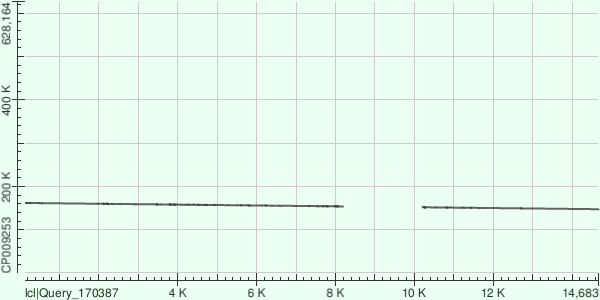
Характеристика всего выравнивания
Total score = 8301, Query cover = 85%,E-value = 0,Ident = 78%
Контиг соответствует учатску хромосомы с координатами [147305;161738]
Всего было получено 2 выравнивания со следующими координатами:
[147305;151796]
[153752;161738]
Все эти выравнивания имеют направленность (+/-) и Expect 0
Процент гэпов варируется от 1 до 3, Identities - от 78 до 81
Контиг впринципе почти полность лёг на геном в обратном направлении, хотя и есть одна небольшая "брешь"
© Иззи Антон,2018





