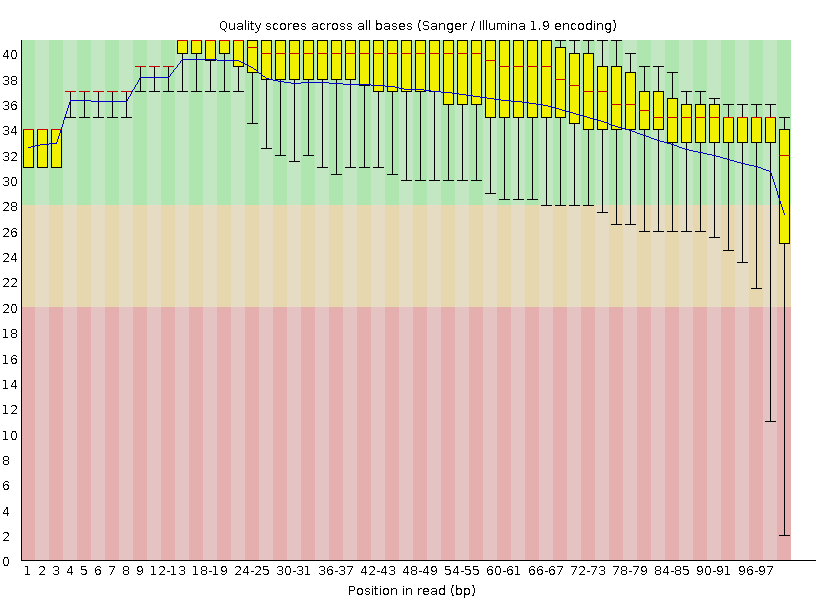

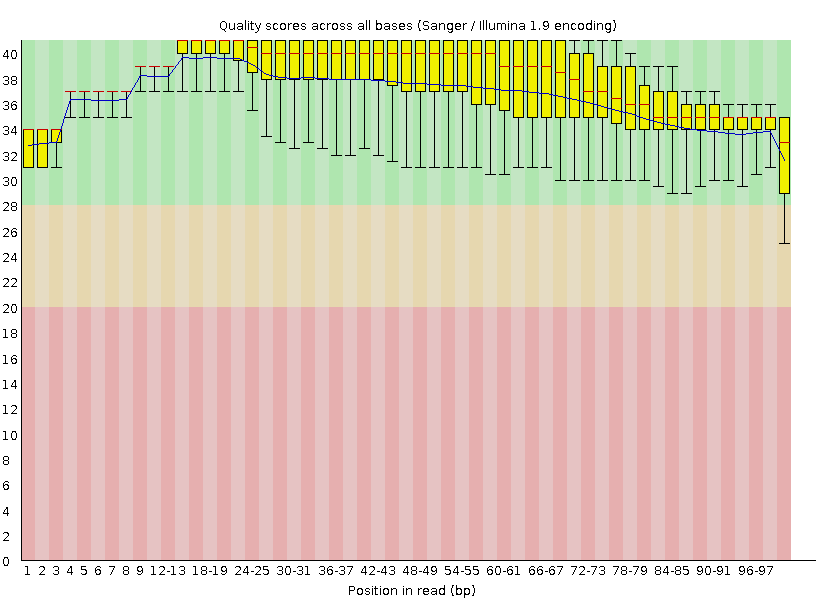
Координаты |
Референсная последовательность |
Чтения |
Тип полиморфизма |
Глубина покрытия |
Качество чтений |
| 21024619 | A | G | Замена нуклеотида | 37 | 221.999 |
| 81467864 | CAT | C | Делеция | 7 | 217.468 |
| 81449335 | C | G | Замена нуклеотида | 65 | 225.009 |
| Координата | SNP | Quality и DP | refgene | dbsnp | 1000 genomes | Gwas | Clinvar |
| 21024619 | A G | 221.999 37 | exonic RNASE9 hom | rs1243647 | 0.752995 | Prostate cancer (gene x gene interaction) | |
| 21026773 | C T | 225.009 42 | splicing RNASE9(NM_001110361:exon4:c.1+1G> A,NM_001110358:exon3:c.1+1G>A) het | rs891297 | 0.313698 | ||
| 81448224 | C T | 26.0194 3 | intronic TSHR het | rs74064786 | 0.0632987 | ||
| 81448282 | C G | 91.0145 5 | intronic TSHR het | rs3783950 | 0.515775 | ||
| 81448382 | T G | 148.134 6 | intronic TSHR hom | rs3783949 | 0.505591 | ||
| 81448782 | T C | 225.009 53 | intronic TSHR het | rs12590262 | 0.196486 | ||
| 81449335 | C G | 225.009 65 | intronic TSHR het | rs10139168 | 0.063099 | ||
| 81449534 | T C | 221.999 24 | intronic TSHR hom | rs3783947 | 0.432308 | ||
| 81449577 | T G | 225.009 32 | intronic TSHR het | rs3783946 | 0.195487 | ||
| 81449939 | A G | 212.009 21 | intronic TSHR het | rs11847808 | 0.063099 | ||
| 81450245 | C T | 77.0075 8 | intronic TSHR het | rs11850795 | 0.0632987 | ||
| 81450590 | C T | 40.0075 9 | intronic TSHR het | rs11850934 | 0.0632987 | ||
| 81450743 | G A | 225.009 26 | intronic TSHR het | rs2284723 | 0.186502 | ||
| 81450967 | C T | 12.272 8 | intronic TSHR het | rs74064794 | 0.0628994 | ||
| 81451009 | G T | 46.0072 18 | intronic TSHR het | rs74064795 | 0.00599042 | ||
| 81451052 | C T | 221.999 35 | intronic TSHR hom | rs12101255 | 0.415136 | ||
| 81451102 | G A | 225.009 51 | intronic TSHR het | rs74064796 | 0.227236 | ||
| 81451229 | C T | 221.999 71 | intronic TSHR hom | rs12101261 | 0.415335 | Graves' disease | |
| 81451454 | T C | 3.54318 2 | intronic TSHR het | rs10140745 | 0.0632987 | ||
| 81451460 | G A | 6.19886 2 | intronic TSHR het | rs10151744 | 0.0638978 | ||
| 81451956 | G A | 222.003 11 | intronic TSHR hom | rs8003061 | 0.415136 | ||
| 81452172 | G A | 32.7667 2 | intronic TSHR hom | rs17111346 | 0.414936 | ||
| 81462283 | T C | 11.3429 1 | intronic TSHR hom | rs1023586 | 0.427516 | ||
| 81467335 | A G | 69.9724 3 | intronic TSHR hom | rs2268462 | 0.183906 | ||
| 81467450 | A G | 74.0075 10 | intronic TSHR het | rs2268463 | 0.183906 | ||
| 81467594 | C A | 105.008 7 | intronic TSHR het | rs2268464 | 0.183906 | ||
| 81467614 | C G | 106.008 9 | intronic TSHR het | rs2268465 | 0.17492 | ||
| 81467860 | T C | 156.008 1 | intronic TSHR het | ||||
| 81468476 | T C | 11.3429 1 | intronic TSHR hom | rs58266067 | 0.361821 | ||
| 81468579 | G A | 9.52546 1 | intronic TSHR hom | rs58241131 | 0.356629 | ||
| 81470024 | A G | 50.0072 5 | intronic TSHR het | rs55960644 | 0.357628 | ||
| 81470054 | T A | 37.7652 2 | intronic TSHR hom | rs55957493 | 0.357628 | ||
| 81471277 | C G | 11.3429 1 | intronic TSHR hom | rs3783944 | 0.164736 | ||
| 81478781 | G C | 5.46383 1 | intronic TSHR hom | rs113158029 | |||
| 81490813 | G A | 9.52546 1 | intronic TSHR hom | rs146358415 | 0.0706869 | ||
| 81490842 | T C | 7.79993 1 | intronic TSHR hom | rs11159482 | 0.921925 | ||
| 81495039 | C T | 6.20226 1 | intronic TSHR hom | rs7161100 | 0.114217 | ||
| 81513417 | C A | 9.52546 1 | intronic TSHR hom | ||||
| 81523495 | C A | 7.79993 1 | intronic TSHR hom | ||||
| 81528412 | T A | 221.999 27 | intronic TSHR hom | rs17111401 | 0.327676 | ||
| 81549595 | G A | 11.3429 1 | intronic TSHR hom | rs8019570 | 0.26238 | ||
| 81557279 | G T | 38.7651 2 | intronic TSHR hom | rs10147011 | 0.286542 | ||
| 81558640 | C G | 10.4247 1 | intronic TSHR hom | rs2075175 | 0.29972 | ||
| 81558806 | C T | 221.999 61 | intronic TSHR hom | rs2075176 | 0.29972 | ||
| 81558965 | A G | 221.999 70 | intronic TSHR hom | rs2241119 | 0.29972 | ||
| 81561425 | G A | 4.13164 1 | intronic TSHR het | rs10142999 | 0.30012 | ||
| 81562808 | A G | 5.46383 1 | intronic TSHR hom | rs2075178 | 0.30012 | ||
| 81562998 | T C | 222.791 18 | exonic TSHR hom | rs2075179 | 0.30012 | ||
| 81566846 | C T | 11.3429 1 | intronic TSHR hom | ||||
| 81574283 | T A | 6.98265 1 | intronic TSHR hom | rs11845715 | 0.833666 | ||
| 102258329 | C T | 9.52546 1 | intronic PPP2R5C hom | ||||
| 102298202 | T C | 221.999 57 | intronic PPP2R5C hom | rs10152015 | 0.057508 | ||
| 102314040 | G A | 3.01618 1 | intronic PPP2R5C het | rs4589480 | 0.136182 | ||
| 102314069 | A G | 7.79993 1 | intronic PPP2R5C hom | rs1678003 | 0.241014 | ||
| 102321657 | A G | 42.7648 2 | intronic PPP2R5C hom | rs1746588 | 0.168131 | ||
| 102329228 | C G | 5.46383 1 | intronic PPP2R5C hom | rs1746585 | 0.165935 | ||
| 102333854 | C T | 10.4247 1 | intronic PPP2R5C hom | rs111453025 | 0.0563099 | ||
| 102334091 | A G | 8.64911 1 | intronic PPP2R5C hom | rs2476516 | 0.373802 | ||
| 102346180 | G A | 6.20226 1 | intronic PPP2R5C hom | rs2749908 | 0.401558 | ||
| 102348268 | G A | 161.003 11 | intronic PPP2R5C hom | rs1741157 | 0.377796 | ||
| 102348274 | T C | 158 13 | intronic PPP2R5C hom | rs1741158 | 0.247204 | ||
| 102348358 | A G | 186.999 32 | intronic PPP2R5C hom | rs1677990 | 0.237021 | ||
| 102349540 | C T | 158.012 22 | intronic PPP2R5C hom | rs1741125 | 0.376997 | ||
| 102349542 | G T | 158.009 22 | intronic PPP2R5C hom | rs1741126 | 0.376997 | ||
| 102349907 | A G | 221.999 81 | intronic PPP2R5C hom | rs2720207 | 0.378395 | ||
| 102351646 | T C | 6.20226 1 | intronic PPP2R5C hom | rs7140798 | |||
| 102351705 | G C | 3.56185 2 | intronic PPP2R5C het | rs60265551 | 0.147963 | ||
| 102353323 | C T | 8.64911 1 | intronic PPP2R5C hom | ||||
| 102356475 | C T | 221.999 47 | intronic PPP2R5C hom | rs1904298 | 0.128794 | ||
| 102359164 | G C | 162 14 | intronic PPP2R5C hom | rs2720221 | 0.38139 | ||
| 102359304 | A G | 219.009 35 | intronic PPP2R5C het | rs2720220 | 0.133387 | ||
| 102359489 | A G | 221.999 37 | intronic PPP2R5C hom | rs2720219 | 0.305711 | ||
| 102359568 | A G | 104.008 15 | intronic PPP2R5C het | rs2749902 | 0.133786 | ||
| 102360745 | T C | 225.009 46 | intronic PPP2R5C het | rs7142002 | 0.112819 | Autism | |
| 102360916 | G A | 148.008 46 | intronic PPP2R5C het | rs2251246 | 0.196086 | ||
| 102361064 | A C | 24.0241 5 | intronic PPP2R5C het | rs7145448 | 0.109026 | ||
| 102361134 | G A | 11.3429 1 | intronic PPP2R5C hom | rs7145940 | 0.123403 | ||
| 102361400 | C T | 38.7651 2 | intronic PPP2R5C hom | rs1741134 | 0.341853 | ||
| 102361737 | G A | 10.4247 1 | intronic PPP2R5C hom | rs59070929 | 0.11262 | ||
| 102364006 | G C | 6.98265 1 | intronic PPP2R5C hom | rs8005273 | 0.134784 | ||
| 102369553 | G A | 3.01618 1 | intronic PPP2R5C het | rs1678035 | 0.381789 | ||
| 102375658 | T C | 37.0187 4 | intronic PPP2R5C het | rs1616161 | 0.170527 | ||
| 102376212 | G T | 84.0932 7 | intronic PPP2R5C het | rs1612684 | 0.354034 | ||
| 102379870 | T C | 11.3429 1 | intronic PPP2R5C hom | rs6575883 | 0.150759 | ||
| 102383175 | G A | 38.7651 2 | intronic PPP2R5C hom | rs60089041 | 0.174521 | ||
| 102391577 | G C | 221.999 23 | exonic PPP2R5C hom | rs3742424 | 0.176717 | ||
| 102392060 | G A | 125.008 24 | UTR3 PPP2R5C(NM_001161726:c.*451G>A,NM_001161725:c.*451G> A,NM_002719:c.*451G>A,NM_178586:c.*451G>A) het | rs11553289 | 0.0297524 |
© Иззи Антон,2018