В данном задании был выдан PDB-файл с приблизительными координатами какого-то белка, практически без какой-либо информации о координатах лигандов или кофакторов. В таком случае есть ресур под названием PDBeFold, который поможет нам установить по схожим хитам что это за белок, какая у него приблизительная функция, какие и в каком примерно месте он присоединяет лиганды или кофакторы и прочую полезную информацию.
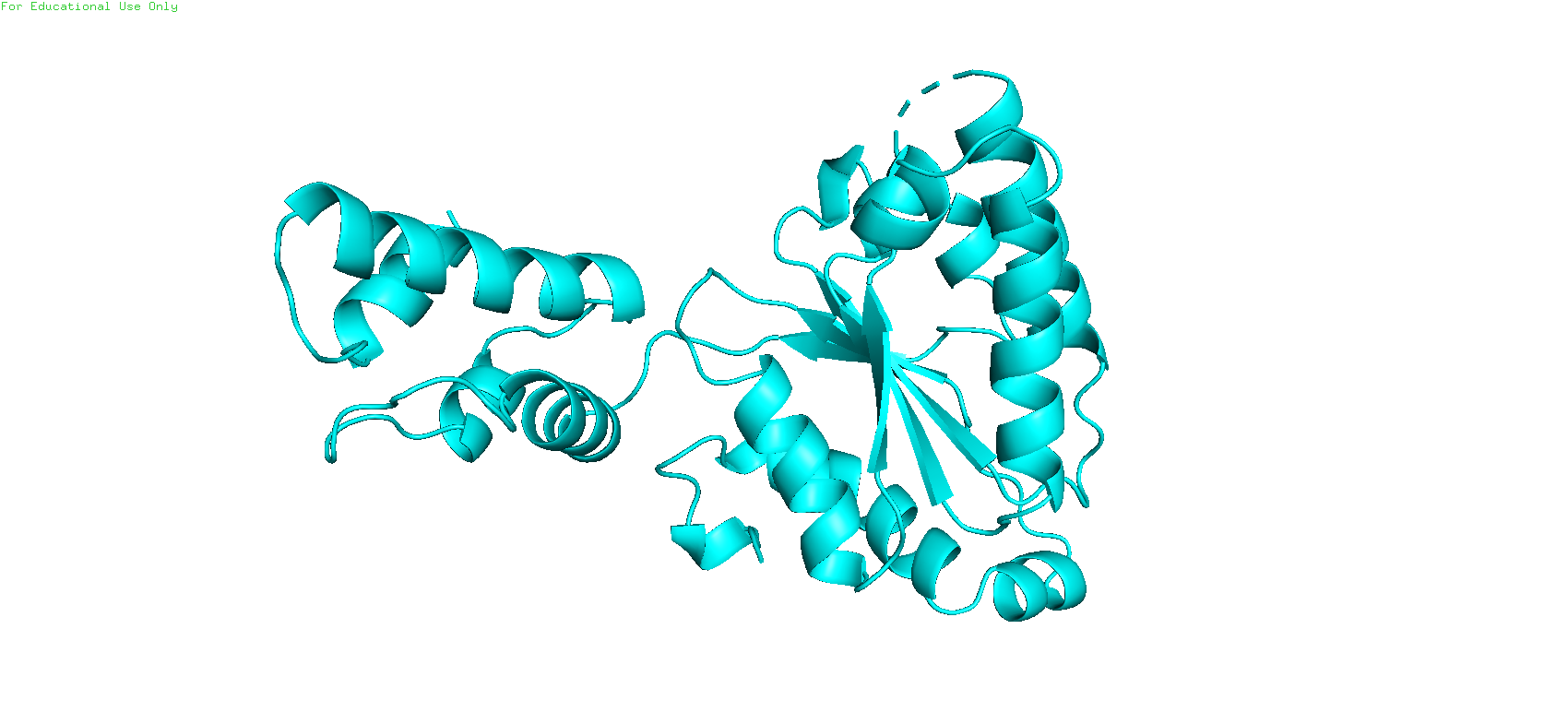
При первом взгляде на струтуру создаётся ощущение, что она состоит из двух доменов. Можно видеть, что из вторичных структур в ней преобладают альфа-спирали, бета-лист всего один. Также заметно, что он представляет собой укладку Россмана (что потом и будет подтверждено в базе данных CATH).

Рис. 1. Структура исследуемого белка
В результате поиска в PDBeFold было установлено, что исследуемый белок больше всего похож на АТФазный домен протеазы FTSH из Thermus thermophilus. Выглядит он следующим образом:
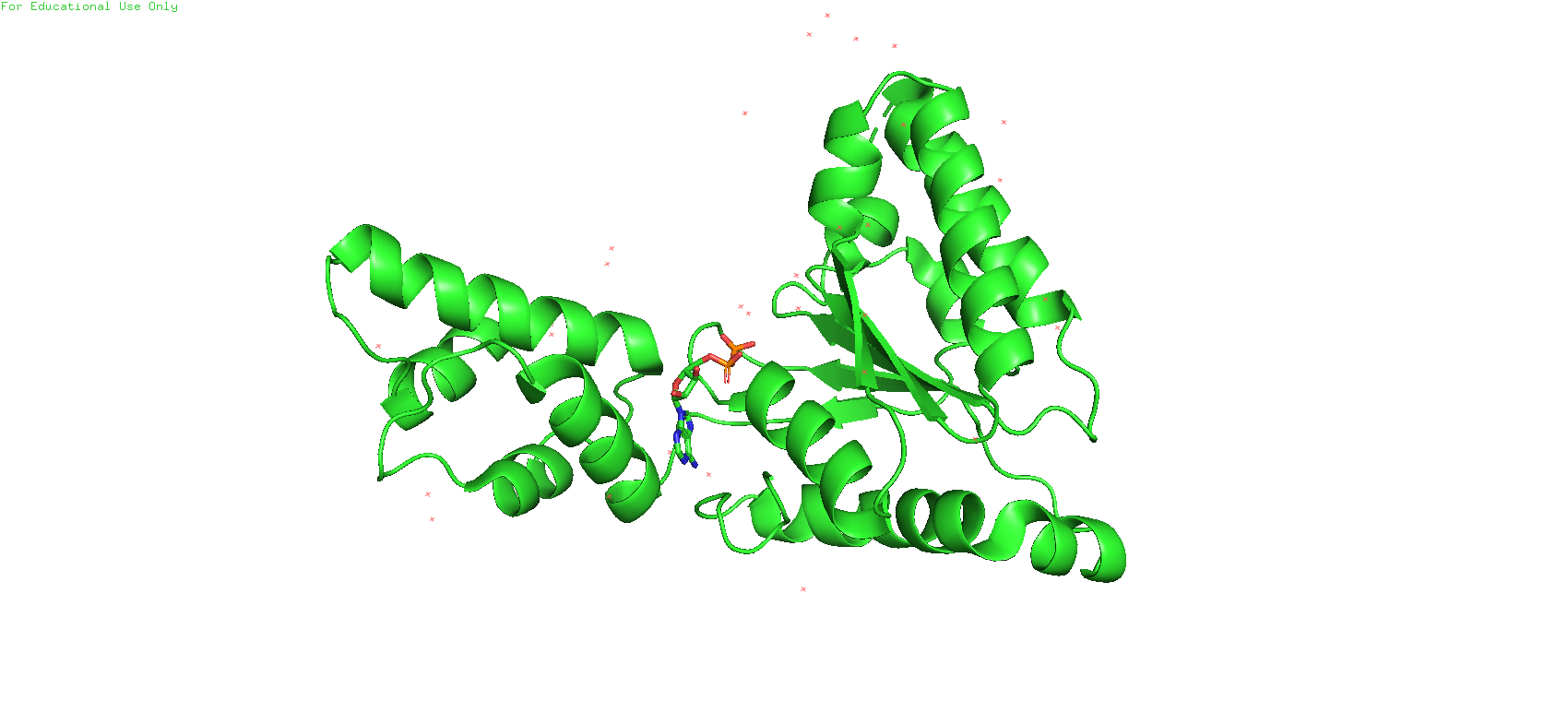
Рис. 2. АТФазный домен протеазы FTSH, можно заметить АДФ в качестве лиганда
В данной структуре также можно наблюдать довольно большое количество альфа-спиралей и всего один бета-лист. Также можно видеть, что лиганд располагается в некотором подобии "кармашка", составленного из 3-х альфа-спиралей и частично из двух петель. PDB_ID - 1IY1.
Если "пристроить" лиганд к нашему исследуемому белку, то он встаёт в том же участке, где и в найденной структуре.
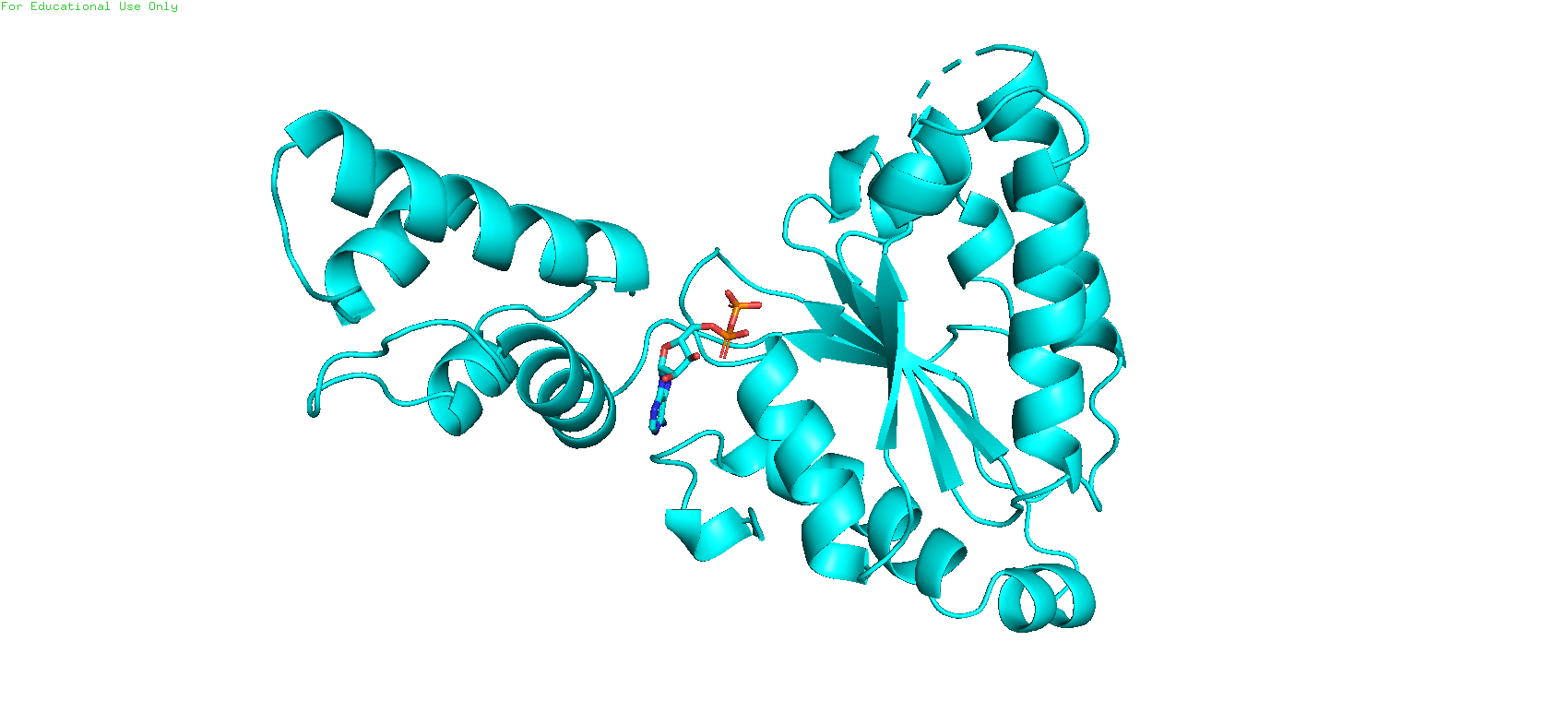
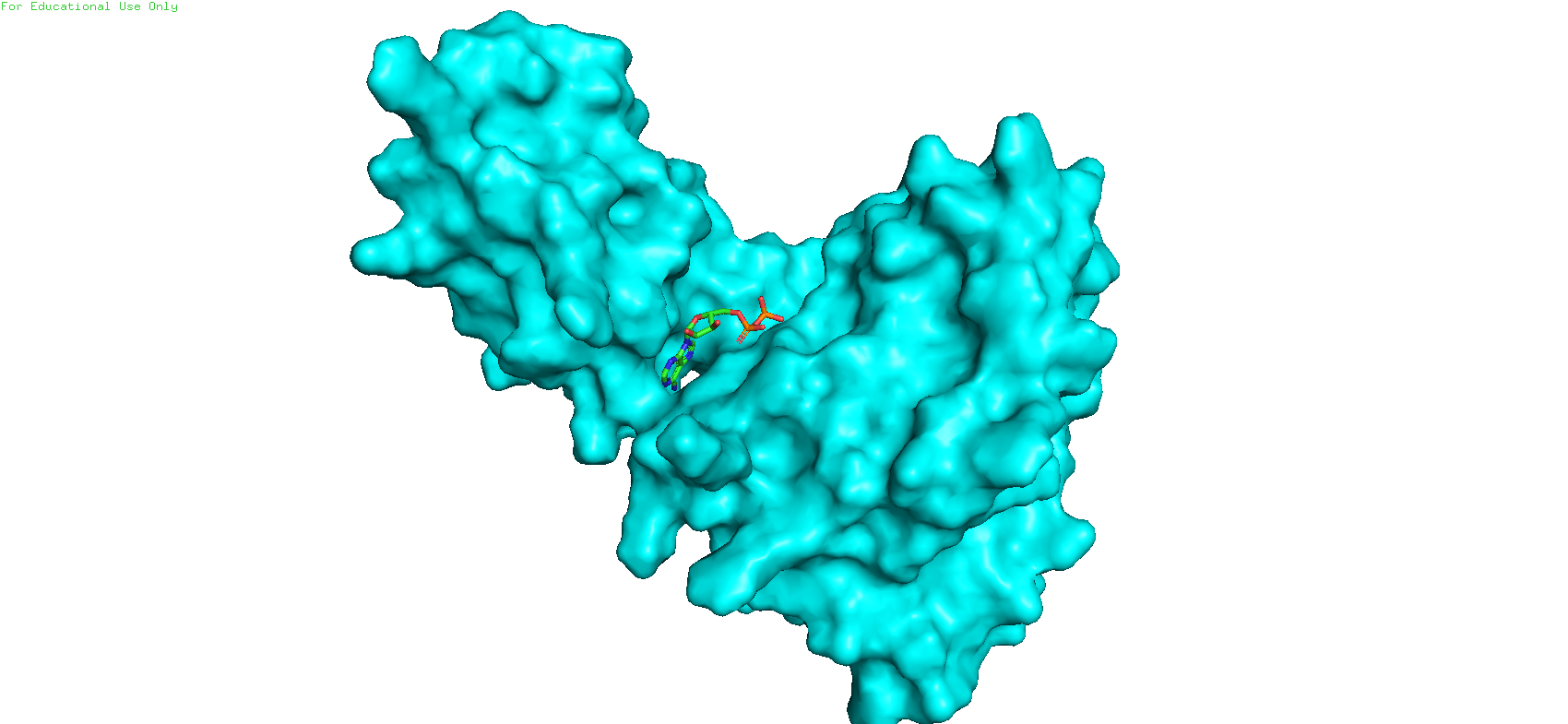
Рис. 3. Присоединение лиганда к исследуемой структуре
Далее в результате поиска по базе данных CATH было установлено, что данный белок по своей доменной организации относится к суперсемейству 3.40.50.300, или P-loop-содержащая нуклеозидтрифосфат гидролаза. Если смотреть просто по классификации CATH Superfamily, то он несёт номер суперсемейства 1.10.8.60, без расшифровки названия.
В результате поиска по литературе была найдена статья, в которой охарактеризована структура протеазы FTSH. Для установления доменной организации данного белка он был обработан трипсином, и далее был извлечён коровый фрагмент весом 27 кДа (при общем весе белка в 56 кДа). Было установлена, что этот коровый фрагмент простирается от валина-146 до лизина-403, и полностью покрывает АТФазный домен, предсказанный по выравниваниям с другими членами ААА АТФаз. Т.о. протеазный и АТФазный домены удалось выделить отдельно, несмотря на то что они работают в довольно тесной связке.
АТФазный домен состоит из двух субдоменов: большой N-концевой и маленький С-концевой. N-концевой субломен состит из центрального бета-листа, составленного из пяти параллельных бета-тяжей, окружённых восемью альфа-спиралями. Малый субдомен состоит из четырёх альфа-спиралей и короткой, бета-лист подобной структуры.
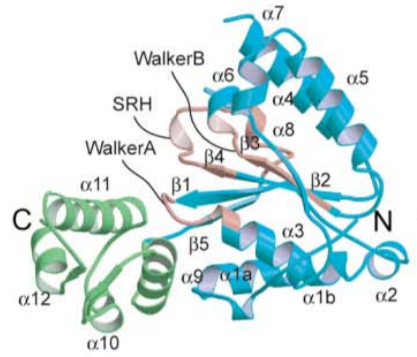
Рис. 4. "Субдоменная" организация АТФазного домена протеазы FtsH (извинения за плохое качество)
Было показано, что лиганды (ADP или AMP-PNP) связываются с Walker A мотивом, располагающимся между β1 и α3. При этом ни в одной кристаллической структуре не было показано наличие каталитически важного иона магния, однако вполне вероятно, что данный ион координируется тирозином-203, а аспартат-225 связывается с ним через молекулу воды, по аналогии с другими нуклеозидтрифосфатазами с Walker A мотивом.
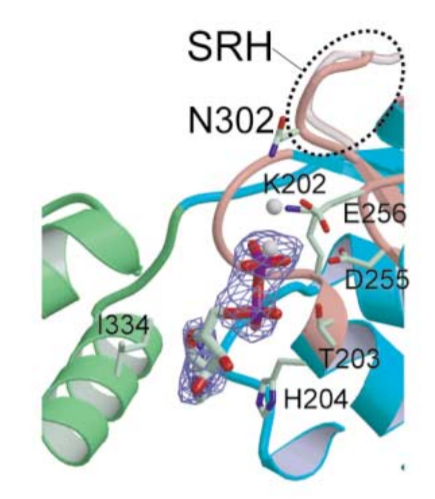
Рис. 5. Лиганд в связке с Walker A мотивом
Картинки рисунков 4 и 5 взяты из статьи с doi: https://doi.org/10.1016/S0969-2126(02)00855-9
Теперь нужно модифицировать поиск в PDBefold. Были убраны в процессе поиска некоторые параметры, и выбраны два белка, которые по последовательности схожи с исходным менее чем на 35%. Это белки с PDB_ID 1nsf (сходство 19%) и 4w5w (сходство 14%).
Данный белок также обладает АТФазной активностью, однако несёт совершенно другую функцию. Если предыдущий фермент был протеазой, то этот является белком, участвующим в связывании мембран. Называется N-ethylmaleimide-sensitive factor, или NSF. С исходным белком у него было 19% сходства, и при первом взгляде их структуры кажутся довольно похожими, особенно в глаза бросается тот самый едиснтвенный бета-лист, составленный из 5 параллельных бета-тяжей. Также вполне можно видеть и нуклеотидсвязывающий Walker A мотив (на рисунке выделен оранжевым). Что ещё интересно, в данной структуре отображён ион магния. Из некоторых изменений также стоит отметить, что одна из петель стала номного длиннее (отмечена циановым), как бы ещё сильнее "загораживая" лиганд.
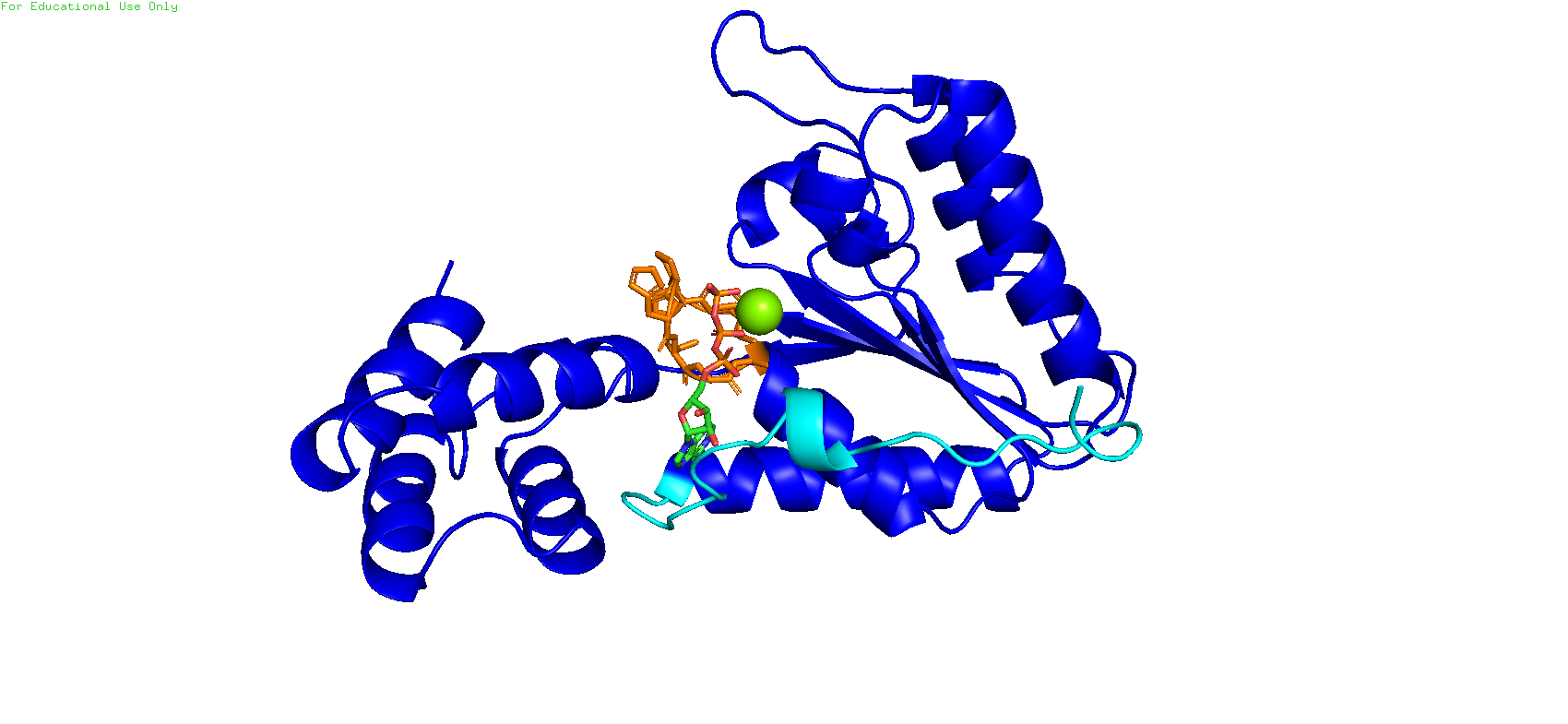
Рис. 6. Отображение структуры с PDB_ID 1nsf
По классификации в базе данных CATH данный белок относится к тому же самому суперсемейству, что и исходный - 1.10.8.60, да и в целом общая классификация идентична.
В данном случае попался моторный белок, который выполняет функцию активатора РуБисКо, вызывая его конформационные изменения с целью выхода из неактивного состояния. Наблюдаются некоторые отличия в структуре, в особенности альяа-спиралей С-концевого субдомена, однако присутствует тот же единственный бета-лист с пятью параллельнвми тяжами. Данный белок также является АТФазой, однако в данной структуре в качестве лигандов было отмечено только две молекулы сульфат-ионов, скорее всего выполняющие роль кофакторов.
причём здесь также можно видеть Walker A мотив (отмечен красным), хотя кажется что сайт связывания выглядит немного иначе, нежели в исходной структуре.
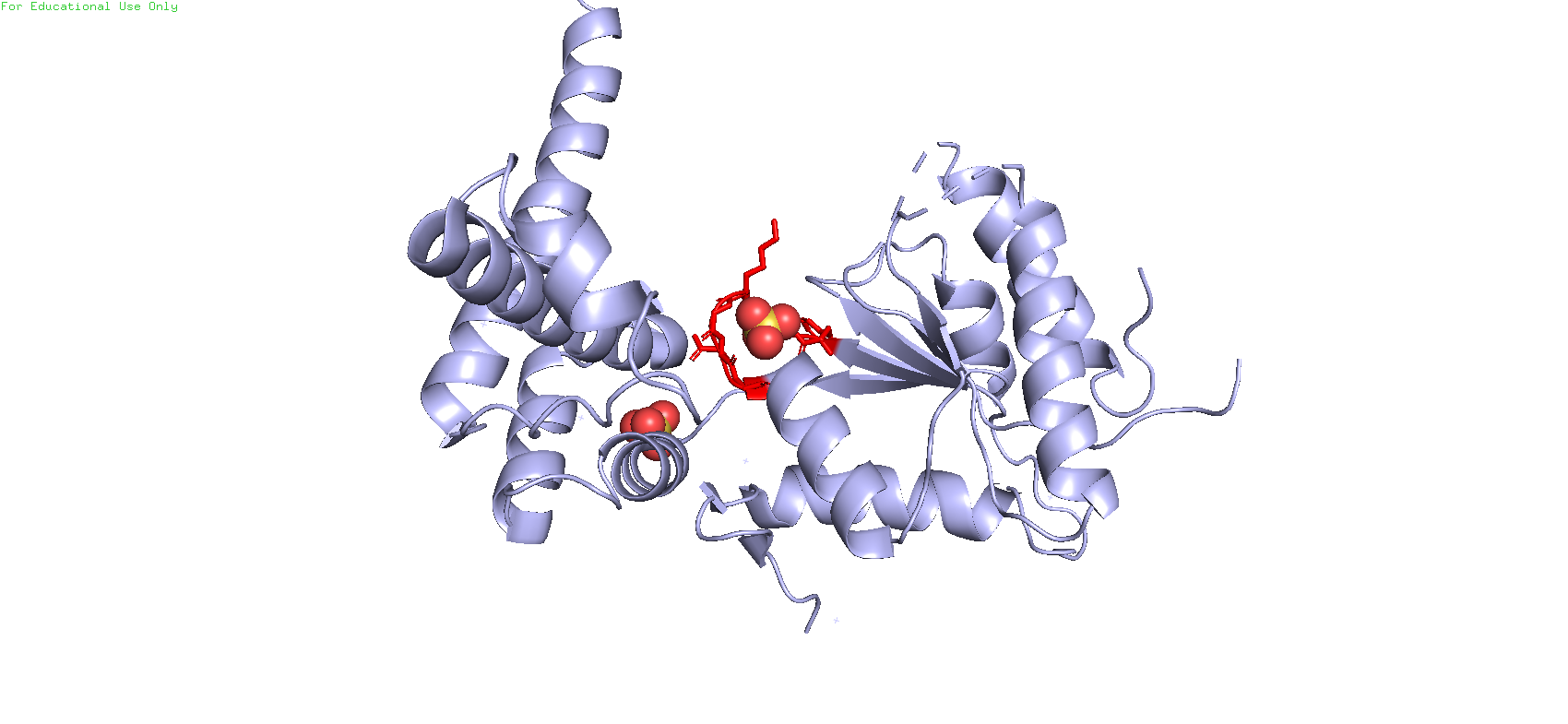
Рис. 7. Отображение структуры с PDB_ID 4w5w
Однако в этот раз классификация подобранного белка в базе данных CATH несколько отличается от таковой у исходного белка. Рассмотренный белок принадлежит к суперсемейству 1.10.8.10, или DNA helicase RuvA subunit, C-terminal domain, а в остальном классификация схожа.
Все эти три белка объединяет по сути одна общая функция - АТФазная, гидролиз АТФ. Однако их гланвные, "рабочие" функции всё же отличаются. FtsH, как говорилось ранее, является протеазой, её основная работа - уничтожать некоторые белки цитоплазматической мембраны, несущих С-концевой сигнал к деградации, вследствие чего FtsH может регулировать общее "качество" белков мембраны; по классификации в базе данных KEGG данная протеаза носит номер EC:3.4.24.-. В случае же NSF, основная функция данного фермента заключается в расплетании белков комплекса SNARE, тем самым обеспечивая завершение процесса слияния мембранных пузырьков; классифицируется данный фермент как EC 3.6.4.6, и называется Vesicle-fusing ATPase.
РуБисКо активаза облегчает карбамилирование РуБисКо в присутствии рибулозобисфосфата, и таким образом снимает его ингибирование, но основная функция - это удаление различных сахарофосфатов, в т.ч. и рибулозобисфосфата, из активного центра РуБисКо чтобы последний мог присоединить СО2 и продолжить катализ. Основная информация по данному ферменту была взята из следующей статьи: https://doi.org/10.1007/BF00017748. Была найлена такде ещё одна статья, которая вполне могла раскрыть и некоторые структурные особенности РуБисКо активазы, не исклюсая и специфические функциональные мотивы, но к сожалению её DOI оказался то ли нерабочим, то ли недействительным (на всякий случай оставлб его здесь: https://doi.org/10.1096/fasebj.27.1_supplement.580.5)
Рассмотрим же структуры этих белков подробнее. Среди них можно вполне возможно выделить один функциональный домен, общий у всех и который отвечает за осуществление их основной функции - это P-loop (он же walker A motif, на рисунках окрашен фиолетовым), и ферменты с таким мотивом называются P-loop containing nucleoside triphosphate hydrolases. Основная реакция, осуществляемая белками с данным мотивом - это собственно гидролиз бета-гамма фосфатной связи в нуклеозидтрифосфатах. Данный P-loop собственно и ответственен за связывание фосфата (бета). Для него характерен особый паттерн - G-x(4)-GK-[TS], где G - глицин, x(4) - любые 4 аминокислоты, К - лизин, [TS] - треонин и серин. Лизин вместе с NH-группами остова является важным звеном в связывании нуклеотида.
Также стоит отметить и наличие Walker B мотива (на рисунках отмечен жёлтым), который в случае с РуБисКо активазой выглядит немного иначе, чем в первых двух. Данный мотив может быть довольно разнообразным по своему аминокислотному набору, однако объединяется это всё в одну черту - наличие отрицательно заряженного остатка (аспартат или глутамат) вместе с тяжом гидрофобных остатков. В таком мотиве чаще всего аспатрат отвечает за координацию иона магния, а глутамат за гидролиз АТФ.
Что же касается поиска специфических функциональных мотивов, то в данном случае не всё оказалось так гладко. Даже несмотря на то, что данные фермента относятся к разным классам по EC (РуБисКо активаза вообще шаперон, если верить классификации в PDB) каких-либо действительно интересных мотивов, которые как-то по-разному могли бы участвовать в процессировании АТФ или связывать какие-либо другие лиганды (можно попробовать сделать исклбчение по поводу РуБисКо активазы, где в структуре были изображены молекулы сульват-аниона, одна из которых находится в "объятиях" P-loop и в непосредственной близости от аспартата Walker B мотива) обнаружены не было, и литература также по этому поводу умалчивает. Можно впринципе отметить в структуре РуБисКо активазы часть петли (отмечена оранжевым), расположенной пространственно ниже P-loop, в которой находится некоторое количесвто ароматики. Такое расположение вблизи активного центра может говорить о том, что данный участок мог бы прочнее удерживать в нём молекулу АТФ, однако это только предположение.
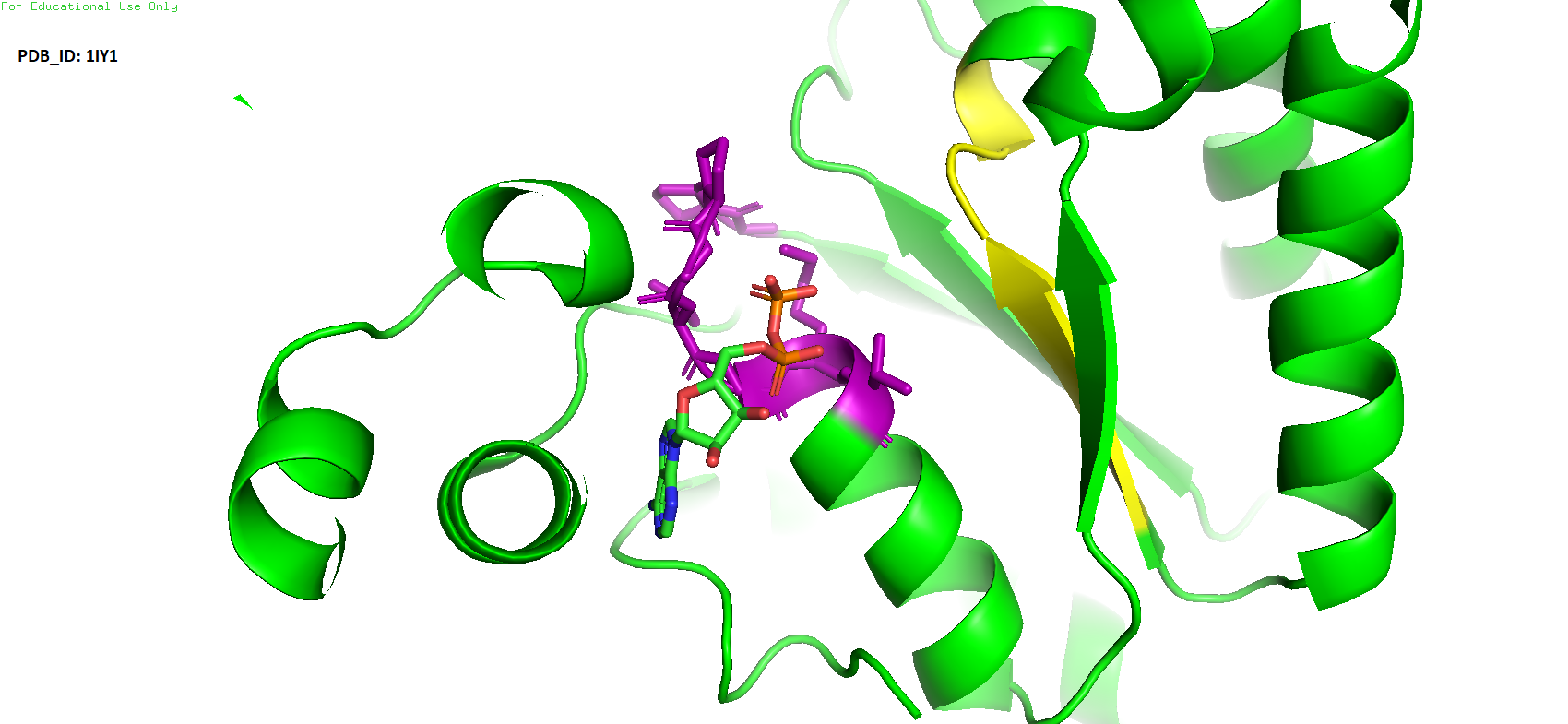
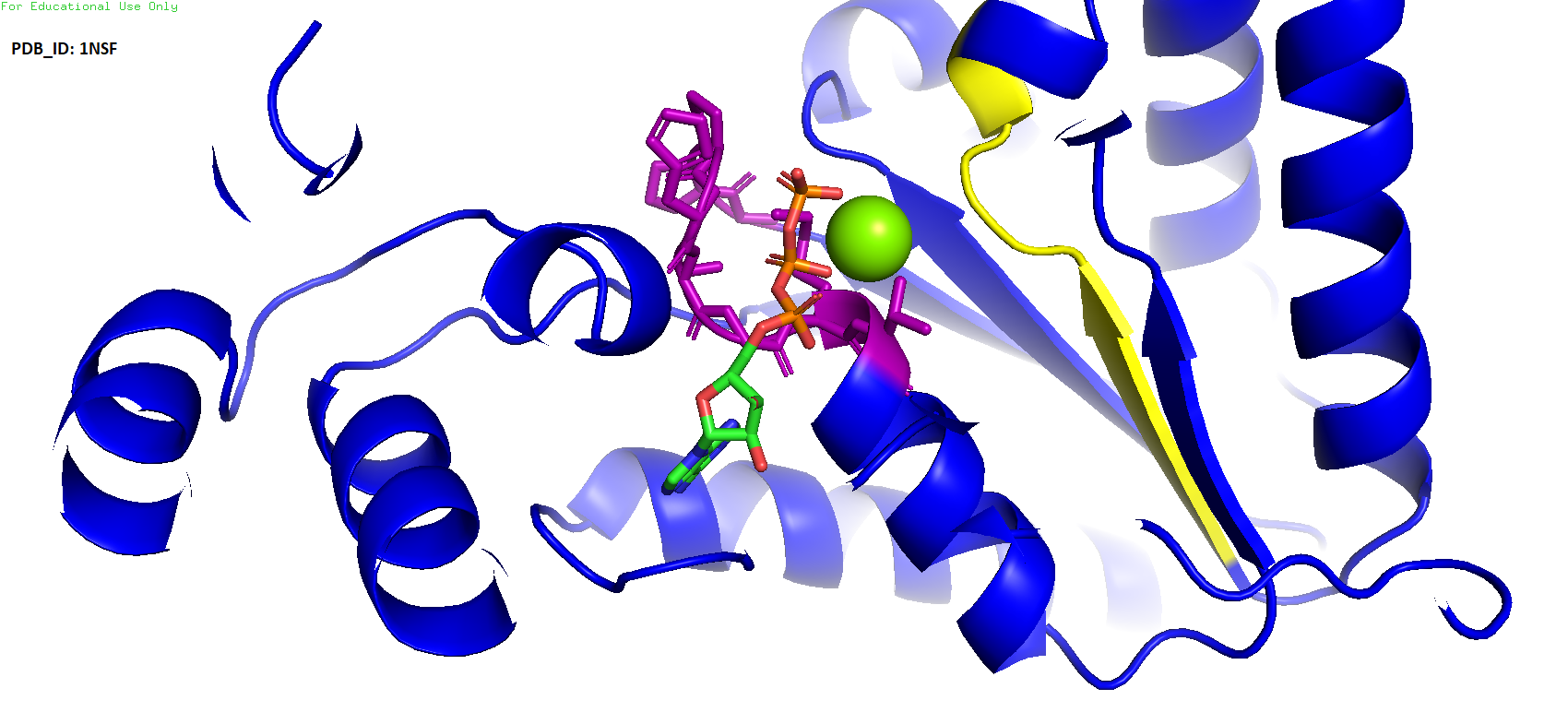
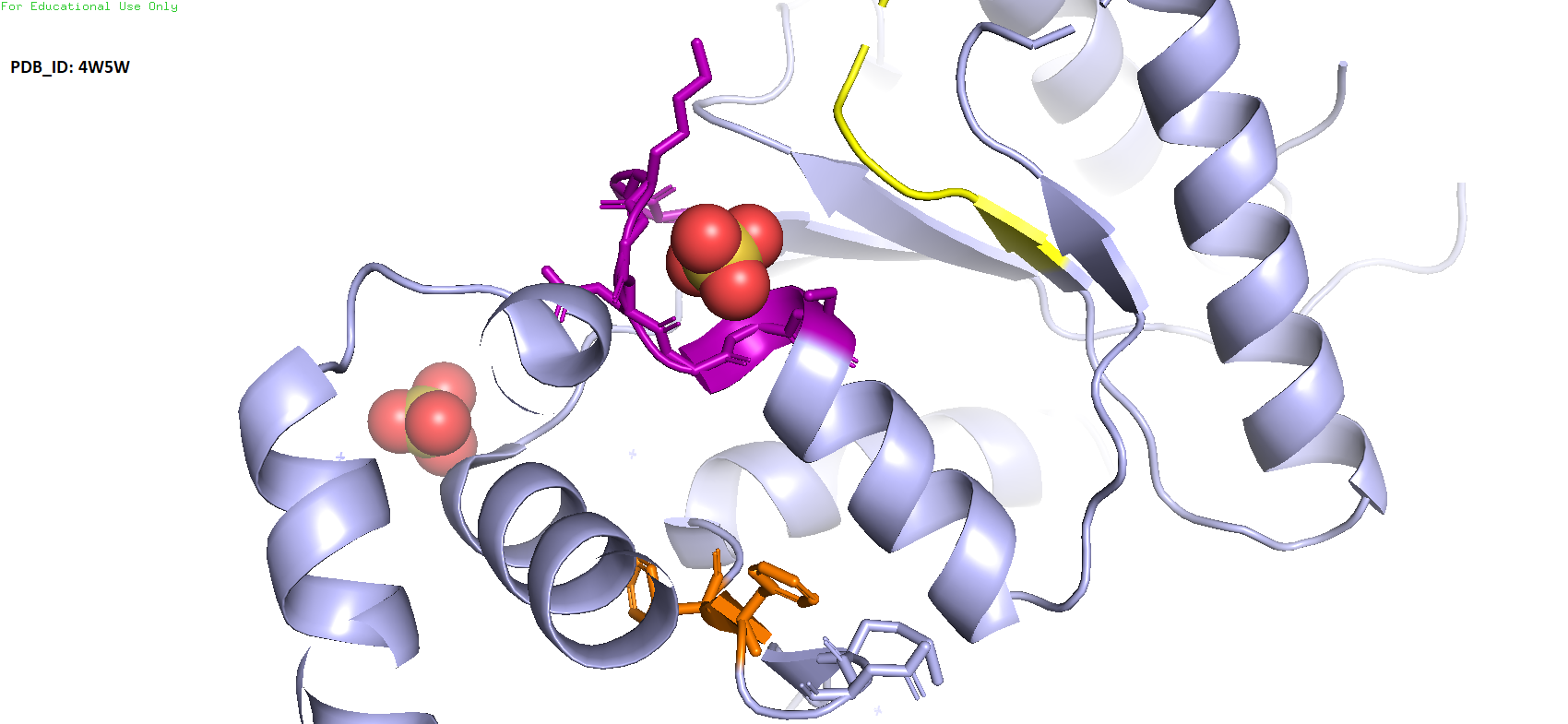
Рис. 8. Функциональные мотивы в трёх найденных белках
© Иззи Антон, 2020