Практикум 10
Поиск нуклеотидных последовательностей
Для выполнения данного задания поиск необходимых нуклеотидных последовательностей осуществлялся на сайте базы данных ENA с помощью следующего запроса:
Отобрались следующие две последовательности для сравнения: хромосома Lactobacillus paragasseri (нуклеотидный AC CP054573.1) и хромосома Lactobacillus kefiranofaciens subsp. kefirgranum (нуклеотидный AC CP098405.1). Таким образом сравнивались вид и подвид другого вида, принадлежащих роду Lactobacillus. Сравнение происходило выдачи двух разных алгоритмов blast для данных последовательностей, а именно сравнивались выдача megablast и blastn (параметры в обоих случаях были взяты по умолчанию). Отбор последовательностей необходимо было произвести так, чтобы эти две выдачи сильно отличались, это и можно наблюдать на Рис. 1 и 2 ниже.
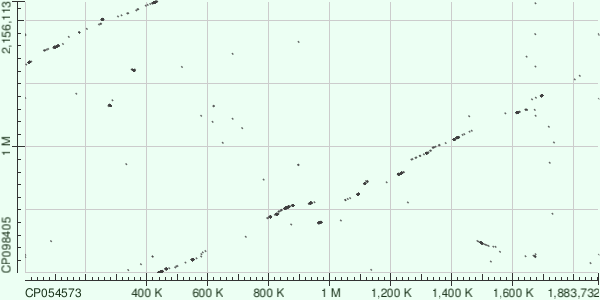
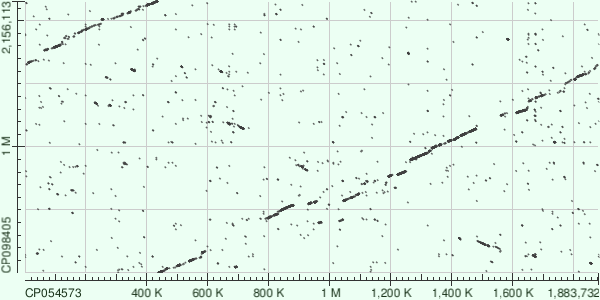
Как видно из Рис. 1 и 2, выдача megablast не слишком информативна (по ней не скажешь о схожести этих двух последовательностей и родстве бактерий, также на ней не видны различия в геномах, о которых написано ниже, это и неудивительно, так как данный алгоритм хорошо подходит только для почти полностью идентичных последовательностей), выдача же blastn гораздо более содержательна, по ней далее и будет проведен анализ и сравнение геномов.
Описание карты локального сходства
Здесь приводится описание карты локального сходства выдачи blastn с помощью вспомогательного изображения Рис. 3:
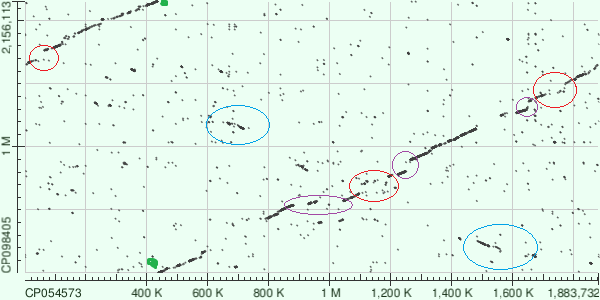
- Глобально можно заметить, что в обоих случаях был выбран один и тот же тип цепи, то есть данные цепи не комплементарны (об этом общий ход прямой на графике).
- Зеленой точкой на карте отмечен разрыв, это говорит о том, что в качестве начала для записи последовательности были выбраны разные точки на кольцевых хромосомах.
- Красным отмечены неконсервативные участки, то есть те, где по всей видимости последовательности крайне слабо похожи, или вовсе эти зоны соответствуют инделям и гэпам в выравнивании, об этом можно сделать вывод на основании того, что в данных зонах как будто бы пропущен участок линии на графике.
- Фиолетовым отмечены зоны инсерций/делеций, в первом случае (самый левый овал) либо произошла крупная вставка в нижней последовательности, либо крупная делеция в боковой, во втором и третьем случаях соответственно произошли либо небольшие вставки в боковой последовательности, либо небольшие делеции в нижней. Об этом можно судить по скачкам линий (по их разрывам и нарушении непрерывности в соответствующих участках).
- Голубым же цветом можно наблюдать транспозицию выделенных участков, которая приэтом сопряжена с инверсией в обоих случаях (наклон линии в другую сторону).
- В целом также можно наблюдать на карте большое число мелких точек и набросков, они соответствуют многочисленным повторам в последовательностях.
В итоге можно заметить, что несмотря на то, что организмы таксономически довольно близки (по сути это просто разные виды одного рода), в их геномах можно обнаружить довольно глобальные отличия и перестройки: делеции, инсерции, повторы, транспозиции и даже инверсии. По всей видимости это связано с конкретными видовыми особенностями данных организмов.