Практикум 14
Подготовка чтений
Для выполнения данного практикума был задан код доступа SRR4240356 (проект по секвенированию бактерии Buchnera aphidicola str. Tuc7). Вся дальнейшая работа осуществлялась в директории /mnt/scratch/NGS/slavik123/pr14. Для начала был скачан архив с чтениями по данному коду доступа:
Далее он был разархивирован:
Затем была выполнена подготовка чтений, на первом этапе нужно было удалить возможные остатки адаптеров. Для этого сперва был создан файл adapters.fa, где объединены все адаптеры из директории /mnt/scratch/NGS/adapters, следующей командой:
После чего адапетры были удалены с помощью программы trimmomatic:
Выдача оказалась следующей (на ней видно, сколько процентов последовательностей оказалось адапетрами), кстати после удаления адаптеров новый файл стал весить 777308708 (изначально 793999866):
На втором этапе все с помощью той же программы trimmomatic с правых концов чтений были удалены нуклеотиды с качеством ниже 20, а также были оставлены только те чтения, у которых длина не меньше 32:
Теперь размер файла стал 744045190, выдача следующая (на ней показано, сколько чтений было удалено на втором этапе):
Подготовка k-меров
После подготовки чтений на основе последнего получившегося файла SRR4240356.fastq2 с помощью программы velveth были созданы k-меры длины k = 31:
Сборка на основе k-меров
На основе созданных служебных файлов из предыдущего пункта с помощью программы velvetg была запущена сборка генома на основе k-меров:
Параметр N50 у данной сборки составляет 65554. Для дальнейшего анализа необходимо собрать информацию о некоторых контигах, для этого была использована информация из созданного в результате последней команды файла contigs.fa. Сперва были найдены 3 самых длинных контига следующей командой:
Самые длинные контиги это: 8 (длина 111962, покрытие 38.660198), 6 (длина 107488, покрытие 34.174030), 10 (длина 80939, покрытие 37.524174). Также были найдены два контига в качестве примера с аномально большим покрытием (команда прошлая, но вместо -k2 использовалось -k3), это контиги 27 - 458.429077 и 17 - 447.494751 (через тире указаны их покрытия).
Анализ
Наконец, с помощью программы megablast на сайте NCBI были произведены выравнивания каждого из 3 самых длинных контигов (смотри прошлый пункт) с банковской хромосомой Buchnera aphidicola (AC — CP009253). В случае с контигом 6 нашлось 18 участков для выравнивания, подробные характеристики можно посмотреть здесь, как видно из Рис. 1, данный контиг того же типа, что и цепь в банке (то есть некомплементарен). Данный контиг ложится на участок 220869-323043 на хромосоме.
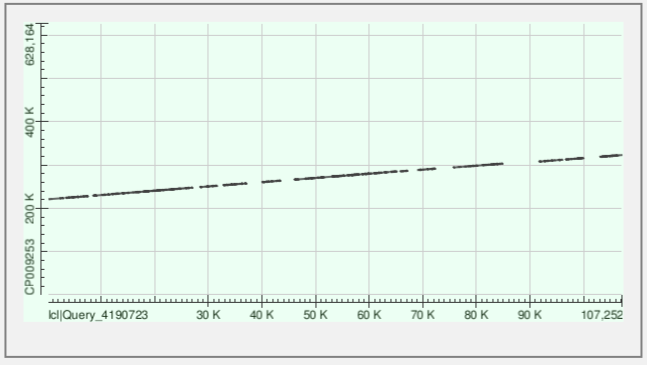
Для контига 8 нашлось 15 участков, подробные характеристики выравниваний лежат здесь, на Рис. 2 мы видим, что и этот контиг того же типа, что и цепь. Ложится он соответственно на участок 451729-555905.
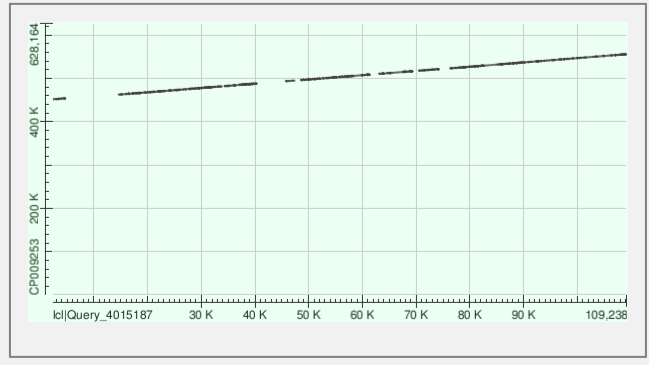
Для последнего контига 10 нашлось 11 участков, подробные характеристики выравниваний лежат здесь, на Рис. 3 мы видим, что этот контиг комплементарен банковской цепи (наклон в другую сторону). Контиг 10 соответствует участку 126623-195400.
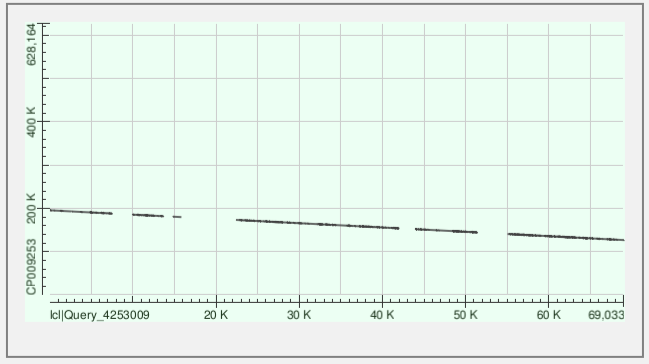
Пробелы в линиях на Рис. 1-3 соответствуют соответственно малоконсервативным участкам или вообще гэпам и инделям.