Практикум 3
Различные формы ДНК
С помощью программы fiber были сгенерированы три различные формы ДНК: А-форма (Рис. 1, ссылка на PDB-файл), B-форма (Рис. 2, ссылка на PDB-файл) и Z-форма (Рис. 3, ссылка на PDB-файл).

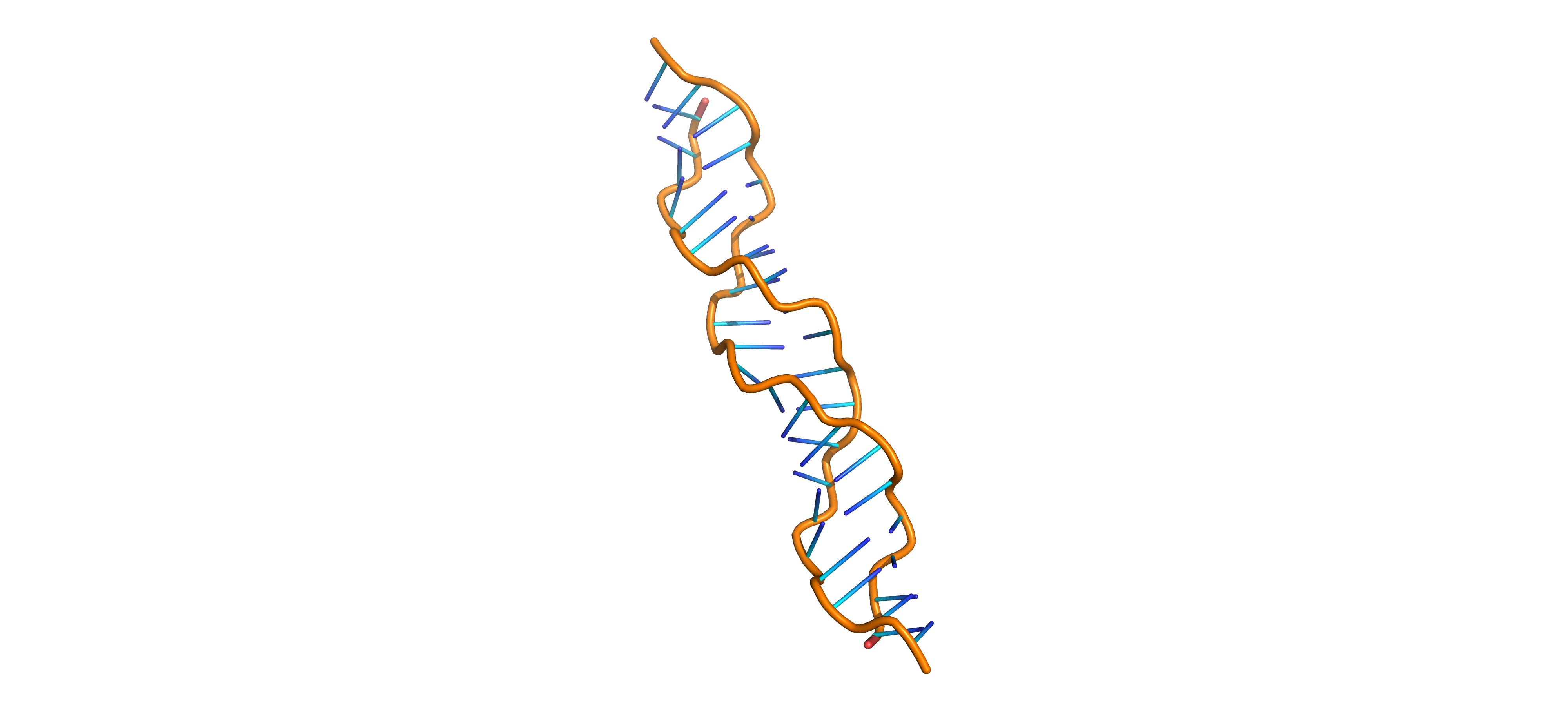
Характеристики различных форм ДНК
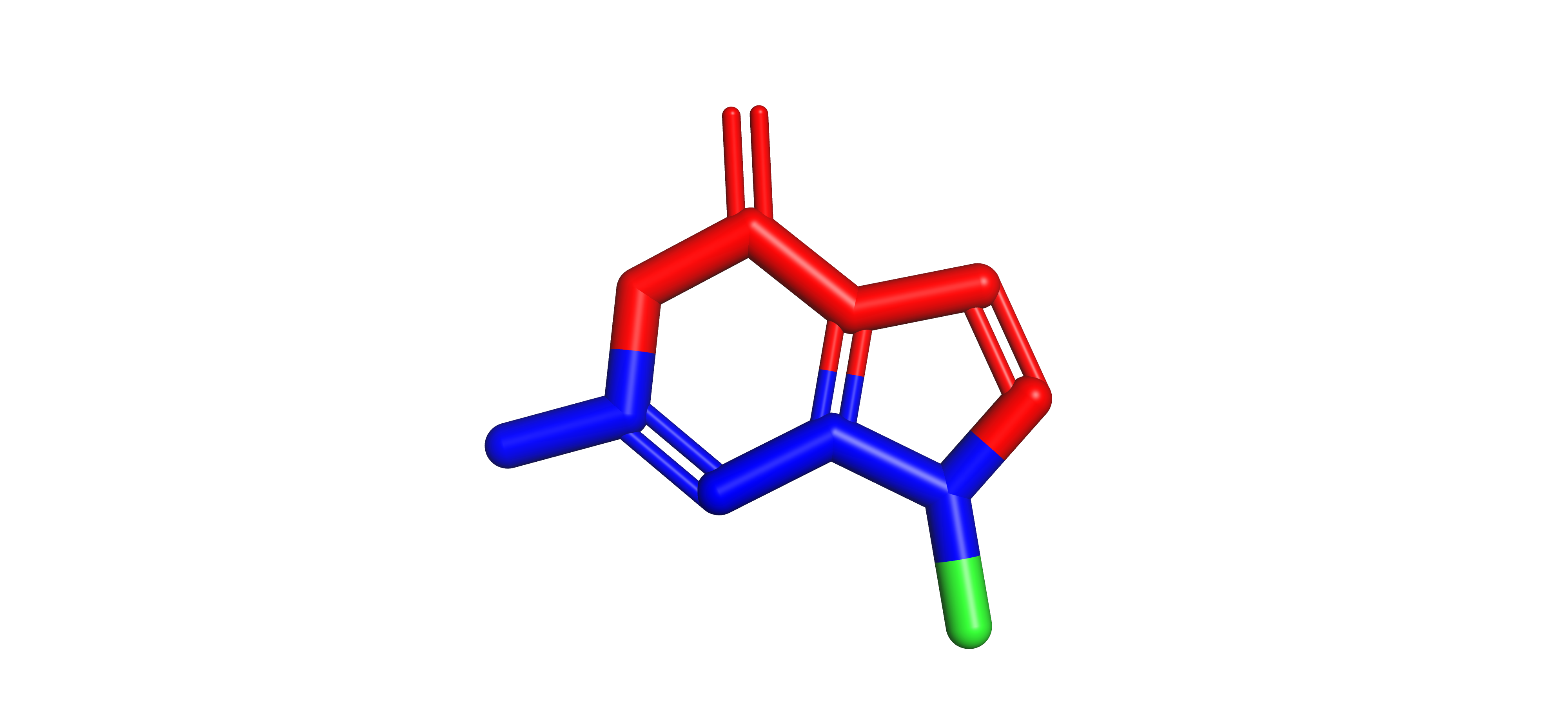
Для анализа была взята B-форма ДНК, структура 1bna, основание G4 (Рис. 4):
- В сторону малой бороздки обращены атомы: N2, C2, N3, C4, N9.
- В сторону большой бороздки обращены атомы: N1, C6, O6, N7, C8, C5.
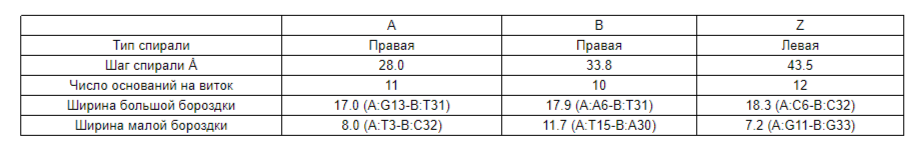
Ниже представлены различные числовые характеристики разных форм ДНК, в Табл. 1 эти данные получены на основе сгенерированных программой fiber моделей из пункта выше, а в Табл. 2 данные на основе структур из PDB (для A-формы это 3v9d, для B-формы это 1bna, для Z-формы предложенная структура 1tne слишком короткая для определения числовых характеристик):

Характеристики заданной структуры тРНК
1. Торсионные углы.
С помощью программы analyze и данных из файла 1ffy.out (который генерируется как результат прогрыммы analyze). Значения всех торсионных углов для данной тРНК можно посмотреть здесь. Как видно, значения этих углов крайне варьируют и в целом не образуют никакой закономерности, поэтому можно сделать вывод, что тяжи заданной тРНК не похожи на А- или B-формы ДНК.
2. Структура и устройство водородных связей.
Из все того же файла 1ffy.out была извлечена полная информация о водородных связях, присутствующих в структуре тРНК, ее можно посмотреть здесь.
Стебли образованы следующими парами нуклеотидов: первый стебель образован парами 1-7 и 72-66, второй стебель – 10-13 и 25-22, третий стебель – 38-44 и 32-26, четвертый стебель – 49-53 и 65-61.
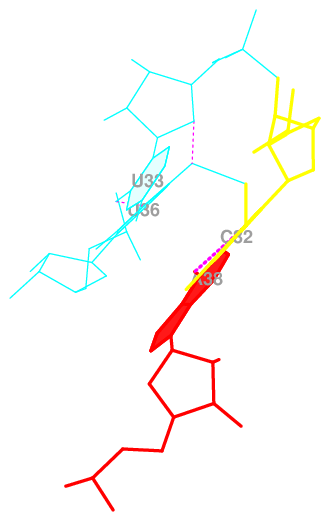
Неканонические пары: U5-G68, G49-U65, U55-G18, U36-U33, A38-C32, A44-G26, G16-U17.
Дополнительные водородные связи, стабилизирующие структуру тРНК: U54-A58, U55-G18, A14-U8, G15-C48, G16-U17, G19-C56, данные нуклеотиды не входят в состав стеблей.
3. Экстремальные площади перекрывания стэкинговых взаимодействий.
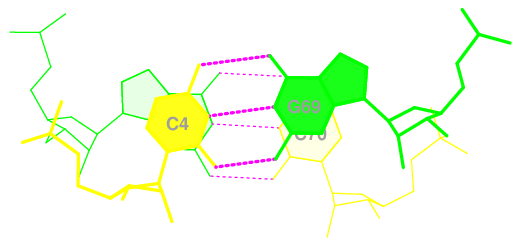
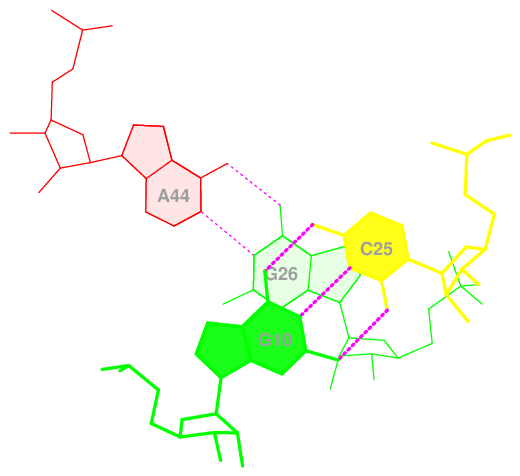
Были также получены данные о площадях перекрывания для двух последовательных пар азотистых оснований, их можно посмотреть здесь. Отсюда видно, что 3 пара G3C4-C70G69 имеет наибольшую площадь перекрывания (11.95, Рис. 5), на втором месте пара 15 U36A38-U33C32 (11.45, Рис. 6), минимальные же значения перекрывания площадей у пар 1 G1G2-C72C71 (1.98, Рис. 7) и 22 A44G10-G26C25 (1.56, Рис. 8).