Практикум 8
Ген, кодирующий δ-субъединицу АТФ-синтазы
Для выполнения последующих двух исследований и анализов необходимо было получить некоторые данные о гене δ-субъединицы АТФ-синтазы для выбранного мною в прошлом практикуме организма (это C. elegans, смотри Практикум 7). Из файлов, скачанных и представленных в прошлом практикуме, были получена следующая необходимая информация о данном гене у данного организма:
- NP_495286.1 – идентификатор δ-субъединицы, ее аминокислотную последовательность можно посмотреть в fasta-файле.
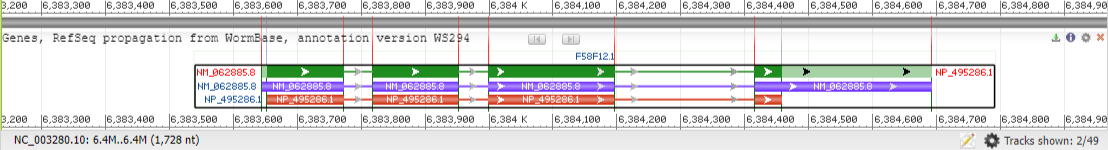
- NC_003280.10 – идентификатор нуклеотидной записи, к которой относится данный ген (оказалось это хромосома 2). Для его получения был использован первый идентификатор.
- neighbourhood_of_gene.fasta – фаста-файл, содержащий участок с координатами 6383239-6384966 их второй хромосомы, данный участок содержит исследуемый ген 6383645-6384692, ниже предсьавлено изображение данного участка (Рис. 1) (данные получены благодаря второму идентификатору):
BLAST
АТФ-синтаза – один из самых известных и распространенных белков среди аэробных организмов. Мне захотелось проверить его консервативность на примере структуры гена и конкретного белка, входящего в состав АТФ-синтазы (собственно δ-субъединицы моего ранее выбранного организма).
Для проведения данного исследования необходимо было выбрать таксон, достаточно далекий от исходного организма (поскольку интересует именно консервативность глобально, а не среди родственников), поскольку мой организм принадлежит к первичноротым животным, то таксон выбирался среди вторичноротых. Выбор был среди наиболее всем известных представителей: семейство Кошачьи (Felidae) и Собачьи (Canidae). Методом тыка был выбрал первый таксон. Поиск нужной информации происходился с помощью двух типов программы BLAST (об этом ниже) на сайте . База данных для поиска была взята довольно известная база данных RefSeq Genome Database, в которой было 15 геномных сборок, входящих в этот таксон. Программы были следующие:
- Программа blastn – для начало захотелось проверить насколько сильно могут быть похожи именно гены (а точнее даже экзоны) данной субъединицы, поэтому и использовалась эта программа (для поиска небольших похожих нуклеотидных последовательностей среди неблизкородственных организмов). На вход подавался фрагмент из файла n_gene.fasta (описанный выше), а именно интересующий меня ген (в программе указывались параметры from и to, координаты гена в участке известны, если перевести их на счет с 1, то будет 407-1454). Параметры были взяты по умолчанию (E-value = 10, word_size = 11). С результатами выдачи можно ознакомиться в этом файле. Оказалось 137 находок, причем найдено среди всех 15 представителей, однако если внимательно изучить результаты выравнивания, то можно обнаружить, что, во-первых, среди находок нет генов именно δ-субъединицы, во-вторых, как видно на Рис. 2, в выравнивание в основном попали граничные области экзонов и интронов, плюс участки крайне небольшие (не более 100 нуклеотидов). По итогу видно, что результаты крайне не утешительные (по ним вполне можно сказать, что субъединицы у наиболее известных Кошачьих и нематоды отличаются), впрочем это было вполне ожидаемо, так как blastn работает с небольшими участками, и к тому же не учитывает свойство вырожденности генетического кода, поэтому было проведено второе испытание.
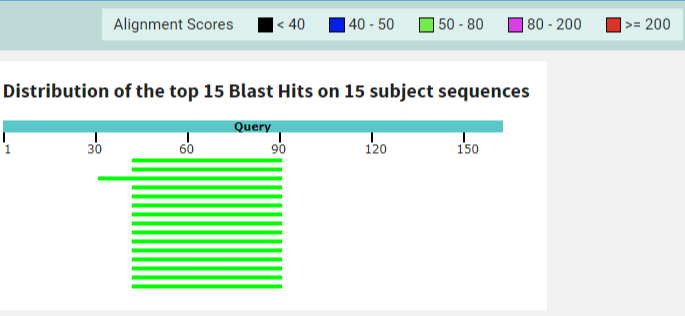
- Программа tblastn – здесь уже в качестве запроса подавалась уже сама последовательность белка (смотри fasta-файл в самом первом абзаце), параметры были взяты также по умолчанию (E-value = 0.05, word_size = 5), данный алгоритм нужен для сравнения белка-запроса с транслированными последовательностями из базы данных, результаты можно увидеть в файле. Как видно из результатов, обнаружилось 15 находок, снова среди всех представителей семейства, также видно, что среди находок именно последовательности δ-субъединицы, а не абы чего, можно наблюдать довольно высокий процент покрытия для локального выравнивания (30%) и довольно высокий процент идентичности (в большинстве более 50%), на Рис. 3 можно видеть, какие участки наиболее оказались схожи у нематоды и Кошачьих. На основании этого, а также крайне низкого значения E-value, можно действительно убедиться, что в общем-то данный белок является высококонсервативным, по крайней мере его последовательность не так уж и сильно отличается между такими разными и далекими животными.

Гены рРНК
В этом задании я попытался отыскать гомологов 16S рРНК (последовательность) и 23S рРНК (последовательность) E. coli у выбранной мною нематоды. Для этого сперва был проиндексирован мой геном (файл genome_seq.fna) следующей командой:
После чего был запущен алгоритм blastn (был выбран именно он, так как он подходит для поиска похожих некодирующих белки нуклеотидных последовательностей даже среди неблизкородственных организмов), в качестве запроса были поданы как раз выше упомянутые последовательности 16S и 23S рРНК. Команды следующие:
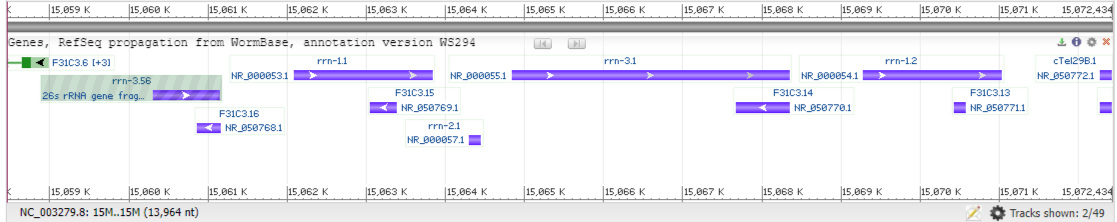
Результаты выдачи blastn можно посмотреть здесь: 16S-таблица и текст, 23S-таблица и текст (текстовый формат был получен аналогичными командами, но без -outfmt 7). Как видно для 16S было обнаружено 10 находок, среди них все находки, относящиеся к хромосоме 1 содержат участки 2 генов 18S рРНК у нематоды, то есть это и есть искомый гомолог для 16S, остальные находки относятся либо к неаннотированным областям, либо к генам белков. Для 23S – 13 находок, и снова те, что относятся к хромосоме 1 содержат участки гена, кодирующего 26S рРНК, а также псевдогена этой рРНК, и это второй искомый гомолог (они аннотированы и аннотация совпадает с ожиданиями для обоих рРНК), остальные находки не относятся к гомологам. На Рис. 4 можно видеть найденный участок с гомологами к рРНК у нематоды:
Известно, что рРНК необходимы для поддержания структуры и работы рибосом, так 16S рРНК помимо структурной функции для малой субъединицы также необходима для связывания с мРНК во время трансляции (по последовательности Шайно-Дельгарно), а 23S рРНК армирует большую субъединицу и катализирует реакцию роста пептидной цепи в ъоде трансляции. Аналогичные функции выполняют и найденные гомологи у нематоды (конкретно 18S и 26S для цитозольных эукариотических рибосом).