Практикум 10
Поиск консервативных мотивов в выравнивании
Для выполнения заданий данного практикума был выбран домен Transcription-initiator DNA-binding domain IBD c идентификатором PF10416. Данный домен является специфичным для вида Trichomonas vaginalis, обладает особой конформацией типа "крыло-спираль-крыло", которая позволяет белку связываться с сайтом TCAPy в участке Inr в ДНК, активируя РНК-полимеразу и тем самым инициируя транскрипцию. В сиде для данного домена содержится 94 последовательности, с помощью программы Jalview был осуществлен поиск консервативных мотивов (предварительно последовательности на 90% идентичные и более были убраны, однако таких не оказалось). В результате был найден один консервативный мотив с высоким IC (столбцы 66-75, ссылка на проект). На основе данного сида вероятный паттерн данного мотива может быть следующим в формате Jalview R.[LIM][VAL][CAV]G[VIL].[WCF]. Далее с помощью команды в консоле:
был проведен поиск по данному паттерну в выравнивании (с выдачей программы можно ознакомиться здесь), в результате в 38 из 94 последовательностей данный паттерн был найден успешно. Данный результат, а также само выравнивание, говорит о том, что наиболее консервативными являются R и G, остальные позиции в мотиве гораздо более вариабельны (однако в пределах одного класса аминокислот). Наконец, в формате Prosite данный паттерн имеет следующий вид: R-x-[LIM]-[VAL]-[CAV]-G-[VIL]-x-[WCF]-x. На одноименном веб-сервисе с помощью ScanProsite был осуществлен поиск по данному паттерну в базе данных SwissProt. В результате было найдено 71 последовательность (ссылка на выдачу), приэтом ни одна последовательность из сида не была найдена, что неудивительно, ведь для выбранного домена по данным из pfam есть всего 252 белка, причем все они не имеют аннотации в SwissProt, где осуществлялся поиск, однако среди найденных последовательностей примечательно, что они из самых разных организмов (от бактерий до млекопитающих, включая человека), белки приэтом однако по функциям довольно разные (судя по данным из Uniprot), так что вероятно эти находки имеют случайное отношение к данному паттерну и мотиву.
Поиск специфичного мотива
Далее с помощью метода UPGMA в Jalview было построено филогенетическое дерево для данного набора последовательностей, после чего была отдельно взята клада с 7 последовательностями (ссылка на проект), в ней был найден на позициях 79-88 специфичный для нее мотив с паттерном K[LI]N[TS]LN[VS]NL[RK]. Это подтверждается тем, что среди всех 94 последовательностей данный паттерн был обнаружен в 6 (как раз из этой клады) с помощью следующей команды:
PSI-BLAST
Для выполнения данного задания был выбран белок со следующим AC: Q7VDL2. Данный белок является ингибитором клеточного деления у цианобактерии Prochlorococcus marinus (strain SARG / CCMP1375 / SS120), которые блокирует образование полярных Z-кольцевых перегородок за счет предотвращения полимеризации белка FtsZ, образующего нити этих колец. Для этого белка с помощью PSI-BLAST был осуществлен поиск семейства гомологов, ниже в Таб. 1 представлена таблица итераций.
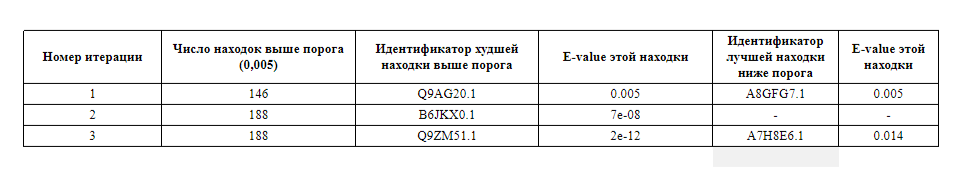
По таблице видно, что PSI-BLAST сумел найти семейство достаточно хорошо всего за 3 итерации (произошла стабилизация результата, разница E-value между худшей правильной находкой и лучшей неправильной довольно большая). Результат последней итерации можно посмотреть здесь. Отсюда видно, что все правильные белки имеют ту же функцию (судя по названию), что и исследуемый.
Поиск мотивов de novo с помощью MEME
Из базы данных Pfam были скачаны последовательности белков с выбранным ранее доменом в формате fasta. С помошью консольной программы meme был проведен поиск de novo мотивов в этих белках:
Выдача программы MEME оказалась следующей. Всего обнаружилось 4 мотива, все они имеют достаточно низкий E-value и обнаружены в большинстве поданных на вход последовательностей. Среди этих мотивов есть как раз тот, что был обнаружен в сиде, на Рис. 1 представлена его лого-диаграмма, на ней хорошо видны две максимально консервативные позиции R и G. Его частота обнаружения самая большая, что подтверждает его значимость и важность для данного домена.
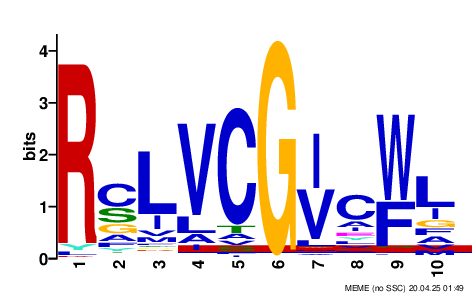
Оценка представленности сайта GATC
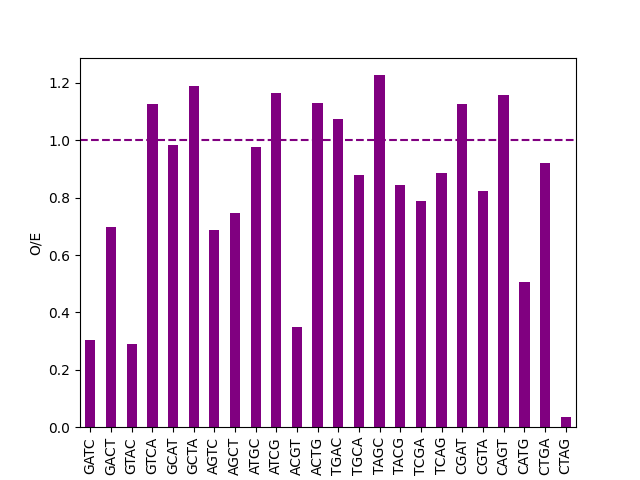
Для проведения оценки понадобился геном моей археи Methanocaldococcus lauensis в fasta-формате, а также набор 24 сайтов. Далее для вычисления контрастов была использована следующая программа:
В результате чего была построена гистограмма (смотри Рис. 2). По ней видно, что интересуемый сайт метилирования у бактерий GATC сильно недопредставлен (значение O/E = 0.3) в геноме моей археи, по всей видимости для нее данный сайт не несет какой-то значимости, вероятно сайтом метилирования является какой-то другой сайт (скорее всего из тех, для кого O/E в районе 1).