Практикум 3
Реконструкция дерева по нуклеотидным последовательностям
Для группы животных из прошлых практикумов было построено дерево, но уже на основании последовательностей 12S рРНК. К сожалению, не для всех видов нашлись их последовательности, поэтому необходимо было заменить их на другие виды, в качестве замен были выбраны наиболее близкородственные организмы (все в пределах того же рода или даже подрода), ниже представлены конкретные замены:
- Вид с мнемоникой PSAVE был заменен на вид Psammomys obesus (мнемоника PSAOB).
- Вид с мнемоникой HYSAF был заменен на вид Hystrix indica (мнемоника 9HYST).
- Вид с мнемоникой SYLPA был заменен на вид Sylvilagus audubonii (мнемоника SYLAU).
- Вид с мнемоникой MYOMA был заменен на вид Myotis muricola (мнемоника MYOMU).
- Вид с мнемоникой SORCI был заменен на вид Sorex minutissimus (мнемоника SORMN).
- Вид с мнемоникой SORCO был заменен на вид Sorex cylindricauda (мнемоника SORCY).
На Рис. 1 представлено сравнение этого дерева, построенного на основе 12S рРНК, полученного с помощью программы FastMe и модели p-distance, с деревом, основанным на таксономии NCBI. Видно, что ошибки реконструкции следующие:
- Образовалась ложная клада (TUPGL, (PIPKU, MYOMU)).
- Образовалась ложная клада (LEMCA, 9HYST) и ((LEMCA, 9HYST), PONPY).
- Образовалась ложная клада ((SORCY, SORMN), PSAOB).
- Глобально же видно, что нарушена структура двух глобальных клад, соответствующих укоренению на дереве таксономии, а именно клада (SORCY, SORMN) переехала в другую глобальную группу, а в первую внедрился вид TUPGL.
- В целом видно, что в данном дереве довольно много ошибок реконструкции, из полностью правильных клад сохранились лишь (SORCY, SORMN), (PIPKU, MYOMU) и (LEPYA, SYLAU).
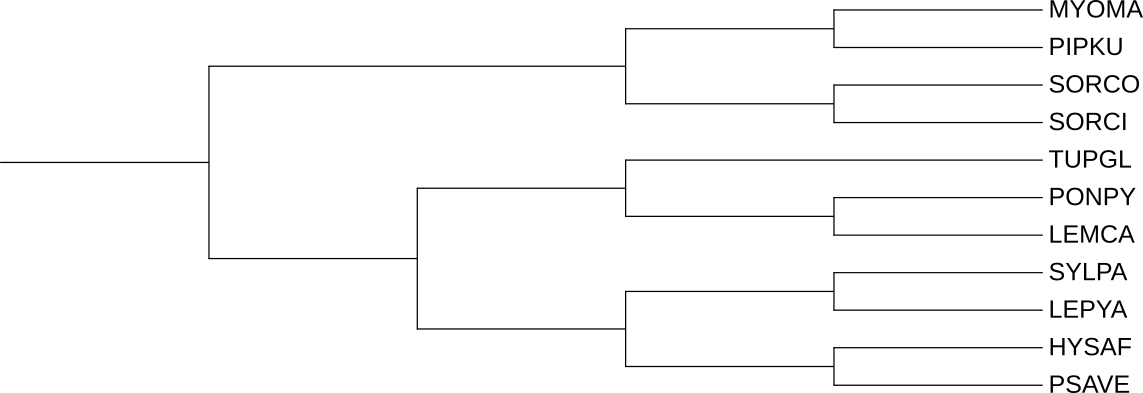

На Рис. 2 представлено сравнение этого же дерева только теперь из одним из деревьев, построенным по белковым последовательностям из прошлого практикума, полученного тоже с помощью программы FastMe и модели p-distance. Можно заметить следующие различия и сходства:
- В обоих случаях вид TUPGL был внедрен в другую глобальную кладу, сохранены клады (PIPKU, MYOMA), (LEPYA, SYLPA), (SORCI, SORCO).
- По сравнению с деревом на основе 12S рРНК в дереве на основе цитохрома сохранено больше правильных клад (LEMCA, PONPY), (PSAVE, HYSAF), а также клада (SORCI, SORCO) осталась в своей правильной глобальной кладе.


Укоренение во внешнюю группу
Для дерева на основе цитохрома и модели p-distance из прошлого практикума была произведена перестройка с использованием внешней группы. В качестве нее был взять вид Carcharodon carcharias (мнемоника CARCH), который в отличие от представителей выбранной группы животных является хрящевой рыбой, а не млекопитающим. На Рис. 3 представлен результат, видно что использование внешней группы не решило глобальной проблемы, вид TUPGL по-прежнему внедрен не в ту глобальную кладу.

Бутстреп
На Рис. 4 показана реконструкция из прошлого практикума на основании белка цитохрома B, построенная программой FastMe и моделью MtREV, перестроенная с использованием бутстреп-поддержки. Как можно видеть, правильные клады (LEPYA, SYLPA), (SORCI, SORCO), (PIPKU, MYOMA) имеют крайне высокий уровень поддержки (100 и 99), что дополнительно подтверждает их корректность. Низкий уровень поддержки клад (PSAVE, (LEMCA, PONPY)) (19), (TUPGL, (SORCI, SORCO)) (43), (HYSAF, (PIPKU, MYOMA)) (40), а также уровень 11 у одной из глобальных клад подтверждает, что они были реконструированы неверно. Однако есть правильно реконструированная клада (LEMCA, PONPY), но она имеет довольно низкий уровень поддержки (47).
