Практикум 4
Составление списка гомологичных белков, включающих паралоги
Для выполнения данного практикума были отобраны протеомы следующих бактерий из директории /P/y22/term4/Proteomes (ниже приведены их мнемоники):
- AROAE
- BRUSU
- PASMU
- PSEMY
- SACD2
- YERPE
- THIDA
Далее необходимо было подготовить эти протеомы для запуска программы blastp, для этого сперва протеомы выбранных бактерий были объединены в один файл следующей командой (вся работа проводилась в отдельной дирректории):
Затем на основе выбранных протеомов была создана локальная база данных для запуска blastp с помщью команды:
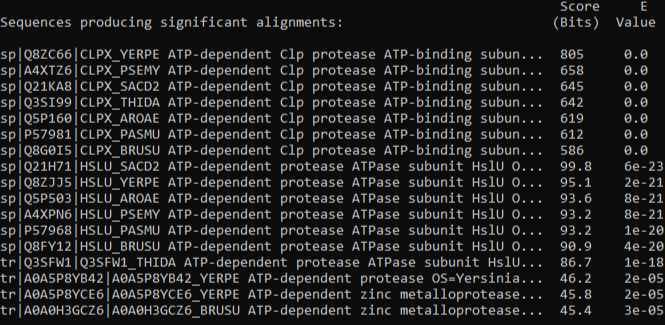
После чего наконец был запущен blastp на основе созданной базы данных, в качестве запроса была подана последовательность белка с мнемоникой CLPX_ECOLI (расположена в файле query.fasta), для отбора достоверных гомологичных белков среди отобранных бактерий был поставлен порог на E-value в 0.0001:
В результате был получен следующий список находок (смотри Рис. 1):
Реконструкция и визуализация
После получения списка находок были собраны в один файл fasta-файлы соответствующих последовательностей, после чего было проведено выравнивание программой muscle, на основании которого после перевода в нужный формат .phy при помощи скрипта было построено дерево реконструкции с помощью программы FastME, в качестве модели была взята MtREV, также была использована бутстреп-поддержка (100 реплик), в общем были проведены действия аналогичные таковым во 2 и 3 практикумах. Результат можно увидеть на Рис. 2. Формулу дерева в формате Newick можно посмотреть здесь.

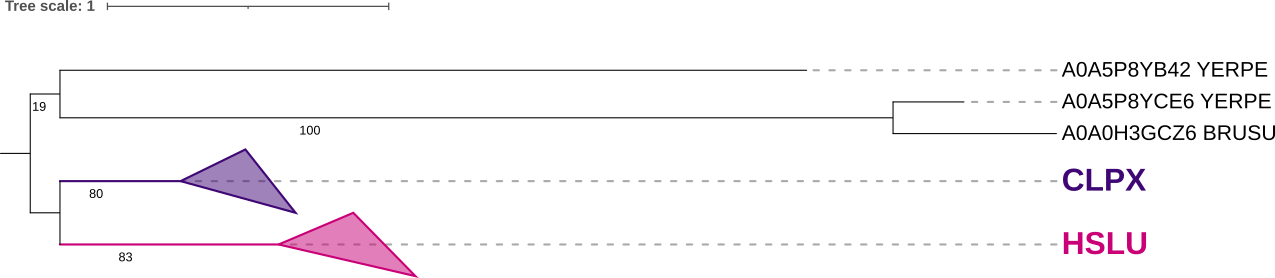
На Рис. 3 можно на построенном дереве с помощью разных цветов показаны различные ортологические группы (наборы попарно ортологичных белков, как минимум 4 последовательности в группе), как видно таких выделилось 2 штуки (CLPX - АТФ-связывающая субъединица АТФ-зависимой протеазы Clp, HSLU - АТФазная субъединица АТФ-зависимой протеазы HslU). На дереве также можно наблюдать примере пар ортологов и паралогов:
- Примеры пар ортологов: CLPX_BRUSU и CLPX_PASMU, HSLU_PSEMY и HSLU_SACD2, A0A0H3GCZ6_BRUSU и A0A5P8YCE6_YERPE.
- Примеры пар паралогов: CLPX_AROAE и HSLU_AROAE, A0A5P8YB42_YERPE и CLPX_YERPE, Q3SFW1_THIDA и HSLU_THIDA.
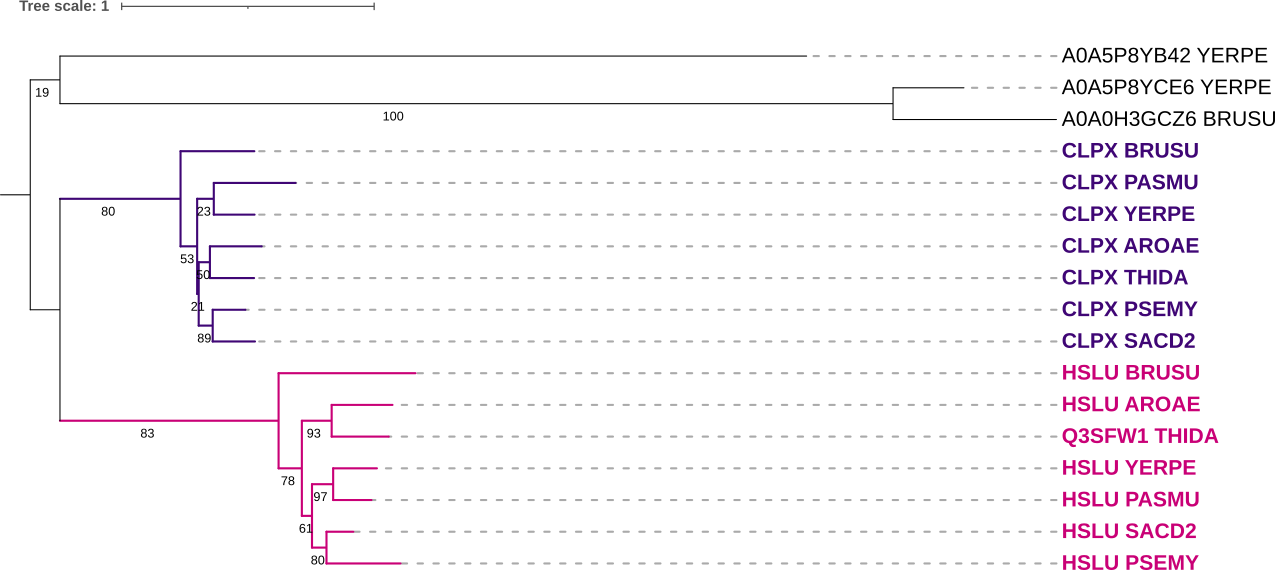
На Рис. 4 выделенные ранее ортологические группы схлопнуты, как уже говорилось таких групп 2, и в обоих группах присутствует последовательность каждой из выбранных 7 бактерий (то есть группы полные), теперь по поводу соответствия реконструкции групп ортологов и филогении бактерий (верная филогения бралась по рекомендации из этого изображения):
- CLPX: соответствует филогении за исключением одной крупной клады: ((AROAE, THIDA), (SACD2, PSEMY)) реконструирована неверно, должно быть ((SACD2, PSEMY), (YERPE, PASMU)).
- HSLU: полностью соответствует филогении.