Практикум 7
Сравнение предсказаний трансмембранных участков в бета-листовом белке
Для выполнения данного задания мною был выбран белок Фибринолизин (он же активатор плазминогена). Ниже приведены некоторые его характеристики на основе данных из OPM, а на Рис. 1 приведена его структура и расположение в мембране:
- Type: Transmembrane.
- Class: Beta-barrel transmembrane.
- Superfamily: Omptin.
- Family: OM protease omptin, OMPT.
- Protein: Plasminogen activator PLA (coagulase/fibrinolysin).
- Species: Yersinia pestis (Чумная палочка).
- Localization: Bacterial Gram-negative outer membrane (внешняя мембрана грам-отрицательной бактерии).
- Uniprot ID: PLA_YERPE.
- PDB ID: 2X55.
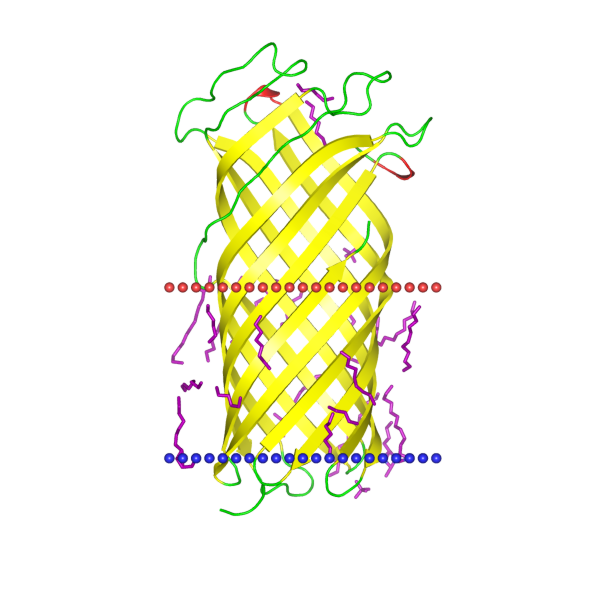
Данный белок активирует Glu-плазминоген у млекопитающего-хозяина (превращает его в плазмин) путем расщепления пептидной связи между Arg560 и Val561 (каталитическая активность EC:3.4.23.48). Плазмин разрушает фибриновые сгустки (осуществляет фибринолиз), что позволяет чумной палочке мигрировать из первоначального очага инфекции далее вглубь организма. Также данный белок способен разрушать аргининовые связи в белках.
С помощью программы DeepTMHMM были предсказаны трансмембранные участки у этого белка (на вход программе подавалась последовательность белка в fasta-формате из соответствующей записи в Uniprot). На Рис. 2 приведена графическая выдача программы, с текстовой выдачей можно ознакомиться здесь.
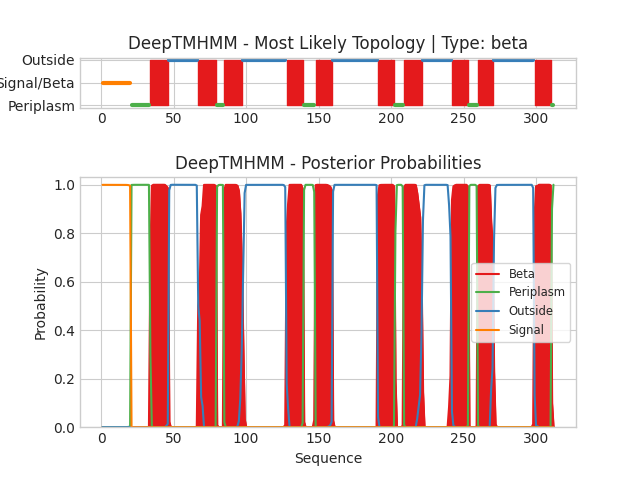
Ниже в Таб. 1 приведены трансмембранные участки для фибринолизина, которые предсказаны программой DeepTMHMM и которые представлены в OPM:
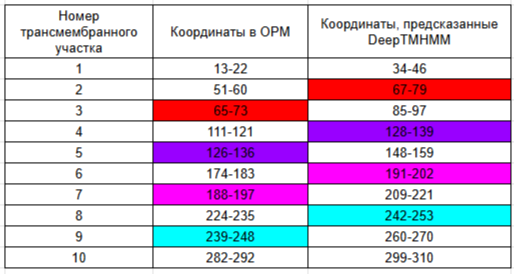
Из результатов в таблице видно, что в обоих случаях трансмембранных участков 10 штук, приэтом лишь 4 участка нормально перекрываются (хотя границы могут выходить на 7 АК, что плохо при длине участка в среднем в 10-12 АК). Кажется, что результаты предсказаний не совпадают, однако, стоит отметить, что первые 20 АК в пептидной последовательности являются сигнальным участком (а последовательность для DeepTMHMM была взята из Uniprot, в ней 312 АК, а в OPM всего лишь 293 и в PDB-структуре), также можно заметить, что если прибавить 20 остатков к границам участков OPM, то перекрытия будут гораздо лучше. Поэтому было решено вырезать первые 20 АК из последовательности fasta, и снова запустить DeepTMHMM. Новая текстовая выдача, новая графическая выдача на Рис. 3, а в Таб. 2 новое сравнение:
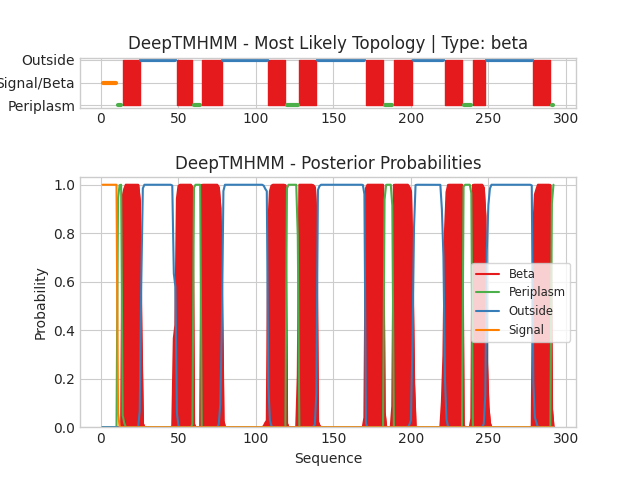
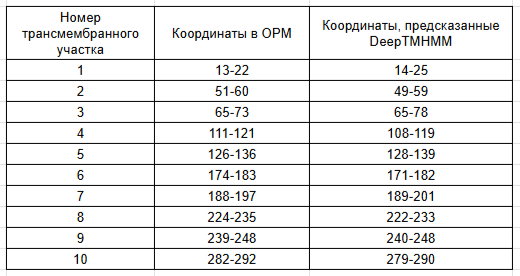
Теперь результаты предсказаний совпадают отлично, 10 участков и все перекрываются, границы выходят в среднем на 2-3 АК. Однако все равно программа DeepTMHMM делает небольшие косяки (например, первые 10 АК она определяет как сигнальную последовательность, это видно в текстовой выдаче, хотя истинная сигнальная последовательность была удалена).
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Для выполнения данного задания был дан белок Бактериородопсин I. Ниже приведены некоторые его характеристики на основе данных из OPM, а на Рис. 4 приведена его структура и расположение в мембране:
- Type: Transmembrane.
- Class: Alpha-helical polytopic.
- Superfamily: Rhodopsin-like receptors and pumps.
- Family: Microbial and algal rhodopsins.
- Protein: Bacteriorhodopsin-I.
- Species: Haloquadratum walsbyi (Галофильная архея).
- Localization: Archaebacterial membrane (мембрана археи).
- Uniprot ID: BACR1_HALWC.
- PDB ID: 4WAV.
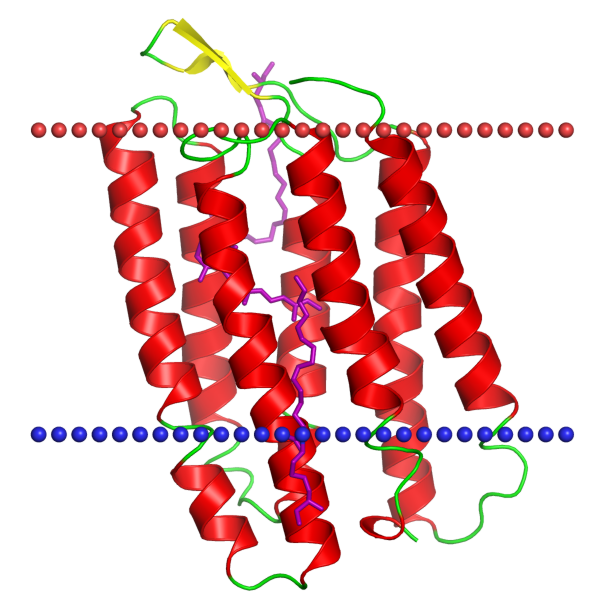
Данный белок является светозависимой протонной помпой у галофильной археии, который под действием света осуществует перенос протона через мембрану, тем самым являясь аналогом хлорофила, обеспечивая преобразование энергии солнца в энергию химических связей.
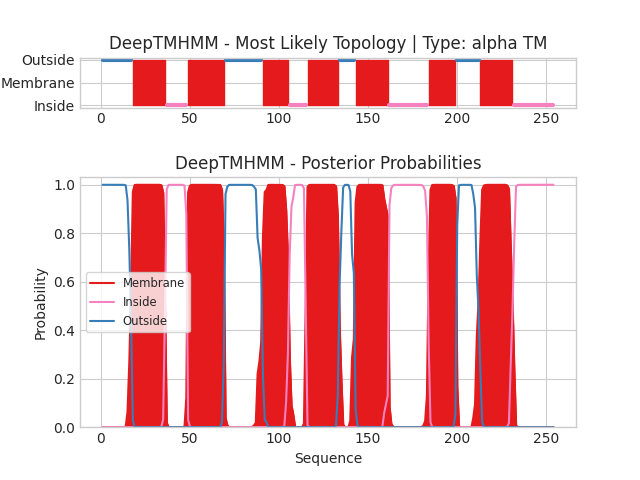
С помощью программы DeepTMHMM были предсказаны трансмембранные участки у этого белка (на вход программе подавалась последовательность белка в fasta-формате из соответствующей записи в Uniprot, сигнальной последовательности у данного белка нет, поэтому вырезать ничего не пришлось). На Рис. 5 приведена графическая выдача программы, с текстовой выдачей можно ознакомиться здесь.
Ниже в Таб. 3 приведены трансмембранные участки для бактериородопсина-I, которые предсказаны программой DeepTMHMM и которые представлены в OPM:
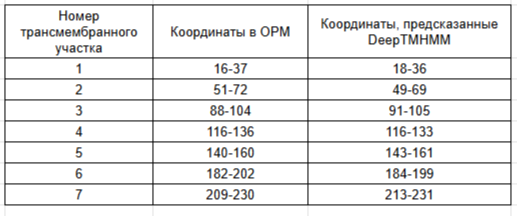
Как видно предсказания совпали успешно, 7 участков в обоих случаях, везде есть перекрывания, границы отличаются незначительно (на 2-3 АК в основном при длине в порядка 15 АК). Сигнальной последовательности программой DeepTMHMM также не было предсказано.