ПРАКТИКУМ 11
1, 2. Выбор и описание семейства доменов
Я решил работать с семейством доменов MFS_MOT1 (код доступа PF16983). Эти домены играют ключевую роль в работе транспортеров, отвечающих за перенос молибдена (в виде молибдат-аниона) через цитоплазматическую мембрану у эукариот. Этот элемент, хотя и содержится в живых организмах в крайне малых количествах, входит в состав активных центров ферментов, участвующих в таких процессах, как метаболизм азота, биосинтез фитогормонов у растений, метаболизм пуринов и детоксикация сульфитов.
Выравнивание full белков, содержащих домены этого семейства, содержит 2386 последовательностей, тогда как в выравнивании seed их 151. Белки с доменами семейства MFS_MOT1 можно поделить на группы в соответствии с 11 различными архитектурами. При этом достаточно хорошо представлены лишь 2 из них: MFS_MOT1 x 2 (1141 белок) и MFS_MOT1 (77 белков). Можно заметить, что для большинства белков (кроме 77 с архитектурой MFS_MOT1) характерно наличие двух доменов семейства MFS_MOT, разделенных несколькими трансмембранными участками. Ни для одного белка, содержащего домены из исследуемого семейства, неизвестна 3D-структура (хотя для некоторых из них она была предсказана, например, UniProt ID MOT1_ARATH). Обсуждаемые белки встречаются у представителей всех 3 доменов: Archaea (91 последовательность), Bacteria (165) и Eukaryota (975). HMM профиль выравнивания был создан 21 октября 2021 года и имеет 111 позиций.
3. Карта локального сходства белков с разной доменной архитектурой
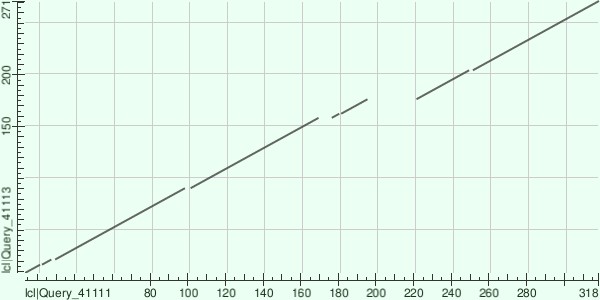
Для выполнения задания я выбрал белок MOT1_ARATH с доменний архитектурой MFS_MOT1 x 2 и белок A0A364NGX4_9PLEO с архитектурой MFS_MOT1. В парное локальное выравнивание вошли аминокислотные остатки с 14 по 318 первого белка (первый с N-конца домен семейства MFS_MOT1) и остатки с 9 по 271 второго, на его основании была построена карта локального сходства (Рис. 1). По горизонтальной оси отложены остатки белка MOT1_ARATH, а по вертикальной - белка A0A364NGX4_9PLEO. "Разрывы" в линии на карте соответствуют инделям в выровненной последовательности второго. Судя по всему, в ходе эволюции произошло несколько инсерций в гене белка MOT1_ARATH и/или несколько делеций в последовательности A0A364NGX4_9PLEO (оба изменения, по сути, ведут к одному результату). Для определения конкретных событий необходимо также провести анализ предковой последовательности, включив ее в выравнивание.
4. Выделение подгрупп доменов семейства MFS_MOT1
Я скачал выравнивание seed (151 последовательность) по АС (PF16983) через Jalview и открыл его. Чтобы уменьшить число строк, я удалил избыточные последовательности (совпадающие на 80%). В выравнивании их осталось 109. Для выделения групп было построено филогенетическое дерево, отражающее эволюцию последовательностей. В результате, они разбились на 4 явные и достаточно крупные группы (далее - группы 1, 2, 3 и 4). Только 3 последовательности не вошли ни в одну из них.
По итоговому выравниванию видно, что домены группы 1 похожи на домены группы 2, а последовательности группы 3 - на таковые из группы 4. Я решил сравнить первые две группы, противопоставив их оставшимся.
Домены групп 1 и 2 похожи друг на друга по количеству и положению гэпов в выравнивании, однако в их последовательностях есть несколько заметных отличий. Например, в доменах первой группы в позиции 14 помимо лейцина и изолейцина достаточно часто встречается треонин. В колонках 15 и 17 у них стоят очень консервативные остатки лейцина и серина, гомологичные соответственно глицину и аланину у большинства доменов группы 2. Более полный список различий я оформил в виде таблицы (Табл. 1).
| Колонка: | 14 | 15 | 17 | 21 | 43 | 82 | 85 | 114 | 123 | 134 |
|---|---|---|---|---|---|---|---|---|---|---|
| Группа 1 | T, L, I | L | S | V, ... | -, P, ... | V, P, G | A, ... | Q, H | S, T, ... | K, L |
| Группа 2 | L, I, ... | G, T, ... | A, S | T, ... | V, ... | P, ... | G, A | H, ... | T, S, G | ... |
Различия между парами групп гораздо глубже. Например, консервативному остатку глицина в позиции 9 доменов групп 3 и 4 соответствуют пролин и другие аминокислоты у белков первых двух групп, а остаткам аспартата и пролина в колонках 9 и 16 гомологичны остатки глутамина и аспарагина.
Более того, эти различия состоят как в гомологичных аминокислотных остатках, так и в распределении инделей. В частности, в большинстве последовательностей групп 3 и 4 нет остатков, гомологичных таковым из колонок 12, 31 - 37, 40 - 43, 53, 105 - 107 и 145 - 146 в группах 1 и 2. При этом обратное верно для колонок 54, 56, 57, 90 - 94, 112, 118 и 119.
5. Таблица со всеми белками из Uniprot с доменом семейства MFS_MOT1
Чтобы найти в UniProt все белки с доменами из семейства MFS_MOT1, я использовал данный запрос:database:(type:pfam pf16983)
Различия в систематике организмов, имеющих такие белки, имеются уже на уровне домена (superkingdom).