ПРАКТИКУМ 8
Общая информация о белке и органзме
Данный идентификатор соответствует цикло(L-лейцил-L-лейцил)-синтазе YvmC бактерии Bacillus licheniformis (UniProt ID: CDLS_BACLD, AC: Q65EX3). Этот фермент катализирует реакцию синтеза вторичного метаболита циклодилейцина, используя в качестве субстрата 2 молекулы лейцил-тРНК. Данный циклодипептид является биохимическим предшественником железосодержащего внеклеточного красного пигмента пульхерримина (Bonnefond et al., 2011). Bacillus licheniformis – почвенная спорообразующая бактерия, используемая в биотехнологии для получения ряда ферментов и антибиотиков (Rey et al., 2004).
Сведения о белке
Цикло(L-лейцил-L-лейцил) синтаза (циклодилейцин-синтаза) бактерии Bacillus licheniformis - мономерный белок с молекулярной массой 28550 Da, состоящий из 249 аминокислотных остатков. Классификация фермента: EC 2.3.2.22 (трансфераза). Как упоминалось выше, функция фермента состоит в АТФ-независимом биосинтезе циклодилейцина (cLL) из лейцил-тРНК. Данная реакция является частью метаболического пути, приводящего к синтезу пигмента пульхерримина. Фермент может также включать в состав циклодипептидов похожие на L-лейцин аминокислоты с неполярными боковыми радикалами. В первичной структуре фермента нет нестандартных аминокислот, посттрансляционные модификации отсутствуют, при сворачивании белка не образуются дисульфидные мостики между остатками цистеина. Активный сайт сформирован остатком 37, возможно, остатки 40 и 184 также играют важную роль в каталитической активности. За связивание субстрата отвечают участки с 180 по 184 и с 209 по 210 остаток, а также остатки 40 и 204 и, возможно, 88, 98 и 180. 3D-структура фермента определена экспериментально методом рентгеновской кристаллографии. Во вторичной структуре белка участки параллельного бета-листа чередуются с альфа-спиралями и неструктурированными петлями. Запись Q65EX3 (Swiss-Prot) входт в состав кластеров UniRef100_Q65EX3 (3 записи), UniRef90_Q65EX3 (31 запись) и UniRef100_Q65EX3 (190 записей) в качестве репрезентативной последовательности. При этом сидом в этих кластерах является запись UniParc UPI0001FB33EC (предполагаемый неохарактеризованный белок yvmC, 257 аминокислотных остатков).
Распространенность и эволюция белка
Для оценки распространенности цикло(L-лейцил-L-лейцил) синтаз был составлен запрос:
name:"cyclodileucine synthetase" OR name:"cyclo l-leucyl-l-leucyl synthase"Выдача состояла из 10 записей (5 аннотированных из Swiss-Prot). При этом среди них нашлись белки эукариот:
(name:"cyclodileucine synthetase" OR name:"cyclo l-leucyl-l-leucyl synthase") taxonomy:"Eukaryota [2759]"
По данному запросу нашлись 2 записи из TrEMBL с идентификаторами A0A6S7GTI0 и A0A1V2L8H3. Они соответствуют цикло(L-лейцил-L-лейцил) синтазам горгонарии Paramuricea clavata и дрожжей Cyberlindnera fabianii соответственно. Наличие гомологии между бактериальной и эукариотическими версиями фермента было проверено с помощью запроса:
(name:"cyclodileucine synthetase" OR name:"cyclo l-leucyl-l-leucyl synthase") cluster:(uniprot:q65ex3 identity:0.5) taxonomy:"Eukaryota [2759]"
В выдаче по этому запросу не было ни одной записи. Соответственно, либо эукариотические ферменты вовсе не гомологичны прокариотическим, либо горизонтальный перенос генов цикло(L-лейцил-L-лейцил)-синтазы произошел в этих случаях достаточно давно, и с того времени они успели достаточно сильно измениться.
Далее я решил составить запрос, позволяющий примерно оценить древность исследуемого фермента и похожих на него белков:
(cluster:(uniprot:q65ex3 identity:0.5) NOT cluster:(uniprot:q65ex3 identity:0.9)) NOT taxonomy:"Bacillus [1386]"
Его целью является определение списка таксонов (в данном случае, родов), представители которых, вероятно, получили ген этого белка от общего предка. Второе условие должно исключить организмы, которым он достался относительно недавно путем горизонтального переноса. Третье условие было добавлено для удобства обработки результатов, так как без него большую часть выдачи составляют белки бактерий из рода Bacillus. В итоге, предполагаемые ортологи исследуемого белка были найдены у представителей 6 родов: Alkalihalobacillus, Bacillus, Caldibacillus, Geobacillus, Paenibacillus и Priestia. 5 из них входят в состав семейства Bacillaceae, и все они являются частью порядка Bacillales. Таким образом, можно предположить, что предковый белок возник в пределах этих таксонов.
Задание 1
В качестве исследуемого протеома выбран референсный протеом с идентификатором UP000000606 размером в 4164 белка, принадлежащий Bacillus licheniformis (strain ATCC 14580 / DSM 13 / JCM 2505 / CCUG 7422 / NBRC 12200 / NCIMB 9375 / NCTC 10341 / NRRL NRS-1264 / Gibson 46).
Я буду сравнивать его с референсным протеомом UP000001355 бактерии Bacillus pumilus (strain SAFR-032), состоящим из 3700 белков. Эта непатогенная спорообразующая бактерия интересна прежде всего своей способностью выдерживать дозы ультрафиолетового излучения и концентрации пероксида водорода, которые являются смертельными для многих других микроорганизмов (Stepanov et al., 2016). На этом различии и будет сосредоточено сравнение.
Оба протеома достаточно хорошо изучены. Для протеома Bacillus licheniformis количество записей в Swiss-Prot составляет 565 (примерно 13,6% от общего числа записей), а для протеома Bacillus pumilus - 425 (11,5%), что несколько меньше. Первый имеет статус CPD "Standard", а второй - "Close to Standard", что указывает на возможную неполноту второго протеома. При этом для обоих протеомов сумма долей дуплицированных и представленных одной копией белков из референсных ортологичных групп (BUSCO: Single + Duplicated) превышает 99%, и составляет 99,8% для исследуемого протеома и 99,1% для контрольного протеома. Таким образом, протеом Bacillus licheniformis все же изучен несколько лучше.
Для скачивания протеомов были использованы следующие команды bash:
curl 'https://www.uniprot.org/uniprot/?query=proteome:up000000606&format=txt&compress=yes' > UP000000606.swiss.gz
curl 'https://www.uniprot.org/uniprot/?query=proteome:up000001355&format=txt&compress=yes' > UP000001355.swiss.gz
Задание 2
Для сравнения долей трансмембранных белков были составлены запросы:
annotation:(type:transmem) proteome:up000000606
annotation:(type:transmem) proteome:up000001355
Количество трансмембранных белков в протеоме B. licheniformis составляет 994 (23,9% от общего числа), а в протеоме B. pumilus - 896 (24,2%). В плане доли трансмембранных белков различия между протеомами, в целом, незначительны.
Чтобы оценить доли ферментов, я использовал следующие запросы:
ec:* proteome:up000000606
ec:* proteome:up000001355
В протеоме исследуемой бактерии 766 белков (18,4% белков) являются ферментами, а в случае контрольного организма число ферментов составляет 712 (19,2%). Эти значения также отличаются незначительно.
Для определения возможной причины аномальной устойчивости B. pumilus (strain SAFR-032) я решил сравнить также количества белков, которые могли бы ее обеспечивать. В моем случае, это белки, участвующие в процессах репарации ДНК и спорообразования. Поиск белков я проводил на основе присутствия соответствующих ключевых слов (по полю KW):
keyword:"DNA repair [KW-0234]" proteome:up000000606
keyword:"Sporulation [KW-0749]" proteome:up000001355
Судя по результатам запросов, оба вида обладают одинаковым количеством белков, участвующих в репарации ДНК (всего 35 белков) и примерно равным числом белков, задействованных в образовании спор (33 у B. licheniformis и 30 у B. pumilus). Видимо, причина различия между ними кроется не в количестве этих белков, а в особенностях их структуры и функционорования, или она вообще никак не связана с вышеупомянутыми процессами.
Задание 3
Далее я решил сравнить количество и долю оксидоредуктаз (от общего количества ферментов) в протеомах исследуемых бактерий, а также распределение этих ферментов по подклассам. Отличия в степени представленности этих белков могли бы объяснить разную приспособленность бактерий к высоким концентрациям активных форм кислорода.
Для этого была написана программа на языке python, доступная по ссылке в GoogleColab:

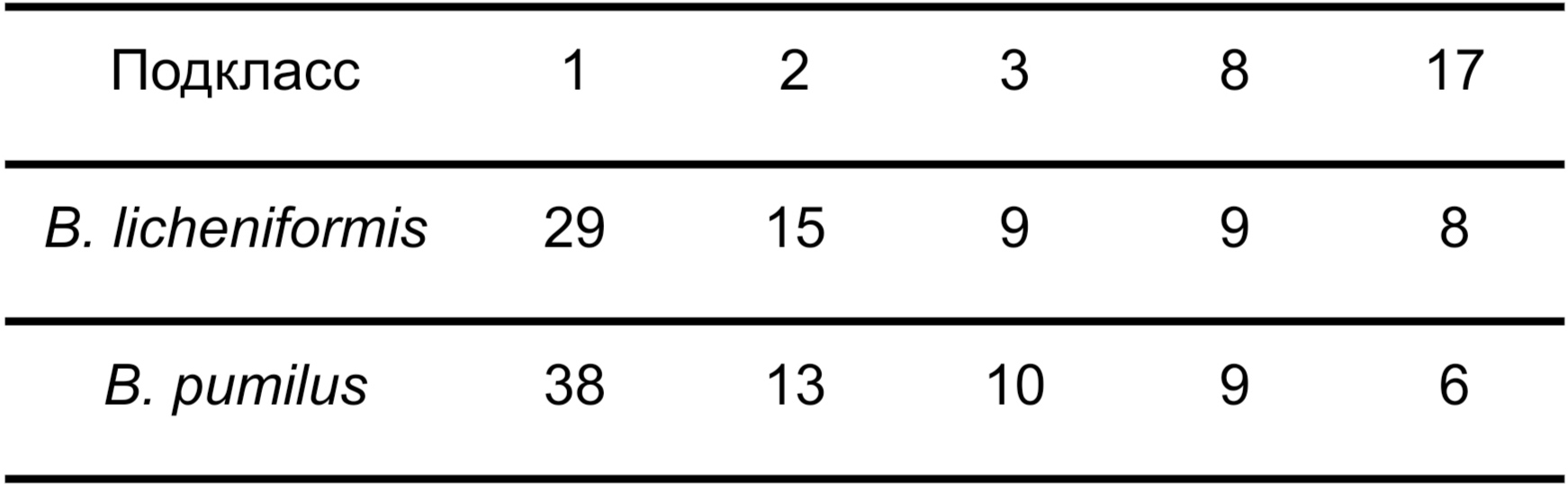
Доли и количества оксидоредуктаз оказались примерно равными (114, 14,9% у B. licheniformis и 112, 15,7% у B. pumilus). В целом, похожи и степени представленности различных подклассов (Табл. 1), однако у B. pumilus в протеоме значительно больше оксидоредуктаз из подкласса 1 (33,9% от числа всех оксидоредуктаз; 25,4% у B. licheniformis). Характерной особенностью данных ферментов является использование CH2-OH групп в качестве восстановителей. Возможно, этот факт имеет какое-то отношение к высокой устойчивости B. pumilus (strain SAFR-032), однако прямую связь между этими явлениями установить сложно.
Ссылки на источники
Bonnefond, L., Arai, T., Sakaguchi, Y., Suzuki, T., Ishitani, R., & Nureki, O. (2011). Structural basis for nonribosomal peptide synthesis by an aminoacyl-tRNA synthetase paralog. Proceedings of the National Academy of Sciences of the United States of America, 108(10), 3912–3917. ссылка на статью
Rey, M. W., Ramaiya, P., Nelson, B. A., Brody-Karpin, S. D., Zaretsky, E. J., Tang, M., Lopez de Leon, A., Xiang, H., Gusti, V., Clausen, I. G., Olsen, P. B., Rasmussen, M. D., Andersen, J. T., Jørgensen, P. L., Larsen, T. S., Sorokin, A., Bolotin, A., Lapidus, A., Galleron, N., Ehrlich, S. D., … Berka, R. M. (2004). Complete genome sequence of the industrial bacterium Bacillus licheniformis and comparisons with closely related Bacillus species. Genome biology, 5(10), R77. ссылка на статью
Stepanov, V. G., Tirumalai, M. R., Montazari, S., Checinska, A., Venkateswaran, K., & Fox, G. E. (2016). Bacillus pumilus SAFR-032 Genome Revisited: Sequence Update and Re-Annotation. PloS one, 11(6), e0157331. ссылка на статью