Практикум 10
Выбранные последовательности
В ходе поиска подходящих последовательностей среди полных геномов бактерий я обнаружил, что для многих таксонов их схожесть слабо соотносится с эволюционной близостью рассматриваемых организмов. Для некоторых родов геномы разных видов оказывались крайне схожими, но бывали и случаи, когда геномы разных штаммов одного вида выравнивались очень плохо. Гораздо удобнее было бы работать с большими таксонами и консервативными последовательностями.
В частности, оказалось, что геномы пластид высших растений по последовательности расположения участков почти идентичны даже для представителей разных семейств одного порядка. Поэтому в данном практикуме я решил работать с пластидной ДНК. Я взял за отправную точку полный геном пластиды двудольного покрытосеменного растения Arabidopsis thaliana (сем. Крестоцветные) и выравнивал его с пластидной ДНК других высших растений по мере понижения родства.
Для поиска последовательностей я пользовался сервисом "Browse by Organism" на сайте NCBI, установив фильтр на уровень сборки (chromosome+). В качестве запросов я использовал видовые названия организмов, а также названия более крупных таксонов, например:
Arabidopsis thaliana
Bryophyta
Выравнивания я строил с помощью BLAST 2 sequences на сайте NCBI, оттуда же я скачивал карты локального сходства. Для поиска подходящих последовательностей я использовал алгоритм blastn с параметрами по умолчанию. Для выбранных геномов я построил 2 карты локального сходства: по выдаче алгоритма blastn (размер слова 7, порог на e-value 0.01) и по выдаче megablast (размер слова 32, порог на e-value 0.05)
Я решил описать карты локального сходства геномов пластид Arabidopsis thaliana (Nucleotide: NC_000932.1) и мха Physcomitrium patens (NC_005087.1)
Описание карты локального сходства
Далее приведены подробное описание диаграммы DotPlot, полученной для рассматриваемых последовательностей из выдачи алгоритма blastn (Рис. 1), а также предположительная последовательность событий в эволюции организации пластидного генома общего предка Arabidopsis thaliana и Physcomitrium patens.
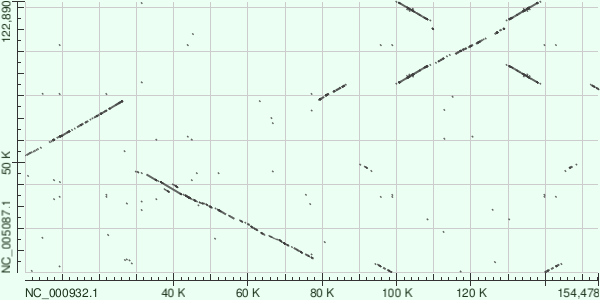
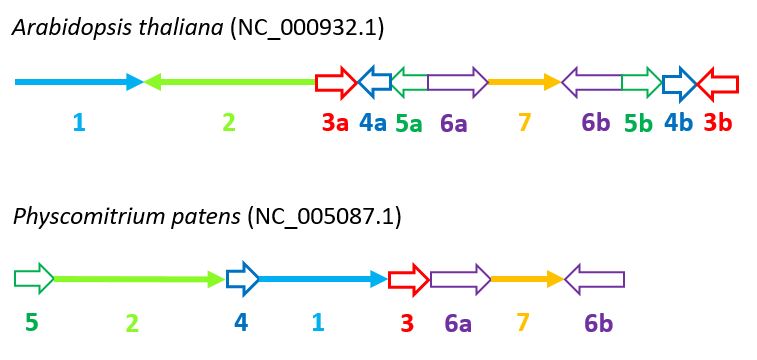
Для удобства отображения последовательности перестроек я решил спроектировать линии диаграммы, соответствующие участкам локального сходства, на оси (последовательности пластидных геномов). Полученным отрезкам я придал направление в соответствии с их относительной ориентацией (в данном случае речь идет именно об относительном направлении, которое для каждой группы соответствующих друг другу участков можно задать по-разному). Полученные участки я пронумеровал (Рис. 2).
Инвертированные повторы
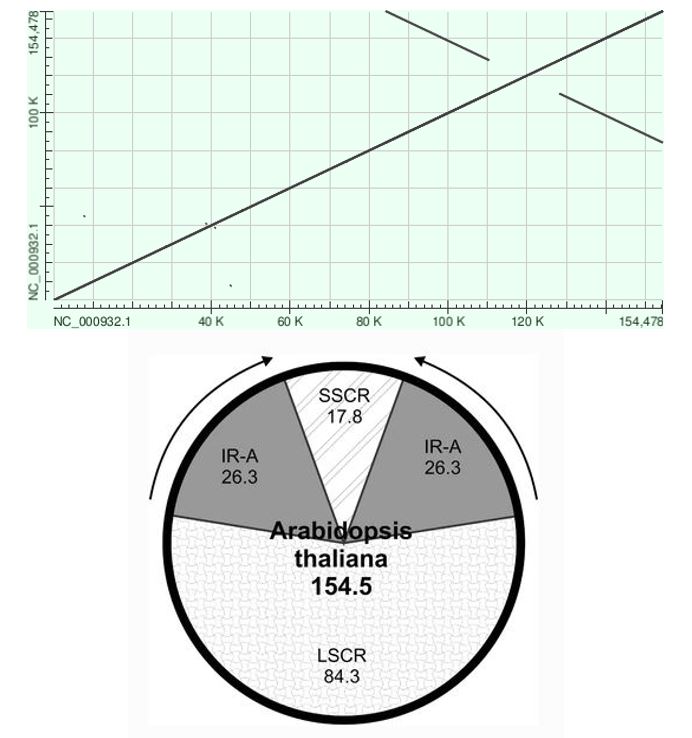
Для начала следует обратить внимание на крестообразную структуру в области 100 - 140 Kb по оси x. Каждая из пар смежных "плеч" креста проецируется на один участок одной из осей и на два участка другой (участки 6 на схеме, Рис. 2). Таким образом, можно сделать вывод, что в каждой из последовательностей есть по 2 инвертированных повтора. Это особенно заметно на диаграмме DotPlot для двух одинаковых последовательностей (Рис. 3). На самом деле, их наличие является общей чертой геномов пластид почти всех исследованных в этом плане высших растений, а также большого числа водорослей. Эти повторы содержат в себе гены рРНК. (Olejniczak et al., 2016)
Различные точки начала
Пластидный геном представлен кольцевой молекулой ДНК, поэтому технически запись его последовательности можно начать с любой точки. В данном случае последовательности геномов отличаются положением и ориентацией участка одного из участков 5, который в геноме Arabidopsis thaliana следует за вторым по счету участком 6, а в геноме Physcomitrium patens стоит в начале последовательности. Учитывая, что у второго организма отсутствует пара сегментов 4 и 3 между упомянутыми участками, можно можно заключить, что точка начала последовательности в двух геномах была выбрана немного по-разному.
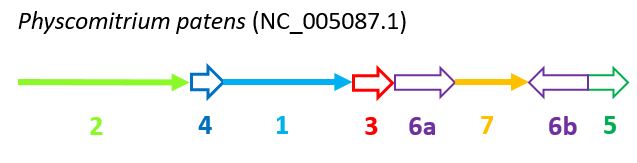
Схема пластидного генома Physcomitrium patens, приведенная в соответствие с таковой для Arabidopsis thaliana, представлена ниже (Рис. 4).
Делеции в инвертированных повторах
У Physcomitrium patens инвертированные повторы полностью совпадают с участками 6. В то же время у Arabidopsis thaliana, пусть это и плохо видно на карте локального сходства, повторы по сути включают также сегменты 3, 4 и 5. Причем вероятнее всего, что именно это состояние является исходным, а в линии, ведущей от общего предка двух организмов к Physcomitrium patens, произошли делеции в инвертированных повторах, приведшие к исчезновению первых по счету участков 4 и 5 и второго участка 3 (Рис. 5).
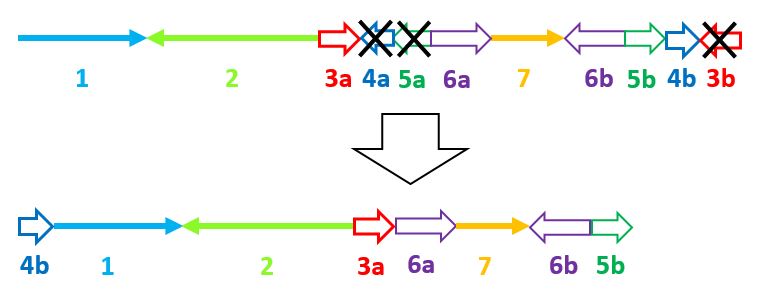
Транслокации и инверсии
Интересно, что при делеции сегмента 3 в составе второго инвертированного повтора образуется блок из участка 1 и второго участка 4, который присутствует в пластидном геноме Arabidopsis thaliana в неизменном виде, однако отличается по положению.
Таким образом, если положить, что оба сегмента 4 изначально входили в состав инвертированных повторов, можно сделать вывод, что изменение порядка расположения участков 1, 2 и 4 также происходило в линии Physcomitrium patens, причем после делеции участка 3.
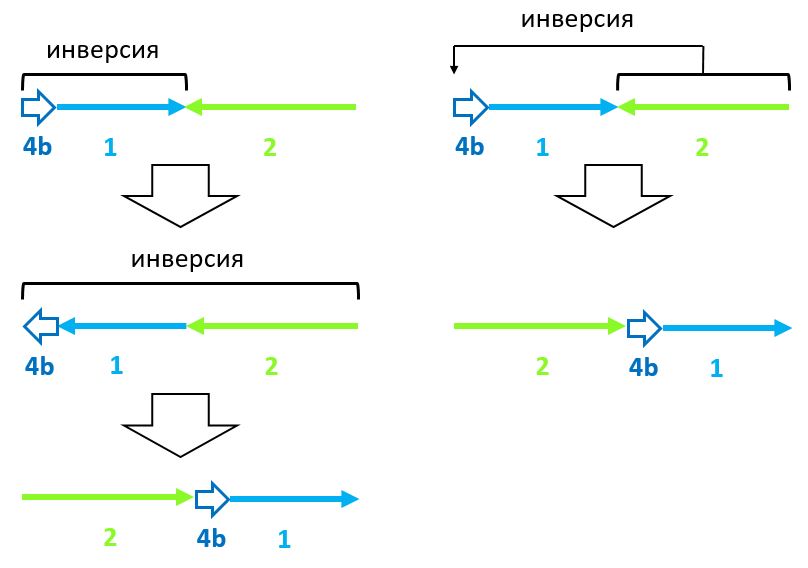
Стоит отметить, что эта перестановка может быть достигнута как минимум двумя различными способами: транслокацией участка 2 с изменением его ориентации или двумя последовательными инверсиями (Рис. 6).
Сравнение выдачи алгоритма megablast с выдачей blastn
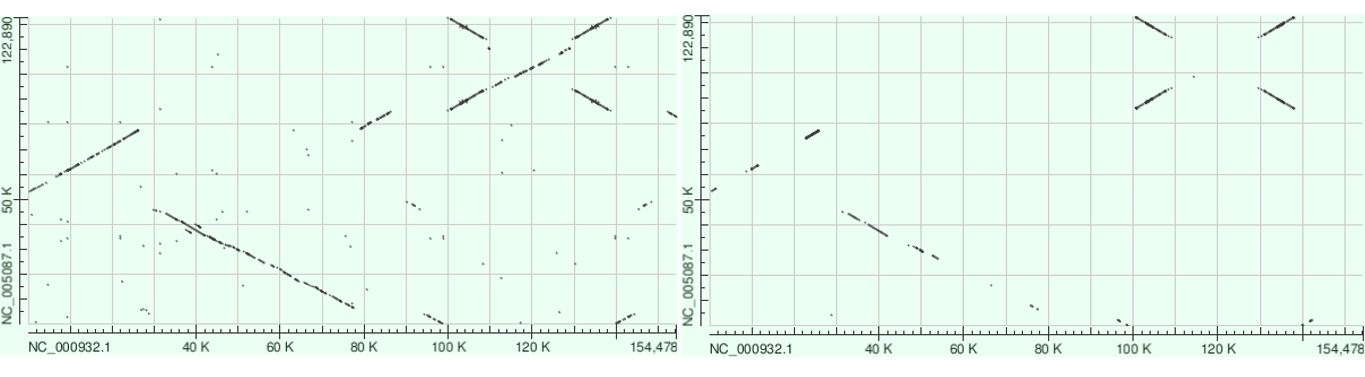
Из-за линейных штрафов за гэпы и большей длины слова алгоритм megablast является гораздо менее чувствительным, чем blastn. Таким образом, он может пропускать хорошие локальные выравнивания, что снижает покрытие последовательности запроса. Соответственно, линии, отвечающие участкам локального сходства между последовательностями, обычно являются гораздо более длинными, четкими и многочисленными на диаграмме DotPlot, построенной по выдаче алгоритма blastn.
Эта общая закономерность соблюдается и в данном случае. На карте локального сходства, полученной с использованием алгоритма megablast, линии, соответствующие участкам 1, 2 и 5, фрагментированы на отдельные отрезки, а линии, отвечающие сегментам 3, 4 и 7 и вовсе отсутствуют. Нет на этой диаграмме и коротких отрезков ("точек"), соответствующих небольшим повторам, распределенным по последовательностям геномов. При переходе от blastn к megablast незначительные изменения притерпевают только линии участков 6 в составе инвертированных повторов. Вероятно, это объясняется повышенной консервативностью содержащихся в них генов рРНК.
Ссылки на источники
Olejniczak, S.A., Łojewska, E., Kowalczyk, T. et al. Chloroplasts: state of research and practical applications of plastome sequencing. Planta 244, 517–527 (2016). https://doi.org/10.1007/s00425-016-2551-1