Практикум 15
1. Подготовка чтений с помощью Trimmomatic
В данном практикуме я работал с SRA ID SRR4240378. Для скачивания файла с чтениями я использовал команду wget:
wget "ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/008/SRR4240378/SRR4240378.fastq.gz"
Перед удалением адаптеров я решил собрать все их последовательности в один файл (adapters.fasta). Для этого была выполнена следующая команда:
seqret "/mnt/scratch/NGS/adapters/*" "fasta::adapters.fasta"
Далее для имеющихся чтений была два раза запущена программа Trimmomatic. Сначала были удалены остатки адаптеров:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240378.fastq.gz SRR4240378_noadapters.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7 2> adapters_log.txt
При этом, согласно выдаче программы (файл adapters_log.txt), количество чтений уменьшилось с 4420587 до 4338744, а 81843 (1.85% от изначального числа чтений) было удалено. Исходный файл имел размер 91Мб, для получившегося же файла он составил 89Мб.
Затем с конца чтений были удалены основания с качеством ниже 20, после чего слишком короткие чтения (короче 32 оснований) были отброшены:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240378_noadapters.fastq.gz SRR4240378_trimmed_noadapters.fastq.gz TRAILING:20 MINLEN:32 2> trimming_log.txt
После этого шага число чтений сократилось до 4154738, то есть было удалено еще 184006 (4.24%). Размер файла при этом уменьшился до 84Mб.
2. Подготовка k-меров с помощью velveth.
Для формирования списка k-меров (в данном случае, длины 31), необходимого для сборки генома, была использована программа velveth:
velveth ./velvet 31 -fastq.gz -short SRR4240378_trimmed_noadapters.fastq.gz
3. Сборка контигов с помощью velvetg
Собственно сборка генома (на уровне отдельных контигов) была проведена с помощью программы velvetg:
velvetg ./velvet > velvetg_log.txt
Исходя из текстовой выдачи программы, полученная сборка состояла из 369 контигов, а показатель N50 для нее составил 7028.
Информацию об отдельных контигах я брал из файла stats.txt, созданного программой velvetg, а их последовательности получал из файла contigs.fa с помощью команды seqret:
seqret "fasta::contigs.fa:NODE_129_*" "fasta::contig_129.fasta"
Пользуясь функциями текстовых таблиц (Google spreadsheets), я отсортировал контиги по их длине и покрытию.
Контиги с наибольшей длиной имеют номера 8 (длина: 36746, покрытие: 20.017199), 57 (длина: 19371, покрытие: 20.546642) и 15 (длина: 16745, покрытие: 20.901762).
Некоторые контиги обладали аномально большим покрытием. В частности, для контига 129 оно составило 148170. Было также и несколько контигов длиной в 1 k-мер с покрытием, равным 1 (k-меры, встретившиеся в последовательностях чтений только 1 раз). Однако ни для одного из описанных контигов не было последовательностей в файле contigs.fa. Это связано с тем, что по умолчанию туда включаются только контиги с длиной более 2k. Это отсеивает в первую очередь именно контиги со сравнительно большими и маленькими значениями покрытия (Рис. 1).
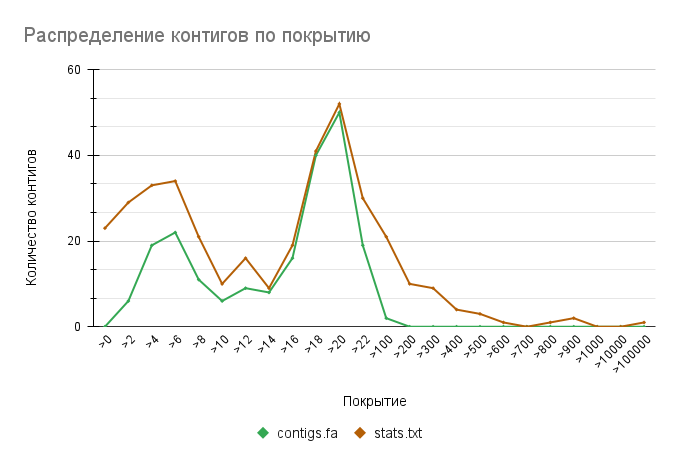
Для получения информации о длине и покрытии контигов, вошедших в файл contigs.fa, я использовал следующий конвейер:
grep "^>" contigs.fa | cut -d "_" -f 2,4,6 | tr "_" "\t" > correct_stats.txt
Полученные данные я также импортировал в текстовую таблицу и отсортировал. Для контигов с известной последовательностью уже не было аномальных значений покрытия. Наибольшее значение составило 102,74839 (для контига 81), а наименьшее - 2,84375 (контиг 166).
4. Анализ контигов с помощью BLAST
Самые длинные контиги (8, 59 и 15) были выравнены с последовательностью генома бактерии Buchnera aphidicola (EMBL AC CP009253) с помощью BLAST на сайте NCBI. Для этого я использовал алгоритм megablast с параметрами по умолчанию. Ниже представлены соответствующие карты локального сходства и таблицы с характеристиками всех выравниваний для каждого из контигов.
Контиг 8
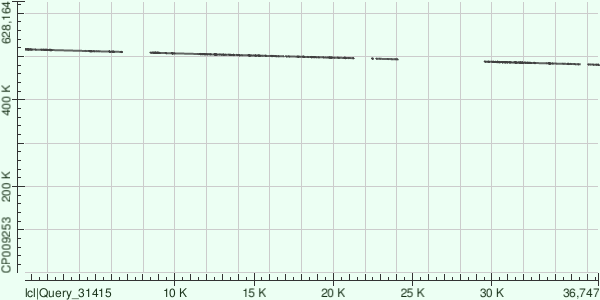
| Участок генома | Процент идентичности |
Процент гэпов |
|---|---|---|
| 500370 - 508806 | 76% | 4% |
| 510438 - 516539 | 79% | 2% |
| 481997 - 488106 | 74% | 4% |
| 496111 - 500325 | 75% | 3% |
| 493487 - 494864 | 80% | 0% |
| 480874 - 481545 | 82% | 2% |
| 495033 - 495148 | 90% | 4% |
Контиг 57
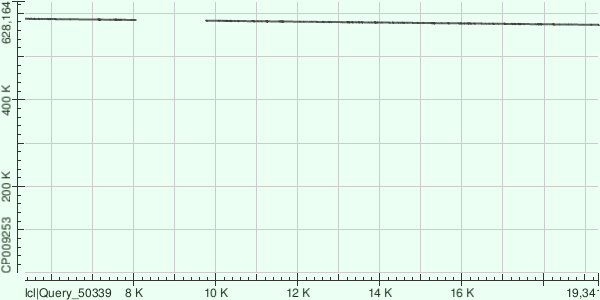
| Участок генома | Процент идентичности |
Процент гэпов |
|---|---|---|
| 573092 - 582686 | 73% | 4% |
| 584329 - 587055 | 76% | 3% |
Контиг 15
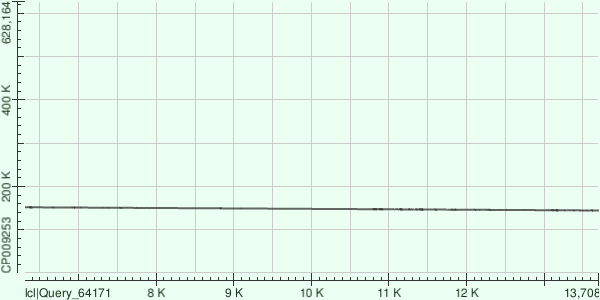
| Участок генома | Процент идентичности |
Процент гэпов |
|---|---|---|
| 144368 - 151796 | 78% | 3% |
Ожидаемо, самое большое покрытие имело выравнивание для самого длинного, 8-го контига (Табл. 4). Он же "лидирует" и по общему весу выравниваний, которых для данного контига больше всего. При этом для самого короткого, 15-го контига выравнивание только одно.
| Номер контига | Длина контига (bp) | Покрытие | Общий вес | Процент идентичности |
|---|---|---|---|---|
| 8 | 36776 | 73% | 13826 | 75.62% |
| 57 | 19401 | 63% | 4532 | 73.43% |
| 15 | 16775 | 44% | 4423 | 77.80% |