Практикум 3
Задание 1
С помощью программы fiber пакета 3DNA я построил 3 формы ДНК-дуплекса с последовательностью (GATC)x5 ((GC)x5 для Z-формы). Для этого на kodomo были выполнены следующие команды:
fiber -seq=GATC -rep=5 -a gatc-a.pdb
fiber -seq=GATC -rep=5 -b gatc-b.pdb
fiber -seq=GATC -rep=5 -z gatc-z.pdb
Ссылки на скачивание файлов:
Задание 2
Для выполнения этого и последующих заданий я использовал пакет nglview в JupyterLab на kodomo (jupyter notebook можно скачать по ссылке).
Для описания я выбрал тимин в позиции 7 цепи А структуры PDB 1BNA (B-форма). В программе MarvinSketch я нарисовал структурную формулу тимина, после чего покрасил атомы, обращенные в сторону большой бороздки в красный цвет, а атомы, обращенные в сторону малой, - в синий (Рис. 1).
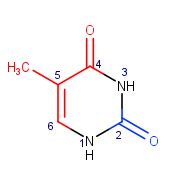
- Атомы, обращенные в сторону большой бороздки: [DT]7:A.C4, [DT]7:A.C5, [DT]7:A.O4, [DT]7:A.C7, [DT]7:A.C6.
- Атомы, обращенные в сторону малой бороздки: [DT]7:A.C2, [DT]7:A.O2.
Далее я визуализировал в jupyter notebook полученные с помощью программы fiber структуры (Рис. 2).
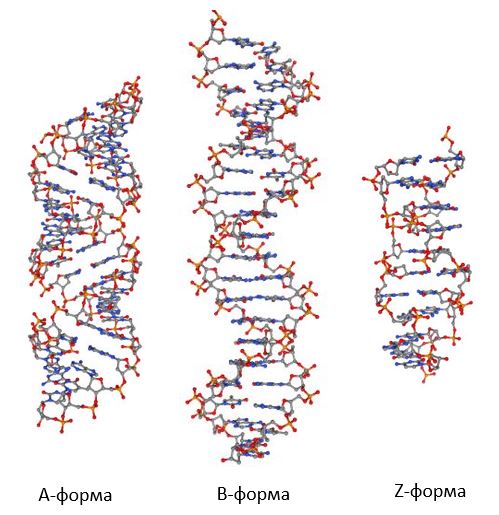
Пользуясь функциями nglview, я измерил для них различные параметры, представив результат в виде таблицы (Табл. 1). Количество нуклеотидных остатков в витке спирали для Z-формы оказалось больше 10, поэтому была создана еще одна структура с большей длиной цепи:
fiber -seq=GC -rep=15 -z longer_z.pdb
| A-форма | В-форма | Z-форма | |
|---|---|---|---|
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28,03 | 33,75 | 43,50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Å) | 7,98; [DA]10:A.P-[DG]25:B.P |
17,21; [DA]10:A.P-[DC]28:B.P |
7,20; [DG]7:A.P-[DG]17:B.P |
| Ширина малой бороздки (Å) | 16,81; [DA]10:A.P-[DG]33:B.P |
11,69; [DA]10:A.P-[DT]35:B.P |
18,30; [DC]2:A.P-[DC]16:B.P |
Ширина большой и малой бороздок измерялась от атома [DA]10:A.P для А- и В-форм и от атомов [DG]7:A.P и [DC]2:A.P соответственно для Z-формы.
Задание 3
Для анализа структуры тРНК я скачал из базы данных PDB файл 1o0b.pdb и выполнил для него конвейер:
find_pair -t 1o0b.pdb stdout | analyze
Упр.1.
В полученном файле 1o0b.out я нашел раздел, содержащий информацию о торсионных углах в нуклеотидных остатках молекулы, а также раздел с классификацией отдельных динуклеотидов в ее составе по структурной схожести с ДНК-дуплексом той или иной формы. Участки данной тРНК, образующие двойную спираль, по структуре ближе всего к А-форме.
Упр.2.

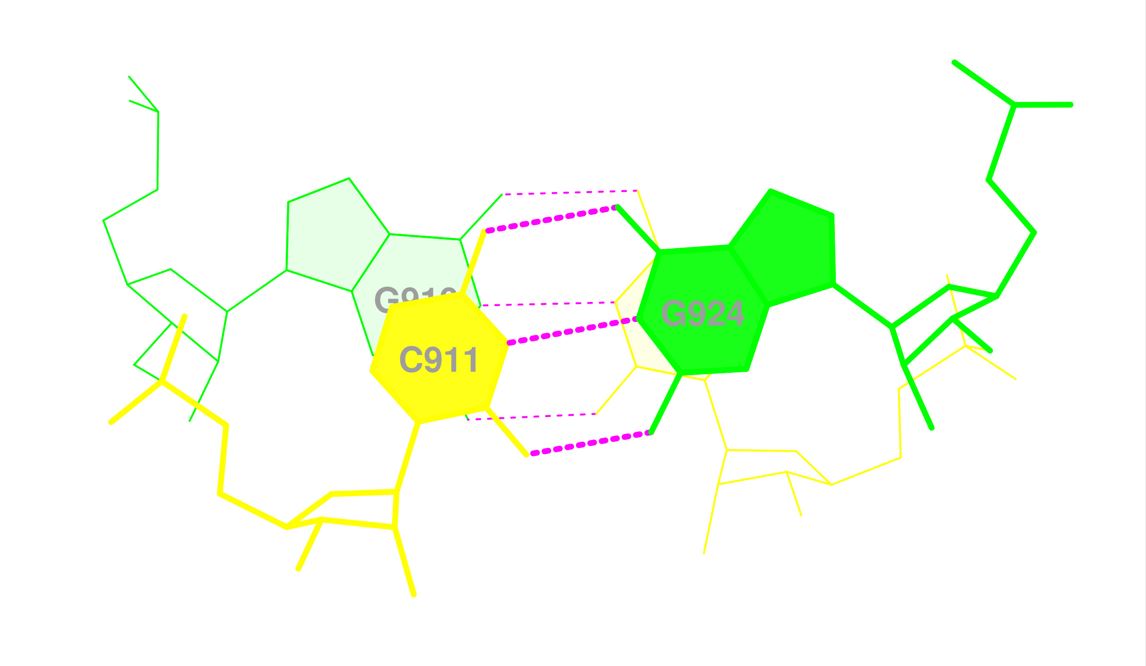
В структуре данной тРНК имеется 4 стебля (Рис. 3, стебли отмечены синими скобками) с координатами:
[G]902/[C]971-[A]907/[U]966; [C]949/[G]965-[G]953/[C]961; [A]937/[U]933-[C]944/[A]926; [G]910/[C]925-[C]912/[G]923
Эти координаты соответствуют номерам остатков в файле .pdb. На деле, 902-ой остаток в данном случае соответствует первому остатку в составе тРНК.
Вне стеблей образуются следующие пары азотистых оснований:
[U]954/[A]958; [U]955/[G]918; [A]913/[A]945; [A]914/[U]908; [G]915/[C]948; [G]919/[C]956.
В структуре данной тРНК имеется 9 неканонических пар (строки 12 - 15, 17, 21, 25 - 27 на Рис. 3).
Упр.3.
Перекрывание было наибольшим для динукдеотидов 11 (10,29), 20 (11,52) и 22 (11,52).
Я нашел в файле stacking.pdb соответствующие структуры, вырезал их и построил изображения (показано на примере 22-го шага):
ex_str -22 stacking.pdb step22.pdb
stack2img -cdolt step22.pdb step22.ps