Практикум 8
Задание 1


Для поиска мотивов из базы данных Pfam был выбран домен NifQ (AC: PF04891). Он входит в состав одноименного белка, участвующего в ранних стадиях биосинтеза железо-молибденового кофактора, который является важнейшей частью активного центра нитрогеназ - ферментов, осуществляющих восстановление молекулярного азота до аммиака (Siddavattam et al., 1993).
Выравнивание seed для данного домена содержит 50 последовательностей длиной порядка 160 аминокислотных остатков. При пороге percentage identity 100% окрашиваются несколько колонок во второй половине выравнивания, которые вместе с рядом менее консервативных позиций можно сгрупперовать в два мотива.
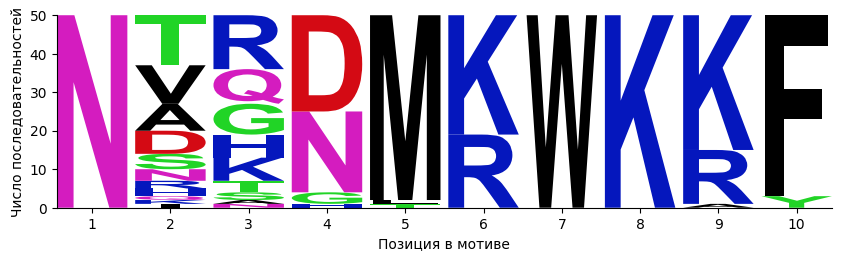
Первый из найденных мотивов (Рис. 1, 2) имеет следующие паттерны:
- Jalview
N.{4}[KR]WK[KRA][FY] - PROSITE (fuzzpro)
N-x(4)-[KR]-W-K-[KRA]-[FY]
Триптофан является достаточно редкой аминокислотой, при этом в данном случае он окружен сразу тремя положительно заряженными остатками. Наряду с высокой консервативностью некоторых позиций мотива это позволяет сделать предположение о его важной функциональной роли в белке.
Второй мотив (Рис. 3, 4) почти сразу следует за первым, однако очень сильно отличается от него по составу и организации:
- Jalview
C.{7,8}C.{2}P.C.{2}C.{5}C - PROSITE (fuzzpro)
C-x(7,8)-C-x(2)-P-x-C-x(2)-C-x(5)-C
По сути, мотив состоит из 5 остатков цистеина и одного остатка пролина, разделенных вариабельными участками фиксированной длины. Было показано, что атомы серы в остатках цистеина координируют ионы металла, что играет важную роль в работе белка (Siddavattam et al., 1993).

Из-за близости найденных мотивов в выравнивании (они разделяены всего 5 колонками) их можно чисто формально сгруппировать в один мотив, однако из-за больших структурных и, вероятно, функциональных различий между ними я решил рассматривать их по-отдельности.
Поиск по паттернам каждого из двух мотивов в Jalview дал 50 находок в соответствующих участках выравнивания.
Выдача от поиска в базе данных SwissProt по первому мотиву содержала 11 запиисей, из которых 4 принадлежали NifQ и его гомологам (NIFQ_SINFNб, NIFQ_KLEPNб, NIFQ_AZOVI и NIFQ_RHOCA), а остальные 6 не имели с ними видимой связи. Поиск по второму паттерну дал 20 белков, среди которых были все те же записи для NifQ наряду с двумя записями для металлотионеина-2 (цистеин-богатый белок, связывающий ионы тяжелых металлов) и 13 записями для разных форм кератина, для которого также характерно высокое содержание цистеина.
Задание 2
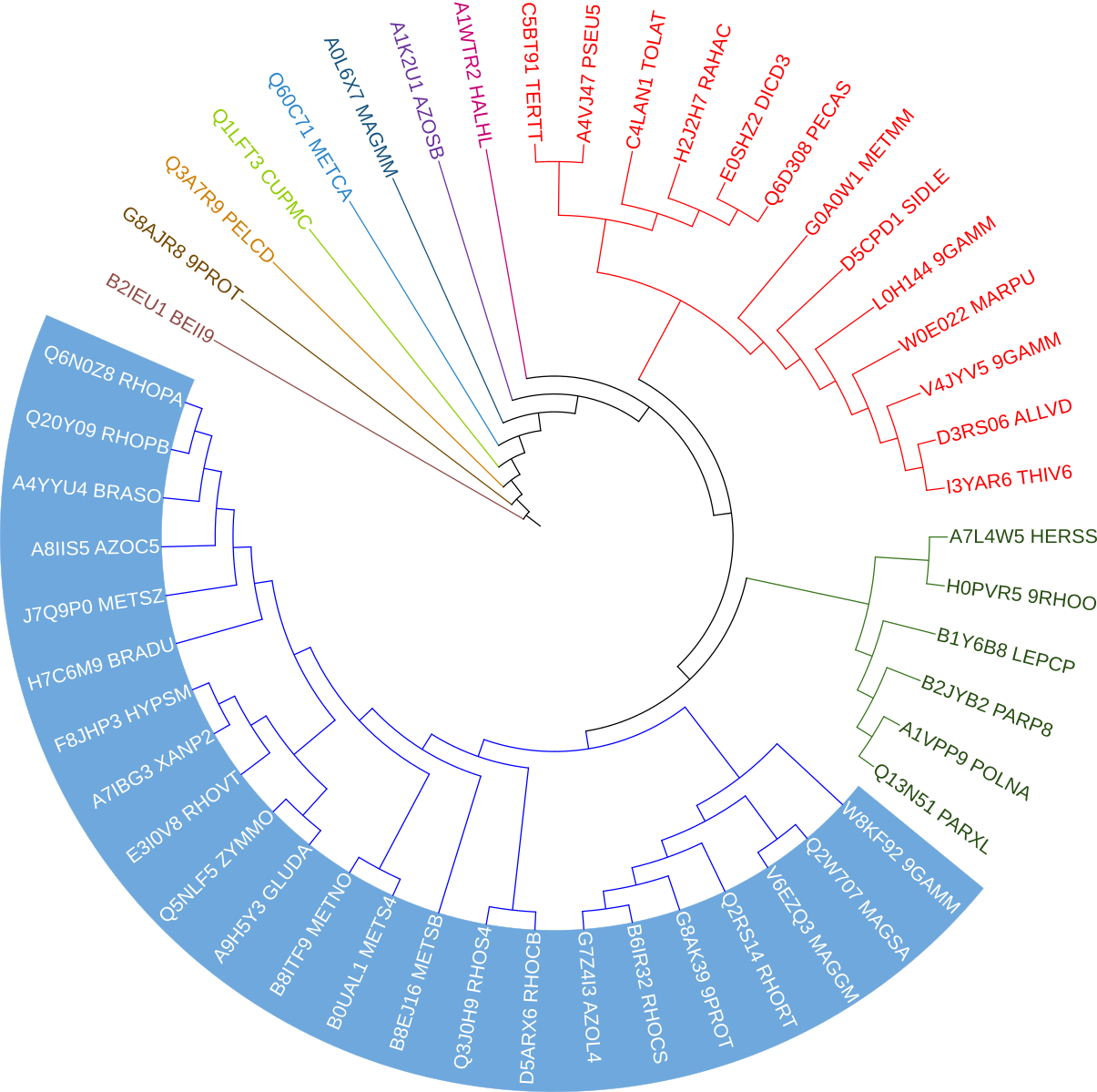
По выравниванию seed для домена NifQ было построено филогенетическое дерево методом Neighbour Joining (Рис. 5). После этого последовательности выравнивания были разбиты на группы в соответствии с его кладами. В 23 последовательностях, отмеченных на рисунке синей заливкой, в позициях 105 и 106 в выравнивании стоят два консервативных остатка аргинина. Легко заметить, что эти белки составляют обособленную кладу. В большинстве последовательностей других групп эти колонки заняты различными незаряженными остатками. Исключение составляют белки Q3A7R9_PELCD, G8AJR8_9PROT и G8AJR8_9PROT, у которых одна из вышеупомянутых позиций все же занята остатком аргинина.
Сам по себе участок "RR" встречается в выравнивании относительно часто, однако он находится в непосредственной близости от еще одной группы консервативных остатков. Таким образом, для найденного мотива можно записать следующий паттерн:
RR.{5}HLW
Поиск по нему в Jalview дает только 23 находки в последовательностях выделенной клады.
Задание 3
Для выполнения этого задания была выбрана запись с AC P19954 в базе данных SwissProt, принаддлежащая рибосома-связывающему белку PSRP1 хлоропластов шпината (Ribosome-binding factor PSRP1, chloroplastic; Spinacia oleracea).
Для последовательности выбраного белка был запущен поиск Psi-blast через сайт NCBI. При этом был использован порог по E-value 0.005 для итераций Psi-blast и 1 для отображения находок (при значении по умолчанию 0.05 не было ни одной "неправильной" находки). Поиск проводился в базе данных SwissProt до стабилизации списка "правильных" (прошедших порог 0.005) находок. Результаты каждой итерации приведены в таблице ниже (Табл. 1).
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 17 | P30334.1 | 0.004 | P0AD49.2 | 0.064 |
| 2 | 28 | P9WMA8.1 | 0.003 | Q0C0T0.1 | 0.027 |
| 3 | 28 | P9WMA8.1 | 7e-13 | A5DDJ4.2 | 0.018 |
Уже начиная с третьей итерации, число находок с E-value меньше 0.005 прекратило изменяться. При этом худшая "правильная" находка на 11 порядков отличалась по E-value от лучшей неправильной, что является свидетельством высокой обособленности полученной группы. Таким образом, данное семейство является хорошо обоснованным с точки зрения сходства последовательностей входящих в него белков.
Задание 4
В данном задании было оценено соотношение ожидаемого и наблюдаемого количества сайтов "TA" в геноме бактерии Chlorobium limicola штамма DSM 245 (RefSeq AC: GCF_000020465.1).
Для подсчета реального числа искомых участков была использована программа wordcount пакета EMBOSS:
wordcount c_limicola_genomic -wordsize 2 -filter | grep "^TA"
Их количество составило 101248.
Далее было рассчитано ожидаемое значение с учетом GC-состава (51%) и длины (2763181 п. н.) генома:
μ = 0.2452 × 2763181 = 165859.94
Ожидаемое количество сайтов "TA" оказалось примерно на 64612 (в 1.64 раза) больше наблюдаемого. Для оценки значимости различия был использован Z-тест (см. практикум 6). Значение Z-статистики составило -163.64, что соответствует крайне малому P-value (рассчитать его так и не получилось, так как все использованные для этого методы выдавали 0). Таким образом, можно утверждать, что количество участков "TA" в геноме Chlorobium limicola действительно занижено.
Литература
- Siddavattam, D., Singh, M., & Klingmüller, W. (1993). Structure of the nifQ gene from Enterobacter agglomerans 333 and its overexpression in Escherichia coli. Molecular & general genetics : MGG, 239(3), 435–440. https://doi.org/10.1007/BF00276942