Практикум 7
В данном практикуме я работал с PDB ID 6DLU. Эта запись соответствует структуре человеческого динамина-1 (Dynamin-1) в комплексе с 5'-гуанилилметилендифосфонатом (GMPPCP) — не гидролизуемым аналогом GTP, в котором ангидридная связь между β- и γ-фосфатами заменена на метиленовый мостик. В клетке динамин ассоциирован с мембраной за счет связывания фосфолипидов и способен образовывать олигомерные комплексы вокруг образующихся эндоцитозных везикул. Этот белок является ГТФазой и использует энергию гидролиза GTP для отделения сформировавшейся везикулы.
Структура 6DLU содержит 748 из 864 остатков белка и не включает C-концевой нативно неупорядоченный пролин-богатый домен. В записи PDB дается ссылка на идентификатор Q05193 (ID DYN1_HUMAN) в базе данных UniProtKB (Swiss-Prot), соответствующий полной последовательности динамина-1 человека.
Задание 1
В этом задании мной был реализован алгоритм DOMAK, после чего он был использован для поиска структурных доменов в динамине-1.
Описание алгоритма DOMAK
Алгоритм DOMAK позволяет выделять структурные домены в белке с использованием координат его атомов. Его идея заключается в том, что остатки в пределах домена имеют больше контактов друг с другом, чем с остатками за его пределами. В данном случае, под контактами обычно понимается простая сближенность атомов. В основе алгоритма — вычисление для всех остатков белка функции SplitValue, характеризующей то, насколько хорошо белок разделяется на домены по данной границе:
SplitValue = (intA/extAB) ∙ (intB/extAB)
intA — число пар контактирующих остатков из домена A (до границы), intB — число число пар контактирующих остатков из домена B (после границы), а extAB — число пар контактирующих остатков из разных доменов.
Пик функции SplitValue означает, что данный остаток разделяет участки последовательности, мало контактирующие друг с другом, но имеющие много контактов внутри себя.
Алгоритм позволяет построить иерархию субдоменов: после определения максимума SplitValue можно аналогично разделить на две части каждый из двух получившихся доменов. Этот процесс можно продолжать до достижения заданного минимального значения SplitValue или минимального размера домена. Модификации алгоритма также позволяют искать домены, состоящие из нескольких сегментов последовательности: для этого необходимо перебирать не одну координату точки разделения, а сразу несколько и соответствующим образом считать SplitValue.
Реализация и применение
Для реализации алгоритма был дополнен шаблон в Google Colab: была написана функция SplitValue, и ее значение было посчитано для всех остатков в последовательности. Дополненный шаблон доступен по этой ссылке. После применения алгоритма к структуре 6DLU был построен график, показывающий зависимость значения SplitValue от позиции в последовательности (Рис. 1).
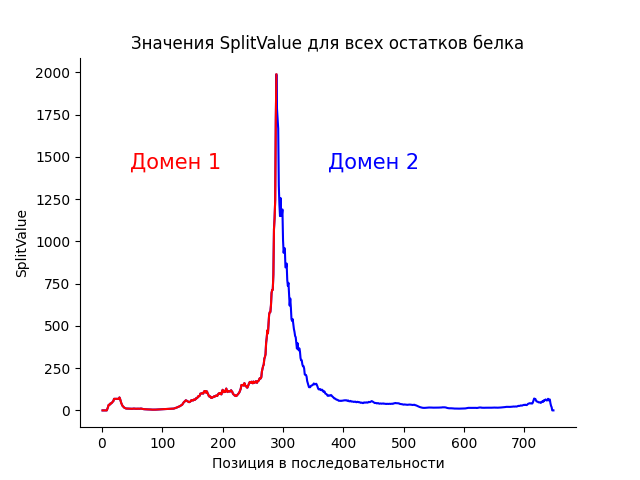
На графике имеется один высокий и узкий пик, соответствующий позиции 288. Таким образом, по результатам работы алгоритма исследуемый белок можно разделить на два домена, слабо взаимодействующих друг с другом. Визуализация такого разделения структуры белка представлена ниже (Рис. 2).
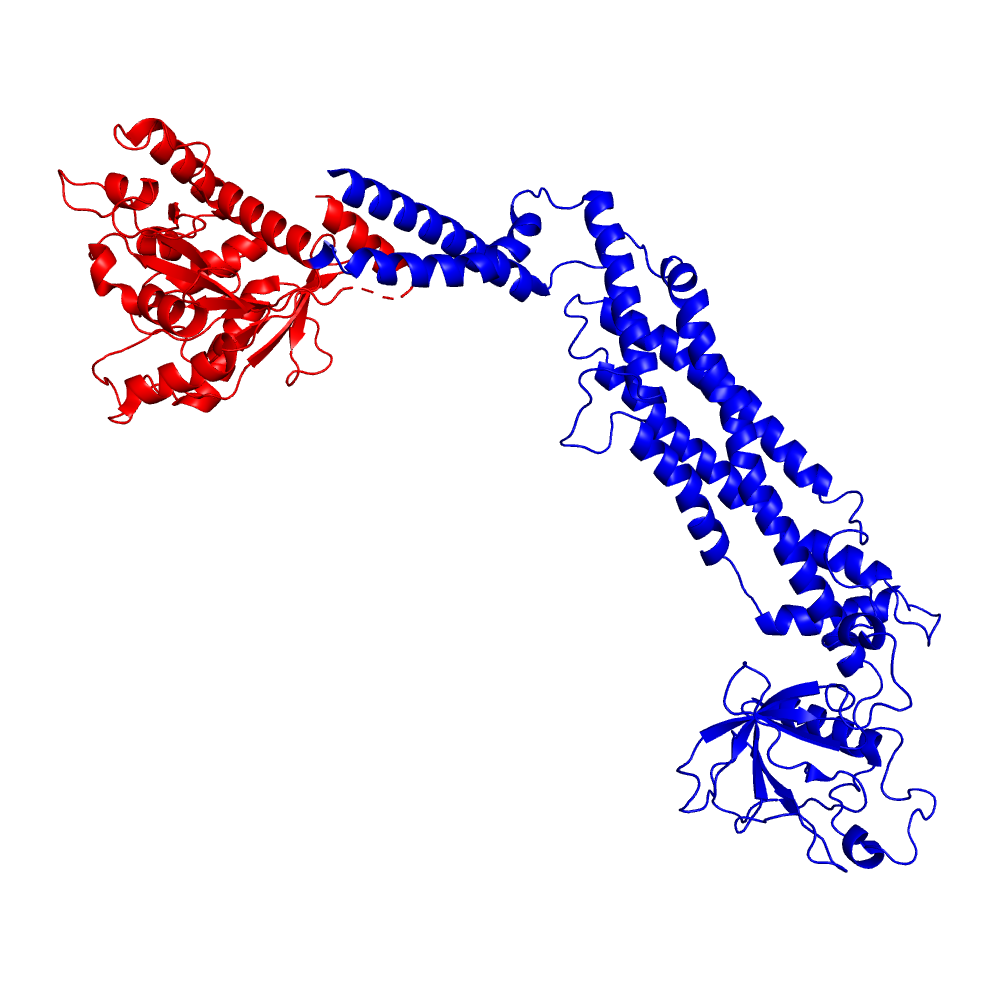
В данном случае оптимальная граница между доменами проходит в конце C-концевой α-спирали домена 1 (показан красным) за несколько остатков до неструктурированной петли. Домен 1 имеет выраженную α/β-структуру с чередованием α-спиралей и β-тяжей, образующих β-лист (трехслойный αβα сэндвич). Второй домен имеет более сложную структуру и явно состоит из нескольких частей, хотя на графике присутствует только один хороший пик. Вероятно, это связано с тем, что крупный фрагмент домена 2, занимающий в структуре белка центральное положение, состоит из двух непрерывных сегментов последовательности. Данная реализация алгоритма не способна выделять такие "составные" домены, так как для них невозможно выделить один остаток, отделяющий их от остальной части белка.
Задание 2
Информацию о структурной классификации белков и об их разбиении на структурные домены можно также найти в базах данных SCOP и CATH. В данном случае, в обеих базах данных отсутствуют записи для модели 6DLU, однако они имеются для других структур, ссылающихся на ту же последовательность в Uniprot (AC Q05193). Для сравнения будут приведены домены, выделенные в модели 3SNH. Соответствующая ей последовательность имеет длину 743 и, по сравнению с 6DLU, не содержит первых трех и последних двух остатков.
SCOP
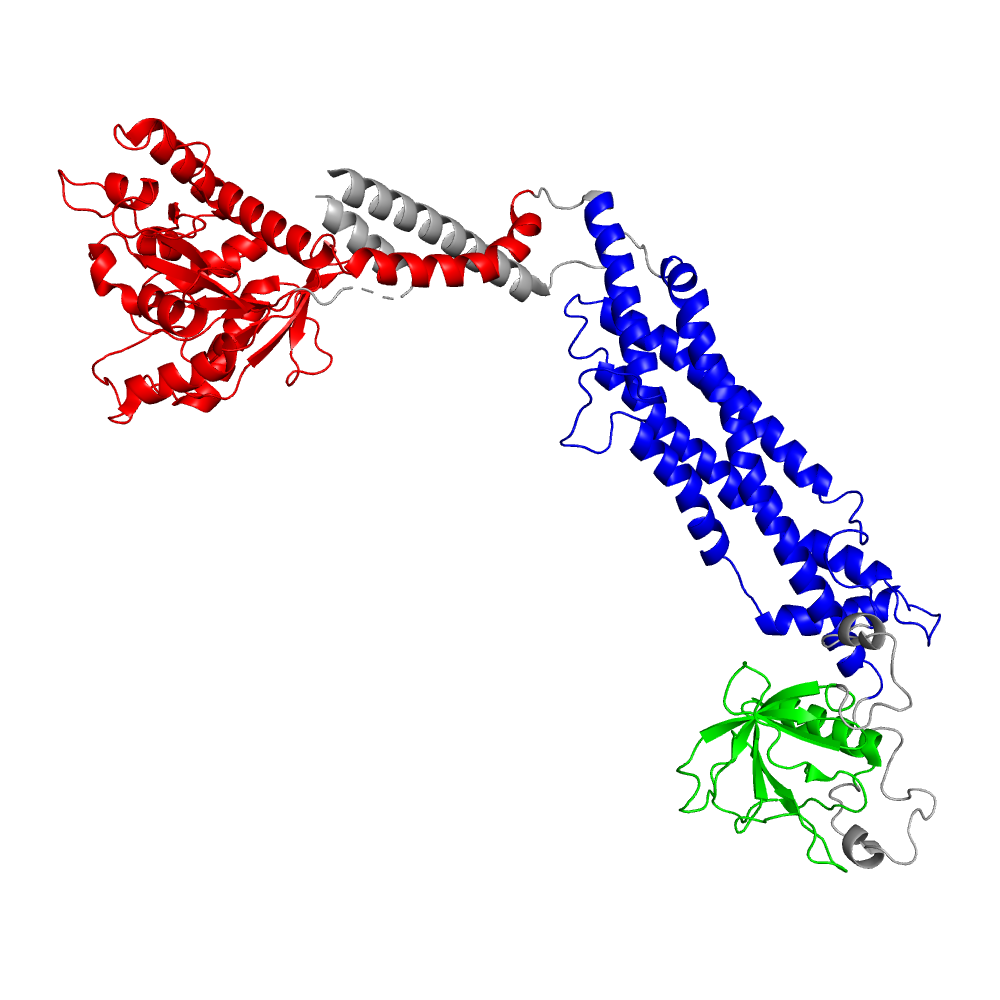
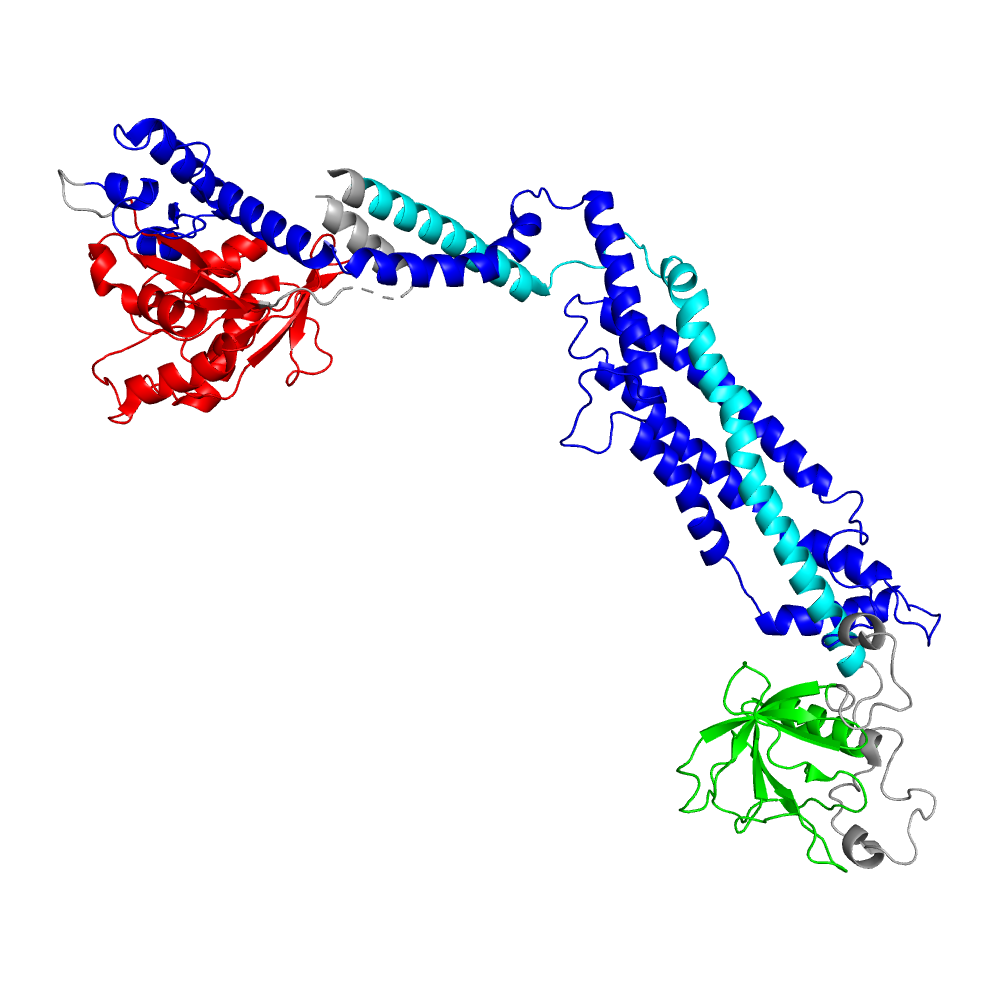
Для динамина-1 в базе данных SCOP содержится три структурных домена (уровня "family"), информация о которых представлена в таблице ниже (Табл. 1). Можно заметить, что все выделенные домены принадлежат разным классам (по составу элементов вторичной структуры). Домен "Dynamin G domain-like" примерно соответствует домену 1 из выдачи DOMAK, однако дополнительно включает первую α-спираль домена 2. Оставшиеся два домена являются частями домена 2 (Рис. 3). Как было замечено ранее, один из них состоит из двух участков последовательности, поэтому они не были разделены алгоритмом.
Ссылка на домены в базе данных SCOP
| Координаты | SCOP ID | Название | Класс |
|---|---|---|---|
| 32-316 | 8058464 | Dynamin G domain-like | α/β |
| 323-496+653-708 | 8058468 | Dynamin stalk region-like | α |
| 518-625 | 8058466 | Pleckstrin-homology domain (PH domain) | β |
CATH
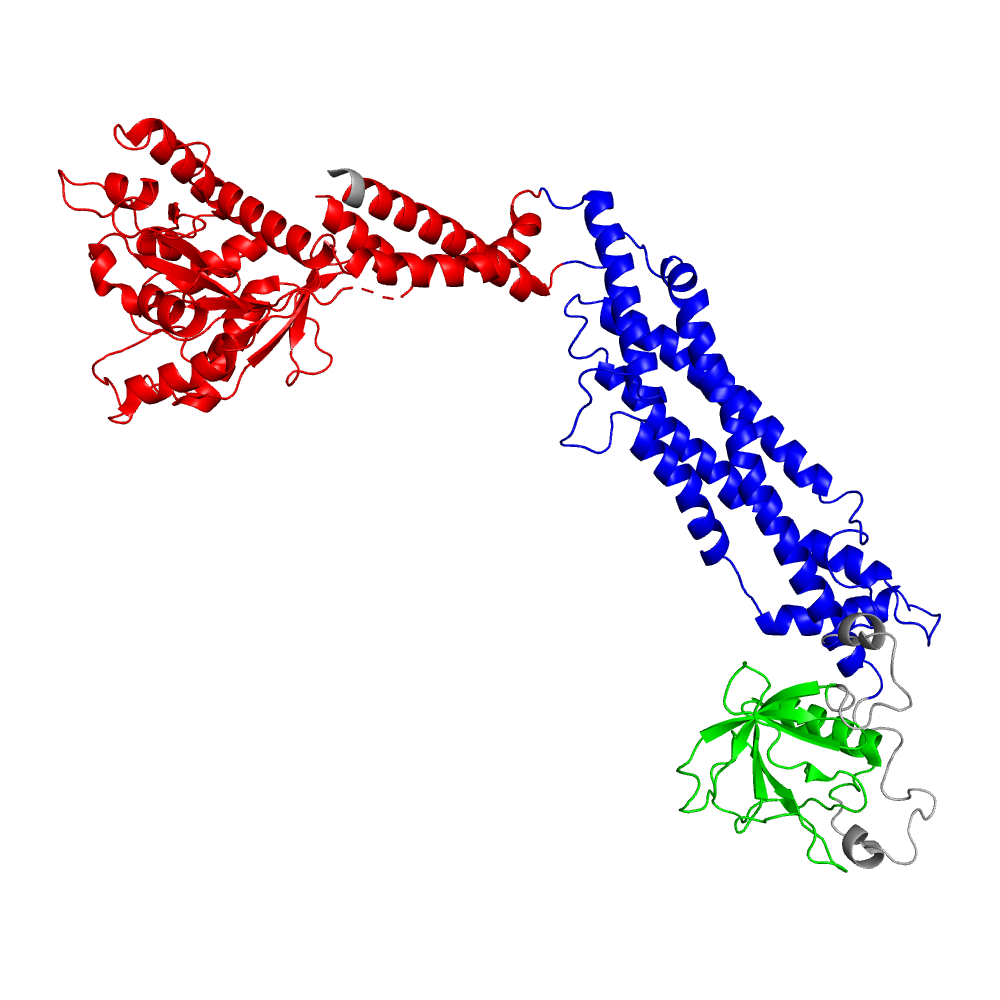
Домены, представленные в базе данных CATH, в целом, сходны с таковыми из SCOP, несмотря на несколько отличающиеся координаты (Табл. 2, Рис. 4). В CATH динамин-1 также подразделяется на N-концевой α/β-домен, α-спиральный домен из двух участков последовательности и вставленный между этими участками домен с преимущественно β-листовой структурой. Важное отличие заключается в том, что в данном случае в состав первого здесь также включается α-спираль, занимающая в укороченном белке C-концевое положение. Таким образом, в отличие от домена 1 из выдачи DOMAK, этот домен включает также "перемычку" из двух α-спиралей, которую алгоритм определил в состав домена 2.
Ссылка на домены в базе данных CATH
| Координаты | Суперсемейство | Топология | Архитектура | Класс |
|---|---|---|---|---|
| 6-317+ 717-745 |
P-loop containing nucleotide triphosphate hydrolases | Rossmann fold | 3-Layer(aba) Sandwich | Alpha Beta |
| 318-499+ 653-716 |
Dynamin, middle domain | Four Helix Bundle | Up-down Bundle | Mainly Alpha |
| 520-628 | Pleckstrin-homology domain (PH domain) | PH-domain like | Roll | Mainly Beta |
Задание 3
InterPro
В полной последовательности динамина-1 был произведен поиск эволюционных доменов с помощью веб-сервиса InterProScan на сайте базы данных InterPro. Этот инструмент использует для поиска доменов различные модели, взятые из разных баз данных, входящих в консорциум InterPro. По этой причине результат поиска может содержать много находок для одних и тех же доменов (Рис. 5).
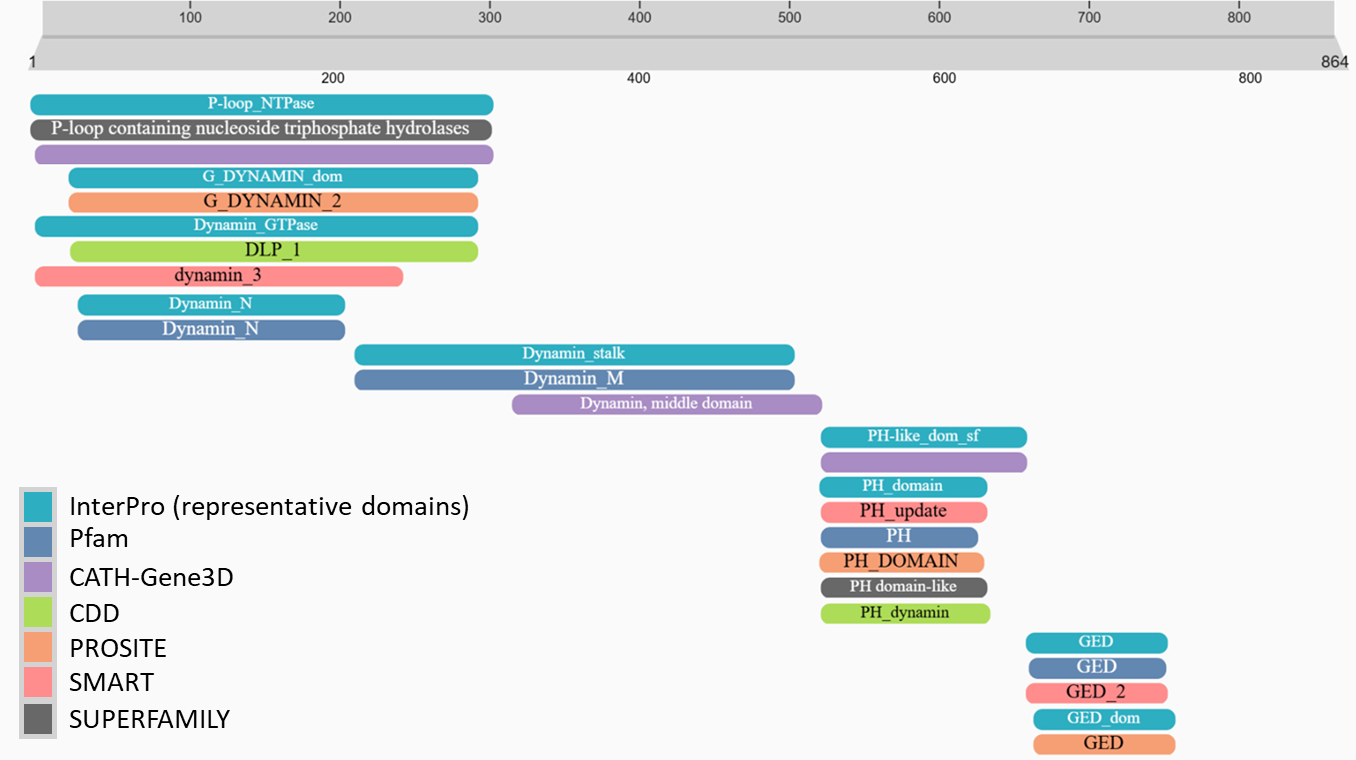
В данном случае, наибольшее количество непересекающихся доменов было найдено по HMM-профилям базы данных Pfam. Этот набор доменов также является наиболее полным, так как покрывает почти всю последовательность за исключением C-концевого нативно неупорядоченного участка.
Информация о выделенных по профилям Pfam в последовательности динамина-1 эволюционных доменов, а также визуализация их положения в структуре белка представлены ниже (Табл. 3, Рис. 5).
| Координаты | ID профиля | Название (короткое имя) |
Число белков |
|---|---|---|---|
| 34-207 | PF00350 | Dynamin family (Dynamin_N) |
81k |
| 215-501 | PF01031 | Dynamin central region (Dynamin_M) |
38k |
| 520-621 | PF00169 | PH domain (PH) |
299k |
| 656-744 | PF02212 | Dynamin GTPase effector domain (GED) |
28k |
Согласно Pfam последовательность динамина-1 включает 4 эволюционных домена. С N-конца расположен большой ГТФазный домен, обеспечивающий связывание и гидролиз GTP. Согласно Pfam, чаще всего он является в белках единственным (26k белков), а также достаточно часто встречается вместе с доменами, не имеющими отношения к динамину. Судя по всему, об этих белках известно достаточно мало, но можно предположить, что они также являются ГТФазами. Следующий за Dynamin_N домен Dynamin_M встречается в динаминах и динамин-подобных белках и отвечает за их димеризацию. После него идет PH-домен. По сравнению с остальными тремя доменами, он содержится в наибольшем количестве белков и доменных архитектур. Помимо динаминов этот домен встречается в протеинкиназах, регуляторах малых G-белков, ассоциированных с цитоскелетом белках, фосфолипазах C млекопитающих и т. д. Чаще всего этот домен участвует в обеспечении белок-белковых взаимодействий или в определении клеточной локализации. В случае динамина-1 он обеспечивает связывание мембранных фосфолипидов. Наконец, C-концевой ГТФазный эффекторный домен (GED), встречающийся в динаминах и родственных им белках, активирует ГТФазную активность домена Dynamin_N в олигомерных комплексах.
В сравнении с разделениями на домены в SCOP и CATH, в Pfam центральный участок, соответствующий α-спиральному структурному домену, дополнительно разбит на Dynamin_M и GED. Это вполне ожидаемо, так как в последовательности эти два участка чаще всего сильно разнесены. Dynamin_M дополнительно содержит концевой участок структурного α/β-домена, а GED — C-концевую α-спираль белка. Домены, состоящие из одного сегмента полипептидной цепи (Dynamin_N и PH) имеют аналоги в SCOP и CATH, а занимающий краевое положение Dynamin_N был также выделен алгоритмом DOMAK (вместе с небольшим участком Dynamin_M).