import __main__
__main__.pymol_argv = [ 'pymol', '-X', '1920', '-Y', '1080']
import pymol
pymol.finish_launching()
from pymol import cmd,stored
cmd.reinitialize()
cmd.do('''
bg_color white
fetch 1lmp, async=0
load 1lmp_mutated.pdb
as lines, n. C+O+N+CA
zoom i. 4+5
#color 1lmp, blue
#color 1lmp_mutated, green
set matrix_mode, 1
set matrix_mode, 1
set movie_panel, 1
set scene_buttons, 1
set cache_frames, 1
set scene_animation_duration, 0
translate [100,0,0], 1lmp_mutated
center all
zoom all
mset 1 x500
mview store
mview store, object=1lmp_mutated
''')
Сохранить полученнное изображение как сцену F0 и ассоциировать с кадром 1.
cmd.do('''
frame 1
show sticks, resi 59
color yellow, organic
scene F0, store
mview store
mview store, object=1lmp
mview store, object=1lmp_mutated
''')
Создать опорный кадр 180: структуры совмещены, установлен вид на остаток 59. На кадрах 440 и 240 показано то же изображение.
cmd.frame(180)
cmd.super('1lmp', '1lmp_mutated')
cmd.show('sticks', 'resi 59')
cmd.color('yellow','organic')
cmd.set_view ('''\
-0.050896551, 0.744614005, 0.665547132,\
-0.993800282, -0.103676051, 0.039991681,\
0.098777518, -0.659392357, 0.745277345,\
0.000723269, -0.000350542, -73.372497559,\
114.090438843, 49.838829041, 25.728450775,\
57.734901428, 88.724609375, -19.999996185''')
cmd.scene('F180', 'store')
cmd.mview('store')
cmd.mview('store', object='1lmp_mutated')
cmd.mview('store', object='1lmp')
Создать опорный кадр 120: структуры совмещены. Таким образом, на кадрах 120-180 меняется только положение камеры, а структуры больше не движутся друг относительно друга.
cmd.frame(120)
cmd.super('1lmp', '1lmp_mutated')
cmd.show('sticks', 'resi 59')
#cmd.zoom('1lmp')
cmd.mview('store')
cmd.mview('store', object='1lmp_mutated')
cmd.mview('store', object='1lmp')
cmd.frame(440)
cmd.super('1lmp', '1lmp_mutated')
cmd.scene('F180', animate=0)
cmd.mview('store')
cmd.mview('store', object='1lmp_mutated')
cmd.mview('store', object='1lmp')
cmd.frame(240)
cmd.super('1lmp', '1lmp_mutated')
cmd.scene('F180', animate=0)
cmd.mview('store')
cmd.mview('store', object='1lmp_mutated')
cmd.mview('store', object='1lmp')
Создать две сцены F1 и F2, на которых показан остаток из моделей 1lmp и 1lmp_mutated соответственно.
cmd.hide('sticks', '1lmp and resi 59')
cmd.show('sticks', '1lmp_mutated and resi 59')
cmd.scene('F1', 'store')
cmd.hide('sticks', '1lmp_mutated and resi 59')
cmd.show('sticks', '1lmp and resi 59')
cmd.scene('F2', 'store')
По очереди показывать (по 5 раз) полученные сцены.
for i in range(10):
cmd.frame(255 + 15*i)
if i%2 == 0:
cmd.scene('F1', animate=0)
elif i%2 == 1:
cmd.scene('F2', animate=0)
cmd.mview('store')
cmd.mview('interpolate')
Полученный фильм:
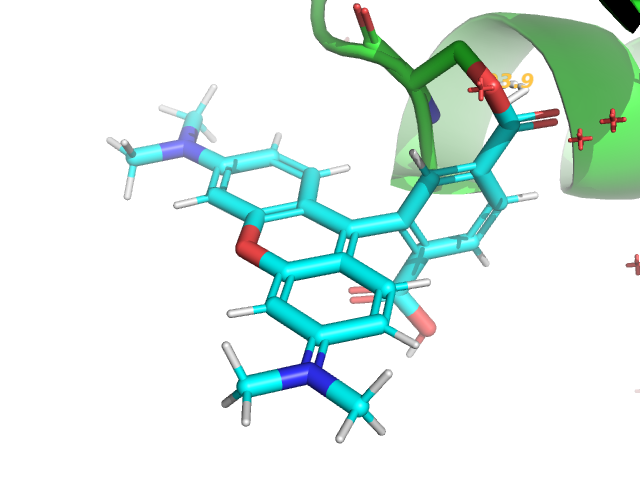
Связывание белка из модели с флюоресцентной меткой TAMRA¶
Сложноэфирная связь между меткой (импортирована в формате sdf) и остатком Ser15 белка добавлена с помощью команды fuse. В импортированной структуре метки нет названий атомов, поэтому атомы (удаляемый кислород и углерод, с которым образуется связь) заданы как ID.
cmd.reinitialize()
cmd.bg_color('white')
cmd.fetch('1lmp')
cmd.load('TAMRA.sdf')
cmd.show('sticks', 'resi 15')
cmd.remove('TAMRA and idx. 4')
cmd.fuse('TAMRA and idx. 31', '/1lmp/A/A/SER`15/OG')
cmd.set_view ('''\
0.957417309, 0.072724372, 0.279395461,\
0.045899000, 0.917102814, -0.395998716,\
-0.285033405, 0.391960502, 0.874713242,\
0.000000000, 0.000000000, -35.216384888,\
2.882838964, 37.299507141, 15.749759674,\
27.764865875, 42.667903900, -20.000000000''')
Построение цистеиновой альфа-спирали¶
Для построения альфа-спирали построена цепь из 100 остатков цистеина (первый остаток добавлен с помощью команды fragment, удлинение цепи -- с помощью команды attach_amino_acid), затем установлены значения двугранных углов (phi=-64, psi=-41, ссылка на источник значений)
cmd.reinitialize()
cmd.bg_color('white')
cmd.fragment('cys')
cmd.set_name('cys', 'helix')
phi = -64
psi = -41
cmd.edit('idx. 3')
for i in range(100):
cmd.editor.attach_amino_acid('pk1', 'cys')
for i in range(2, 100):
cmd.set_dihedral('resi {0} and name N'.format(i),
'resi {0} and name CA'.format(i),
'resi {0} and name C'.format(i),
'resi {0} and name N'.format(i + 1), phi)
cmd.set_dihedral('resi {0} and name C'.format(i),
'resi {0} and name N'.format(i + 1),
'resi {0} and name CA'.format(i + 1),
'resi {0} and name C'.format(i + 1), psi)
cmd.set_view ('''\
1.000000000, 0.000000000, 0.000000000,\
0.000000000, 1.000000000, 0.000000000,\
0.000000000, 0.000000000, 1.000000000,\
0.000000000, 0.000000000, -93.136375427,\
53.605304718, 31.972217560, -46.689609528,\
38.984195709, 149.368209839, -20.000000000''' )
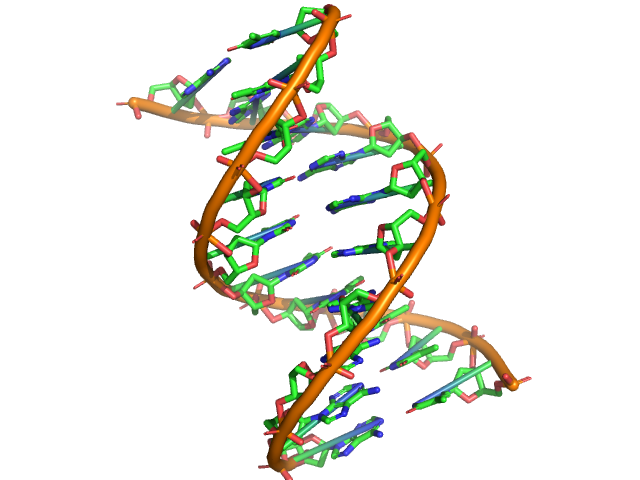
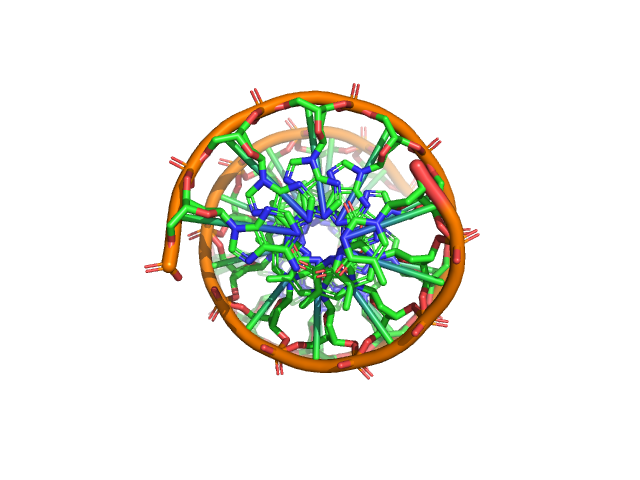
Построение B-формы ДНК¶
Матрица сопоставления соседних нуклеотидов в B-форме получена из структуры 1zf0. Из этой же структуры взята пара нуклеотидов из двух цепей спирали (T4 и A17) и размножена со сдвигом каждой следующей копии, заданным матрицей сопоставления. Полученные нуклеотиды объединены в одну модель (B-helix), между ними созданы связи (команда bond).
cmd.reinitialize()
cmd.fetch('1zf0')
length = 10
cmd.do('''
create pair1, chain A and resi 4 or chain B and resi 17
create pair2, chain A and resi 5 or chain B and resi 16
create 1, pair1
pair_fit pair1, pair2
delete 1zf0
show sticks, 1
alter 1 and chain A, resi=1
alter 1 and chain B, resi={0}
'''.format(length * 2))
transmat = cmd.get_object_matrix("pair1")
cmd.delete('pair1')
cmd.delete('pair2')
for i in range(2, length+1):
cmd.create(str(i), str(i-1))
cmd.alter('{0} and chain A'.format(i), 'resi={0}'.format(i))
cmd.alter('{0} and chain B'.format(i), 'resi={1}'.format(i, length * 2 -i + 1))
cmd.transform_selection(str(i), transmat)
# cmd.fuse("/{0}/A/A/DT`{0}/O3'".format(i - 1),
# "/{0}/A/A/DT`{0}/P".format(i), move=0, mode=0)
cmd.create('B-helix', 'all')
cmd.delete('not model B-helix')
for i in range(2, length + 1):
cmd.bond("/B-helix/A/A/DT`{0}/O3'".format(i - 1),
"/B-helix/A/A/DT`{0}/P".format(i))
for i in range(length + 2, 2*length + 1):
cmd.bond("/B-helix/B/B/DA`{0}/O3'".format(i - 1),
"/B-helix/B/B/DA`{0}/P".format(i))
set_view ('''\
1.000000000, 0.000000000, 0.000000000,\
0.000000000, 1.000000000, 0.000000000,\
0.000000000, 0.000000000, 1.000000000,\
0.000000000, 0.000000000, -102.297409058,\
7.282642365, 31.816791534, 23.586751938,\
80.652053833, 123.942764282, -20.000000000''' )
set_view (\
1.000000000, 0.000000000, 0.000000000,\
0.000000000, 1.000000000, 0.000000000,\
0.000000000, 0.000000000, 1.000000000,\
0.000000000, 0.000000000, -102.297409058,\
7.282642365, 31.816791534, 23.586751938,\
80.652053833, 123.942764282, -20.000000000 )
set_view (\
0.743319869, 0.524730325, -0.414889574,\
0.313025594, 0.275279105, 0.908974886,\
0.591177404, -0.805530608, 0.040365767,\
0.000000000, 0.000000000, -102.297409058,\
7.282642365, 31.816791534, 23.586751938,\
80.652053833, 123.942764282, -20.000000000 )