Предсказание генома производилось для плазмиды CP003562, выделенной из бактерий рода Flammeovirga. Для этих бактерий показана способность к разложению полисахаридов водорослей, в связи с интересом к предназначенной для этого ферментативной системе было выполнено секвенирование [1]. Длина плазмиды — 82913 п.н.; на ней расположено 89 генов, из них 74 — белок-кодирующие.
При выполнении данного задания было произведено сравнение набора генов, предсказанного программой Prodigal для данной плазмиды, с аннотацией генов данной плазмиды в GenBank. Для получения последовательностей и запуска Prodigal были использованы следующие команды:
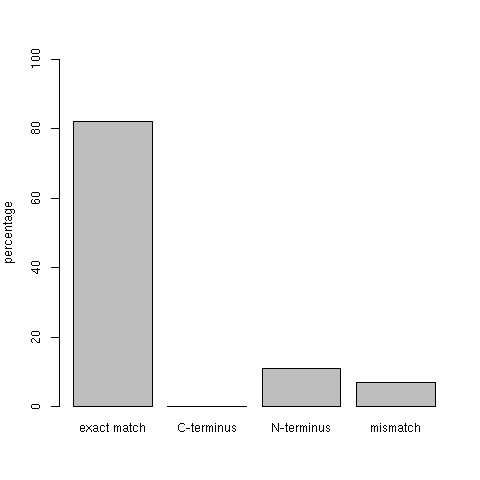
Для сравнения gff файлов был использован скрипт на языке R: script.R. Функция readGFF из пакета rtracklayer считывает особенности из gff-файла в дата-фрейм; затем из полученных дата-фреймов извлекаются только столбцы, содержащие начало, конец и направление особенности, и только строки, соответствующие особенностям типа CDS. Далее к дата-фреймам была применена функция merge (по столбцам start и strand): для особенностей из GenBank (для тех, для которых было возможно) был найден конец особенности из Prodigal, начало и направление которой было таким же, как у этой последовательности из GenBank. Если этот конец совпадает с концом исходной последовательности, то они совпадают; если нет, то они совпадают только по началу (аналогично были найдены последовательности, совпадающие только по концу). Найдем последовательности, совпадающие только по C-концу: это или последовательности на прямой цепи, у которых совпадает только начало, или последовательности на обратной цепи, у которых совпадает только конец (аналогично получим последовательности, совпадающие только по N-концу). Получилось, что точное совпадение с предсказанием Prodigal достигается для 61 из 70 аннотированных генов, по C-концу — 0, по N-концу — 8, нет совпадения для 5 генов (82, 0, 11 и 7 процентов от всех аннотированных генов соответственно). По полученным данным была построена столбчатая диаграмма 1.
Рассмотрим отдельно несколько случаев несовпадения аннотации Prodigal с аннотацией GenBank.