Для выполнения задания были проанализированы хроматограммы, полученные в результате работы капиллярного секвенатора. Далее представлены ссылки на файлы, содержащие исходные данные о хроматограммах:
Характеризуем приведенную хроматограмму. Перед анализом хроматограмм были удалены нечитаемые концевые участки: 1..17 и 710..714 для прямой и 1..37 и 715..723 для обратной. Качество данной хроматограммы представляется мне отличным: соотношение сигнал/шум около 10, пики отличаются друг от друга по высоте примерно в 2-3 раза, разброс уровня шума такой же.
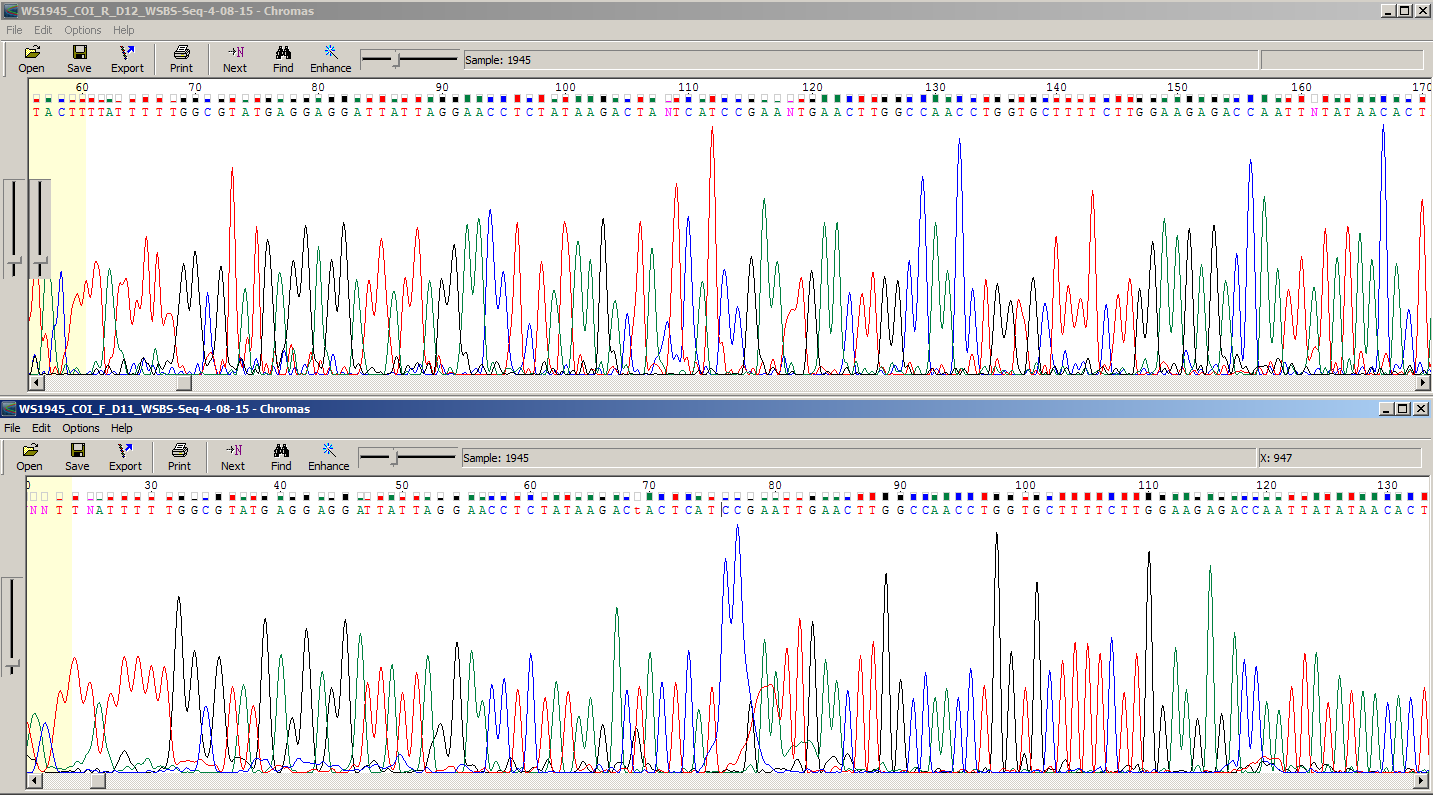
На рисунке 1 изображен участок хроматограмм (прямая снизу, обратная сверху) на котором построение последовательности кажется мне неоднозначным.
Отметим сомнительные участки:
На диаграмме 1 представлено выравнивание полученных последовательностей: прямой и обратной, переведенной в прямую:
Ссылка на файл с проектом JalView: project.jvp
Ссылка на выравнивание полученных последовательностей в формате FASTA: align.fasta
В приведенном ниже примере крупным пикам в большинстве случаев сопутствуют другие, более низкие по уровню, но правильные по форме. Присутствие сравнимых по размеру пиков может быть объяснено либо полиморфизмом, либо присутствием в образце нескольких различных молекул ДНК; поскольку на данном участке дополнительные пики присутствуют в большинстве позиций, предполагается, что в образце есть отличные от секвенируемой молекулы ДНК.