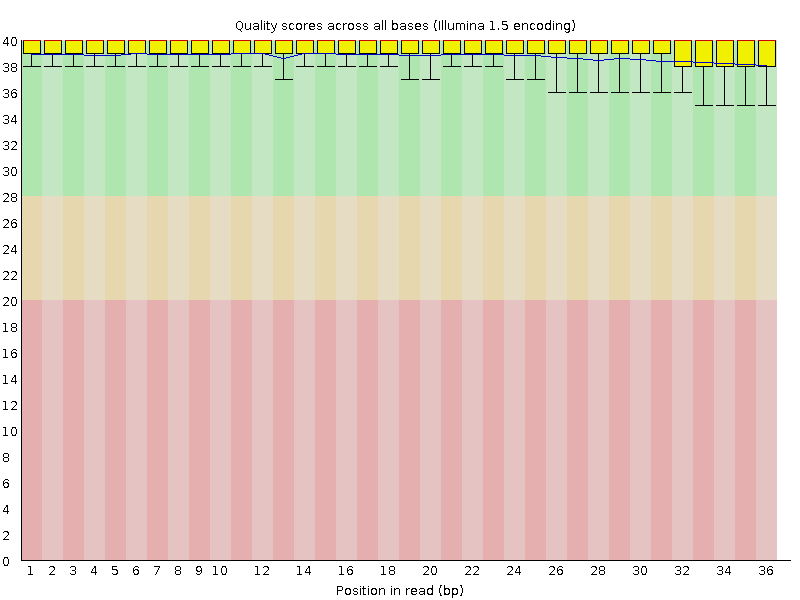
Проведён контроль чтений программой fastqc:
Качество чтений хорошее, нет необходимости фильтровать данные. Для картирования чтений и описания картирования были выполнены следующие команды (команда bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk25.fastq > chipseq_chunk25.sam)
Все 7052 чтения картированы, из них 6546 — на хромосому 14, вероятно, с неё и были получены чтения. Поиск пиков (команда macs2 callpeak --nomodel -n chip25 -t chipseq_chunk25.sorted.bam) возвращает 5 пиков на хромосоме 14. Все пики достаточно достоверны (-log10(p-value) > 16). Ширина равна для них 370, 319, 267, 200, 269. Пики 1 и 5 пересекаются с интронами генов (RAD51B и DCAF5 соответственно). Найденные пики 3 и 4 соответствуют пикам ацетилирования гистона H3K27Ac (часто связяно с регуляцией). Пики показаны в других экспериментах ChIP-seq (пик 1 — в клетках K562 (миелогенная лейкемия), пики 2, 3, 4, 5 — в клетках HUVEC (сосудистый эндотелий человека)). )