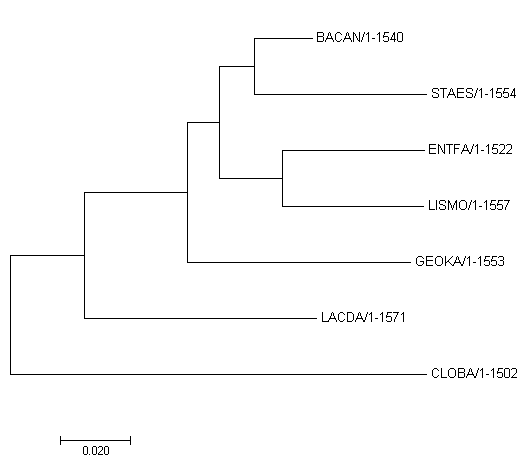
Последовательности 16s рРНК организмов были получены с сайта NCBI (файлы *.frn на ftp). Выравнивание построено в MEGA с помошью MUSCLE, дерево построено методом Maximum Likelihood.
У дерева одна ветвь ({BACAN, GEOKA, LISMO, STAES} против {LACDA, CLOBA}) реконструирована верно, остальные две — нет; качество реконструкции филогении низкое.
Для поиска гомологов был запущен локальный blastp с порогом e-value 0.001, с помощью MView выдача переведена в формат msf, построено выравнивание гомологов в MUSCLE, по нему построено дерево в MEGA методом Maximum Likelihood.
Найдены паралоги (CLPX STAES и HSLU STAES, CLPX BACAN и Q81VX5 BACAN) и группы ортологичных белков (белок CLPX BACSU и прочие формы белка CLPX; белок HSLU STAES и HSLU LISMO). Дупликация гена происходила, например, у LACDA (Q1G869, Q1GBM8) и у общего предка всех этих бактерий (клада, соответствующая CLPX и самая верхняя клада дерева, включающая все организмы). Разделение белков в результате видообразования произошло, например, дважды при отделении общего предка LACDA и ENTFA (белок HSLU, белки Q1GBN8 и Q839B1) и