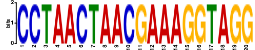
Поиск мотива связывания выполнен для белка IcaR с помощью программы MEME. Найден один мотив длины 20, в каждой последовательности он встречается по одному разу; E-value — 1.8e-017.
A C G T -865 227 -865 -865 -865 227 -865 -865 -865 -865 -865 177 177 -865 -865 -865 177 -865 -865 -865 -865 227 -865 -865 -865 -865 -865 177 177 -865 -865 -865 177 -865 -865 -865 -865 227 -865 -865 -865 -865 227 -865 177 -865 -865 -865 177 -865 -865 -865 177 -865 -865 -865 -865 -865 227 -865 -865 -865 227 -865 -865 -865 -865 177 177 -865 -865 -865 -865 -865 227 -865 -865 -865 227 -865
Лучший (p-value 1.31e-02) похожий аннотированный сайт связывания из выдачи TOMTOM по базе данных regtransbase — NarP_Gammaproteobacteria. По моему мнению, сходство мотивов мало (совпадают отдельные позиции) и несёт скорее случайный характер. К сожалению, получить PWM из RegTransBase на момент написания отчёта (24.04.2017) невозможно, поскольку при переходе по предложенной ссылке возвращена ошибка 500.
При поиске мотива в upstream regions генома Staphylococcus aureus subsp. aureus N315 с помощью FIMO были найдены следующие позиции:
| sequence_name | start | stop | strand | score | p-value | q-value | matched_sequence |
| NP_375784.1|icaR | 9 | 28 | - | 40.6348 | 3.89E-13 | 3.72E-07 | CCTAACTAACGAAAGGTAGG |
| NP_375785.1|icaA | 136 | 155 | + | 40.6348 | 3.89E-13 | 3.72E-07 | CCTAACTAACGAAAGGTAGG |
| NP_375624.1|SA2300 | 558 | 577 | + | 3.9913 | 8.5E-07 | 0.541 | ACGAACTAACGTAAGGTGGA |
| NP_375784.1|icaR | 9 | 28 | + | -1.66957 | 3.42E-06 | 0.992 | CCTACCTTTCGTTAGTTAGG |
| NP_375785.1|icaA | 136 | 155 | - | -1.66957 | 3.42E-06 | 0.992 | CCTACCTTTCGTTAGTTAGG |
| NP_374361.1|sucC | 86 | 105 | + | -2.86087 | 5.19E-06 | 0.992 | CCTAAGTAACAGGAGGATGG |
| NP_373763.1|araB | 132 | 151 | + | -2.86087 | 5.19E-06 | 0.992 | CGCAACTAGCGATATGGAGG |
| NP_374138.1|SA0875 | 408 | 427 | + | -2.86087 | 5.19E-06 | 0.992 | TCAAACTAACGGAGGGTGGC |
| NP_374139.1|murE | 6 | 25 | - | -2.86087 | 5.19E-06 | 0.992 | TCAAACTAACGGAGGGTGGC |
| NP_373305.1|SA0065 | 970 | 989 | - | -2.86087 | 5.19E-06 | 0.992 | CCATAATAGGGAATGGTAGG |
| NP_373778.1|SA0525 | 13 | 32 | + | -4.05217 | 1.14E-05 | 0.992 | CATGACTGACGAAAGACATG |
| NP_374154.1|SA0887 | 124 | 143 | + | -4.05217 | 1.14E-05 | 0.992 | CCAAAGTGCCAAAAGGTACG |
| NP_373478.1|SA0233 | 166 | 185 | + | -4.05217 | 1.14E-05 | 0.992 | CCTACCTCGCTAAAAATAGG |
| NP_375696.1|SA2370 | 175 | 194 | + | -4.05217 | 1.14E-05 | 0.992 | ACCAACTTCCGAAATGTAGA |
| NP_373479.1|SA0234 | 177 | 196 | - | -4.05217 | 1.14E-05 | 0.992 | CCTACCTCGCTAAAAATAGG |
| NP_374599.1|SA1316 | 4 | 23 | + | -5.24348 | 2.06E-05 | 0.992 | AATAACAATAAAAAGGTAGG |
| NP_374232.1|pycA | 98 | 117 | + | -5.24348 | 2.06E-05 | 0.992 | GTTCAATAACGGAAGTTAGG |
| NP_374795.1|dnaI | 119 | 138 | + | -5.24348 | 2.06E-05 | 0.992 | CTGAATTAACGAATGCTATG |
| NP_373339.1|SA0098 | 129 | 148 | + | -5.24348 | 2.06E-05 | 0.992 | TGTAACTAAGGAGATGAAGG |
| NP_375545.1|SA2222 | 262 | 281 | + | -5.24348 | 2.06E-05 | 0.992 | ACTAACTATAGATATGTACG |
| NP_373637.1|set11 | 282 | 301 | + | -5.24348 | 2.06E-05 | 0.992 | ATTAACTAACTTAACGTTGG |
| NP_374526.1|sucA | 283 | 302 | + | -5.24348 | 2.06E-05 | 0.992 | AATGACTAACGAAAGAAAAG |
| NP_374951.1|SA1662 | 285 | 304 | + | -5.24348 | 2.06E-05 | 0.992 | ATTCAGTAACGGAAGTTAGG |
| NP_374526.1|sucA | 287 | 306 | + | -5.24348 | 2.06E-05 | 0.992 | ACTAACGAAAGAAAAGAAGT |
| NP_375456.1|SAS086 | 15 | 34 | - | -5.24348 | 2.06E-05 | 0.992 | TATGATTAACGAAAGGGAGA |
| NP_375062.1|SA1764 | 68 | 87 | - | -5.24348 | 2.06E-05 | 0.992 | TCTAACTAAGGAAGGGTCAT |
| NP_373394.1|capI | 155 | 174 | - | -5.24348 | 2.06E-05 | 0.992 | CCTAACAAACGATAATTACA |
| NP_374430.1|SA1154 | 426 | 445 | - | -5.24348 | 2.06E-05 | 0.992 | ACTAAAAAACGAATTGTAGC |
| NP_373401.1|capP | 22 | 41 | + | -6.43478 | 2.61E-05 | 0.992 | CATGATTAAGGAAAGATAAG |
| NP_375825.1|SA2497 | 162 | 181 | + | -6.43478 | 2.61E-05 | 0.992 | ATTAATTATAGAAAGGTACG |
| NP_374147.1|SA0883 | 183 | 202 | + | -6.43478 | 2.61E-05 | 0.992 | ATAAATTAAAGAAAGGTAGT |
| NP_375624.1|SA2300 | 298 | 317 | + | -6.43478 | 2.61E-05 | 0.992 | TTTAATTAACAAATGATAGG |
| NP_373871.1|vraF | 2 | 21 | - | -6.43478 | 2.61E-05 | 0.992 | TCTAAGTAACAAAACGCATG |
| NP_375826.1|SAS092 | 7 | 26 | - | -6.43478 | 2.61E-05 | 0.992 | ATTAATTATAGAAAGGTACG |
| NP_374146.1|SA0882 | 8 | 27 | - | -6.43478 | 2.61E-05 | 0.992 | ATAAATTAAAGAAAGGTAGT |
| NP_374632.1|SA1349 | 122 | 141 | - | -6.43478 | 2.61E-05 | 0.992 | CCTAACTAATGATATATATC |
| NP_375625.1|SA2301 | 230 | 249 | - | -6.43478 | 2.61E-05 | 0.992 | TTTAATTAACAAATGATAGG |
| NP_374272.1|SA1001 | 747 | 766 | - | -6.43478 | 2.61E-05 | 0.992 | CATAACTAAAGAAAGCTTTA |
| NP_375050.1|SA1755 | 42 | 61 | + | -7.62609 | 2.71E-05 | 0.992 | ACTAACTAAAAAAAAGTACA |
| NP_373877.1|SA0622 | 409 | 428 | + | -7.62609 | 2.71E-05 | 0.992 | TATAACTAACAAAAGATAAT |
| NP_375049.1|SA1754 | 622 | 641 | - | -7.62609 | 2.71E-05 | 0.992 | ACTAACTAAAAAAAAGTACA |
| SAtRNA60|SAtRNA60 | 38 | 57 | + | -8.52174 | 2.75E-05 | 0.992 | CCTTACAAGCAGAGGGTCGG |
| SAtRNA52|SAtRNA52 | 42 | 61 | + | -8.52174 | 2.75E-05 | 0.992 | CCTTACAAGCAGAGGGTCGG |
| SAtRNA06|SAtRNA06 | 43 | 62 | + | -8.52174 | 2.75E-05 | 0.992 | CCTTACAAGCAGAGGGTCGG |
| SAtRNA61|SAtRNA61 | 111 | 130 | + | -8.52174 | 2.75E-05 | 0.992 | CCTTACAAGCAGAGGGTCGG |
| SAtRNA54|SAtRNA54 | 121 | 140 | + | -8.52174 | 2.75E-05 | 0.992 | CCTGATAAGCGTGAGGTCGG |
| SAtRNA04|SAtRNA04 | 121 | 140 | + | -8.52174 | 2.75E-05 | 0.992 | CCTGATAAGCGTGAGGTCGG |
| SAtRNA14|SAtRNA14 | 124 | 143 | + | -8.52174 | 2.75E-05 | 0.992 | CCTGATAAGCGTGAGGTCGG |
| NP_373683.1|gltD | 184 | 203 | + | -8.52174 | 2.75E-05 | 0.992 | CCGGACAACAGTATGGTAGG |
| SArRNA08|SArRNA08 | 229 | 248 | + | -8.52174 | 2.75E-05 | 0.992 | CCTTACAAGCAGAGGGTCGG |
| SArRNA04|SArRNA04 | 715 | 734 | + | -8.52174 | 2.75E-05 | 0.992 | CCTTACAAGCAGAGGGTCGG |
| NP_374727.1|SA1442 | 21 | 40 | + | -9.71304 | 3.22E-05 | 0.992 | CTAACCTTAAGATGGGTAGG |
| NP_374727.1|SA1442 | 21 | 40 | - | -9.71304 | 3.22E-05 | 0.992 | CCTACCCATCTTAAGGTTAG |
| NP_375290.1|SA1979 | 35 | 54 | - | -9.71304 | 3.22E-05 | 0.992 | CCGTGCAAACGATATGTAGT |
| NP_375576.1|opp-1D | 51 | 70 | - | -9.71304 | 3.22E-05 | 0.992 | CATCACTCACGAGTGGTTGA |
| NP_375088.1|SA1789 | 66 | 85 | - | -9.71304 | 3.22E-05 | 0.992 | CCTGTATTACGAAAGCGTGG |
| NP_373479.1|SA0234 | 177 | 196 | + | -9.71304 | 3.22E-05 | 0.992 | CCTATTTTTAGCGAGGTAGG |
| NP_373636.1|set10 | 165 | 184 | - | -9.71304 | 3.22E-05 | 0.992 | CTTCACACCCGATAGATAGG |
| NP_373478.1|SA0233 | 166 | 185 | - | -9.71304 | 3.22E-05 | 0.992 | CCTATTTTTAGCGAGGTAGG |
| NP_373575.1|SA0329 | 452 | 471 | + | -9.71304 | 3.22E-05 | 0.992 | CCAATCTTAGATAAGGTTGG |
| NP_374839.1|tyrS | 497 | 516 | + | -9.71304 | 3.22E-05 | 0.992 | CCGATCCAACGGCAGATAGT |
| NP_374506.1|lysC | 976 | 995 | + | -9.71304 | 3.22E-05 | 0.992 | CCTTCCTAACAATTAGGAGG |
| NP_375100.1|SA1800 | 7 | 26 | - | -10.9043 | 5.53E-05 | 1 | CATTATACACGAAAGGAAAG |
| NP_373567.1|SA0321 | 9 | 28 | - | -10.9043 | 5.53E-05 | 1 | GACATCTAACGAAATGGTGG |
| NP_375099.1|SA1799 | 29 | 48 | + | -10.9043 | 5.53E-05 | 1 | CATTATACACGAAAGGAAAG |
| NP_374700.1|holA | 32 | 51 | + | -10.9043 | 5.53E-05 | 1 | TCAAACGAATGAAAGGGTGT |
| NP_395567.1|SAP031 | 13 | 32 | - | -10.9043 | 5.53E-05 | 1 | CCAAACTAAAATATGGAAAG |
| NP_375784.1|icaR | 13 | 32 | - | -10.9043 | 5.53E-05 | 1 | ACAACCTAACTAACGAAAGG |
| NP_374405.1|SA1131 | 30 | 49 | - | -10.9043 | 5.53E-05 | 1 | TCATACTAATGGACGATAGG |
| NP_373988.1|secG | 32 | 51 | - | -10.9043 | 5.53E-05 | 1 | CCTTATATACGACAAGTAGT |
| NP_375381.1|moaA | 50 | 69 | - | -10.9043 | 5.53E-05 | 1 | CGTAAGTCACGGATGGGACG |
| NP_374640.1|accC | 52 | 71 | - | -10.9043 | 5.53E-05 | 1 | CCTAAATCACGACAAGCGCG |
| NP_373299.1|SA0059 | 73 | 92 | + | -10.9043 | 5.53E-05 | 1 | CCTCCCAAAGAAAAAGGAGG |
| NP_375395.1|SA2077 | 96 | 115 | + | -10.9043 | 5.53E-05 | 1 | CATGAAATACGAAAGGAAGT |
| NP_373698.1|SA0446 | 110 | 129 | + | -10.9043 | 5.53E-05 | 1 | CCAAATATACGAAAGTAAGA |
| NP_375785.1|icaA | 132 | 151 | + | -10.9043 | 5.53E-05 | 1 | ACAACCTAACTAACGAAAGG |
| NP_373451.1|SA0207 | 139 | 158 | + | -10.9043 | 5.53E-05 | 1 | CCTAACCAACTTACGATGTG |
| NP_374664.1|rpmG | 124 | 143 | - | -10.9043 | 5.53E-05 | 1 | CCGTACAAGCTAAAGTTACG |
| NP_374044.1|SA0783 | 158 | 177 | + | -10.9043 | 5.53E-05 | 1 | CACAAGTAGCGATAGGGACG |
| NP_373376.1|drm | 166 | 185 | + | -10.9043 | 5.53E-05 | 1 | CCAAACCTTGAAAAGTTAGG |
| NP_375137.1|groEL | 169 | 188 | + | -10.9043 | 5.53E-05 | 1 | CCTAAAGGACGTAATGTTGT |
| NP_373568.1|SA0322 | 184 | 203 | + | -10.9043 | 5.53E-05 | 1 | GACATCTAACGAAATGGTGG |
| NP_373376.1|drm | 166 | 185 | - | -10.9043 | 5.53E-05 | 1 | CCTAACTTTTCAAGGTTTGG |
| NP_374151.1|SAS028 | 204 | 223 | + | -10.9043 | 5.53E-05 | 1 | GCTATCTGAGTAAAGGGGGG |
| NP_375118.1|sec3 | 188 | 207 | - | -10.9043 | 5.53E-05 | 1 | GCTAACTTACGAATAGATGA |
| NP_374495.1|opp-2B | 276 | 295 | + | -10.9043 | 5.53E-05 | 1 | AGTAACTTACACAAGGAGGG |
| NP_395541.1|cadD | 281 | 300 | + | -10.9043 | 5.53E-05 | 1 | CCCAGATAAAGTGAGGTAGC |
| NP_375543.1|SA2220 | 295 | 314 | + | -10.9043 | 5.53E-05 | 1 | CCTGACTCATTTAAGGAAAG |
| NP_374819.1|ald | 316 | 335 | + | -10.9043 | 5.53E-05 | 1 | CTGAACAAGCAAAAGTTTGG |
| NP_375393.1|FdhD | 312 | 331 | - | -10.9043 | 5.53E-05 | 1 | CATATCTAACAATTGGTTGA |
| NP_395537.1|rep | 394 | 413 | + | -10.9043 | 5.53E-05 | 1 | CCAAACTAAAATATGGAAAG |
| NP_375315.1|hysA | 442 | 461 | - | -10.9043 | 5.53E-05 | 1 | CATTAATAACGTAATGGTGG |
| NP_374077.1|SA0816 | 470 | 489 | - | -10.9043 | 5.53E-05 | 1 | CTCAACAAACGCACCGTATG |
| NP_375119.1|SA1818 | 580 | 599 | + | -10.9043 | 5.53E-05 | 1 | GCTAACTTACGAATAGATGA |
| NP_375032.1|SA1739 | 579 | 598 | - | -10.9043 | 5.53E-05 | 1 | CATCTATATCGAAACGTGGG |
| NP_374197.1|SA0930 | 614 | 633 | + | -10.9043 | 5.53E-05 | 1 | CCCTAACAACTAAAGGTTGT |
| NP_374928.1|SA1639 | 768 | 787 | - | -10.9043 | 5.53E-05 | 1 | TCTAACTAGGGTGTGATAGG |
Из этих позиций первые две имеют достаточно низкий q-value (меньше 0.05) для того, чтобы считать частоту ошибки в этих случаях малой. Поиск проводился только по upstream regions, так как связывание транскрипционных факторов происходит с участками выше гена, и поиск по всему геному может быть медленным.
Эти два гена — сам транскрипционный фактор IcaR и IcaA (N-гликозил трансфераза, участвующая в формировании полисахарида PgaC).
Для поиска сайтов связывания метилтрансфераз были получены upstream-регионы генов из RSAT (используются в MEME): upstreams.fasta, в них сайты искались программой fuzznuc.
icaR:
Start End Strand Pattern Mismatch Sequence
45 48 + test1:AATT . AATT
60 63 + test1:AATT . AATT
83 86 + test1:AATT . AATT
103 106 + test1:AATT . AATT
114 117 + test1:AATT . AATT
139 142 + test1:AATT . AATT
120 123 + test7:CCDG . CCTG
120 123 + test8:CCDS . CCTG
9 12 + test15:CNNR . CCTA
37 40 + test15:CNNR . CATA
90 93 + test15:CNNR . CGGA
111 114 + test15:CNNR . CTTA
120 123 + test15:CNNR . CCTG
127 130 + test15:CNNR . CATA
34 37 + test24:RGCY . AGCC
144 147 + test25:TCAG . TCAG
64 67 + test28:TGCA . TGCA
106 109 + test28:TGCA . TGCA
58 61 + test29:TTAA . TTAA
85 88 + test29:TTAA . TTAA
112 115 + test29:TTAA . TTAA
137 140 + test29:TTAA . TTAA
9 13 + test56:CCTAC . CCTAC
35 39 + test93:GCCAT . GCCAT
119 123 + test128:YSCNS . TCCTG
94 99 + test133:AATATT . AATATT
8 13 + test140:ACCYAC . ACCTAC
136 141 + test161:ATTAAT . ATTAAT
37 42 + test178:CATATG . CATATG
4 9 + test223:CTNNAC . CTTTAC
42 47 + test312:RGTAAT . GGTAAT
79 84 + test332:TTATAA . TTATAA
155 160 + test332:TTATAA . TTATAA
71 82 + test534:GANNNNNNNTAY . GATTGTTGTTAT
65 76 + test542:GCANNNNNNTGT . GCAATAGATTGT
43 55 + test658:GTAANNNNNNGTA . GTAATTGATAGTA
icaA:
Start End Strand Pattern Mismatch Sequence
22 25 + test1:AATT . AATT
47 50 + test1:AATT . AATT
58 61 + test1:AATT . AATT
78 81 + test1:AATT . AATT
101 104 + test1:AATT . AATT
116 119 + test1:AATT . AATT
13 16 + test15:CNNR . CCTA
17 20 + test15:CNNR . CTGA
29 32 + test15:CNNR . CACA
41 44 + test15:CNNR . CAGG
63 66 + test15:CNNR . CAAA
99 102 + test15:CNNR . CAAA
122 125 + test15:CNNR . CATA
129 132 + test15:CNNR . CTTA
136 139 + test15:CNNR . CCTA
137 140 + test15:CNNR . CTAA
141 144 + test15:CNNR . CTAA
145 148 + test15:CNNR . CGAA
33 36 + test17:CTAT . CTAT
93 96 + test17:CTAT . CTAT
111 114 + test17:CTAT . CTAT
127 130 + test24:RGCY . GGCT
55 58 + test28:TGCA . TGCA
97 100 + test28:TGCA . TGCA
24 27 + test29:TTAA . TTAA
49 52 + test29:TTAA . TTAA
76 79 + test29:TTAA . TTAA
103 106 + test29:TTAA . TTAA
159 163 + test38:AGAAA . AGAAA
13 17 + test56:CCTAC . CCTAC
146 150 + test73:GAAAG . GAAAG
10 14 + test128:YSCNS . CCCCC
65 70 + test133:AATATT . AATATT
23 28 + test161:ATTAAT . ATTAAT
122 127 + test178:CATATG . CATATG
10 15 + test186:CCCCCD . CCCCCT
4 9 + test332:TTATAA . TTATAA
80 85 + test332:TTATAA . TTATAA
111 117 + test373:CTATCAV . CTATCAA
31 41 + test441:CACNNNNNTAC . CACTATGTTAC
146 159 + test717:GAANNNNNNNTAAA . GAAAGGTAGGTAAA
Удалим сайты МТаз, не пересекающиеся с сайтами связывания ТФ
icaR:
Start End Strand Pattern Mismatch Sequence
9 12 + test15:CNNR . CCTA
9 13 + test56:CCTAC . CCTAC
8 13 + test140:ACCYAC . ACCTAC
4 9 + test223:CTNNAC . CTTTAC
icaA:
Start End Strand Pattern Mismatch Sequence
136 139 + test15:CNNR . CCTA
137 140 + test15:CNNR . CTAA
141 144 + test15:CNNR . CTAA
145 148 + test15:CNNR . CGAA
146 150 + test73:GAAAG . GAAAG
146 159 + test717:GAANNNNNNNTAAA . GAAAGGTAGGTAAA
Из особенностей наборов можно отметить "общую" для двух генов МТазу 15 (1 сайт у IcaR и 4 сайта у icaA). В геноме штаммов N315 и MW2 метилтранфераз со специфичностью, описанной во вставке 3, нет.