Нуклеотидный BLAST
Определение функции и таксономии нуклеотидной последовательности
Для определения функции и таксаномии нуклеотидной последовательности был выбран алгоритм blastn, так как это чувствительность этого алгоритма, позволит получить находки в случае неконсерватевности исследуемой последовательности. По этой же причине был выбран наименьший размер слова
Выдача BLAST res1.txt
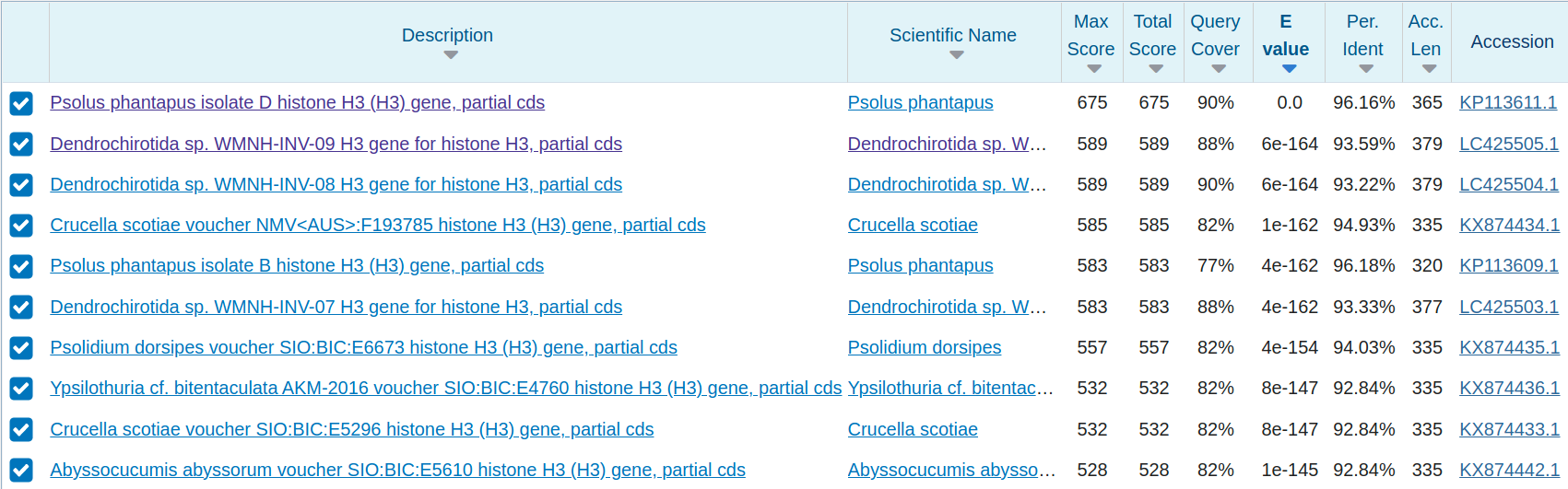
Все найденые последовательности кодирую гистон H3, вероятно и исследуемая последовательность кодирует это же белок. Так же 10 находок с наибольшим скором(рис.1) принадлежат голотуриям, однако для более точной таксаномической идентификации были использованы возможности раздела Distance tree of results (Max Seq Difference = 0.1) (Рис.2). Можно видеть что исследуемая последовательность почти наверняка относится к отряду Dendrochirotida, наиболее близка к роду Psolus и возножно даже принадлижит виду Psolus phantus

Поиск генов белков в неаннотированной нуклеотидной последовательности
Для поиска генов белков был выбран контиг из Wallemia mellicola. Так как по нуклеотдной последоваетльности нужно найти белок, была использована разновидность BLASTx. Чтобы сократить время работы алгоритма был оставлен максимальный размер слова, поиск производился по белкам из базы данных Swiss-Prot, принадлежащих к базидиомицетам, но не к роду Wallemia, чтобы избежать тривиальных находок.
Выдача BLAST доступна по ссылке res2.txtПо результатам можно предположить, что в контиге присутствует ген, кодирующий фактор сплайсинга пре-мРНК CLF1. Об этом говорит то, что выдаче присутствуют три последовательности, которые почти полностью покрываются исследуемым контигом.
Интепретация карты локального сходства гомологичных хромосом двух бактерий
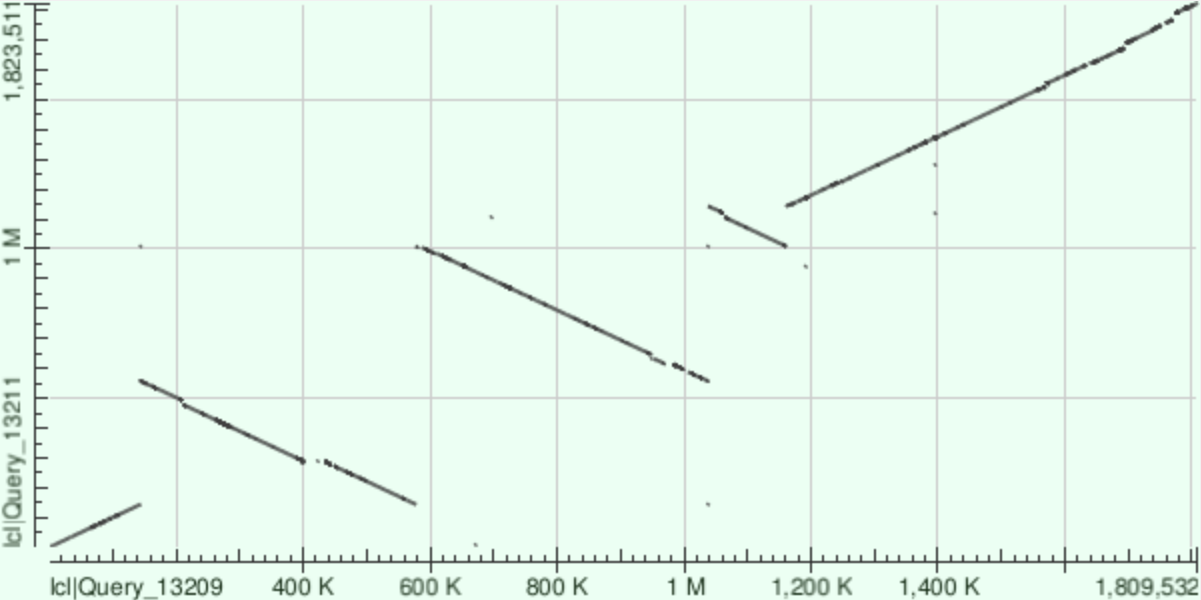
Для сравнения были выбраны 2 бактерии рода Thermatoga: T.pertophila (геном) и T.naphthophila (геном). Для построения карты локального сходства использовался megablast с длинной слова 256(остальные параметры по умолчанию). В результате была получена карта локального сходства, представленная на рис.3. На ней по горизонтали отложен геном T.naphthophila, а по вертикали - T.petrophila. Эта карта демонстрирует 3 инверсии на участках: 150K-580K, 580K-1040K, 1040K-1160K.