Составление списка гомологичных белков, включающих паралоги
| Sequence | Score | e-value |
|---|---|---|
| sp|Q820F8|CLPX_STRAW ATP-dependent Clp protease ATP-binding sub... | 535 | 0 |
| sp|P9WPB9|CLPX_MYCTU ATP-dependent Clp protease ATP-binding sub... | 519 | 0 |
| sp|Q9CBY6|CLPX_MYCLE ATP-dependent Clp protease ATP-binding sub... | 519 | 0 |
| sp|Q8FN57|CLPX_COREF ATP-dependent Clp protease ATP-binding sub... | 518 | 0 |
| sp|Q6NFU7|CLPX_CORDI ATP-dependent Clp protease ATP-binding sub... | 518 | 0 |
| sp|A0JXL2|CLPX_ARTS2 ATP-dependent Clp protease ATP-binding sub... | 516 | 0 |
| sp|B0RAS4|CLPX_CLAMS ATP-dependent Clp protease ATP-binding sub... | 491 | 1E-173 |
| tr|A0K1M3|A0K1M3_ARTS2 ATPase AAA-2 domain protein OS=Arthrobac... | 54.3 | 5E-08 |
| tr|Q8FMH5|Q8FMH5_COREF Putative endopeptidase Clp ATP-binding c... | 47 | 1E-05 |
| tr|Q6NFB1|Q6NFB1_CORDI ATP-dependent Clp protease ATP-binding s... | 45.8 | 3E-05 |
| sp|A0JXB1|RUVB_ARTS2 Holliday junction ATP-dependent DNA helica... | 42.4 | 0.0003 |
| tr|Q82QV8|Q82QV8_STRAW Putative AAA family ATPase OS=Streptomyc... | 41.6 | 0.0003 |
| tr|A0JR82|A0JR82_ARTS2 ATP-dependent zinc metalloprotease FtsH ... | 41.6 | 0.0005 |
| tr|A0K236|A0K236_ARTS2 AAA ATPase, central domain protein OS=Ar... | 41.6 | 0.0005 |
| tr|Q82EE9|Q82EE9_STRAW ATP-dependent zinc metalloprotease FtsH ... | 41.6 | 0.0005 |
| tr|Q82EB8|Q82EB8_STRAW Putative ATP-dependent Clp protease OS=S... | 41.6 | 0.0007 |
| sp|P9WQN3|FTSH_MYCTU ATP-dependent zinc metalloprotease FtsH OS... | 41.2 | 0.0007 |
| tr|B0RHW4|B0RHW4_CLAMS ATP-dependent zinc metalloprotease FtsH ... | 41.2 | 0.0007 |
| tr|Q6NF92|Q6NF92_CORDI ATP-dependent zinc metalloprotease FtsH ... | 40.8 | 0.001 |
Реконструкция и визуализация
Реконструкция филогении была произведена с помощью программы MEGA с алгоритмом Maximal Likehood. Было получено следующее дерево(tree.nwk), визуализация которого представлена ниже.
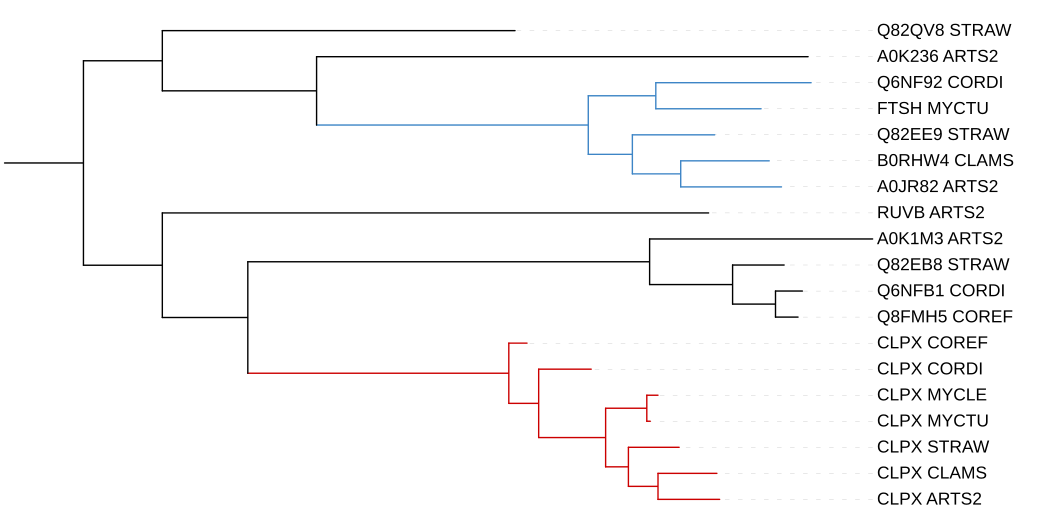
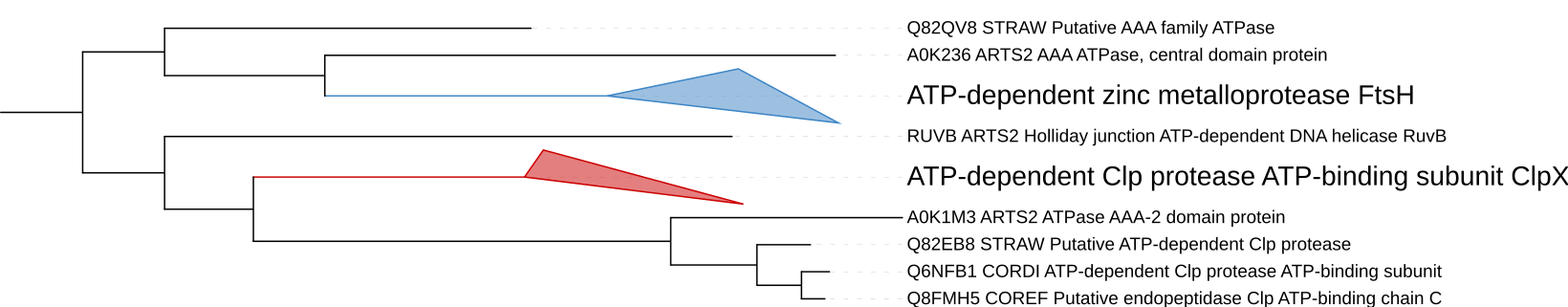
Из найденных белков паралогами являются, к примеру, CLPX_MUCTU и FTSH_MUCTU, A0JR82_ARTS2 и A0K236_ARTS2, CLPX_CLAMS и B0RHW4_CLAMS.
Ортологами являются CLPX_COREF и CLPX_CORDI, A0JR82_ARTS2 и FTSH_MUCTU, Q6NF92_CORDI и B0RHW4_CLAMS.