Трансмембранные белки
1. Знакомство с базой данных OPM
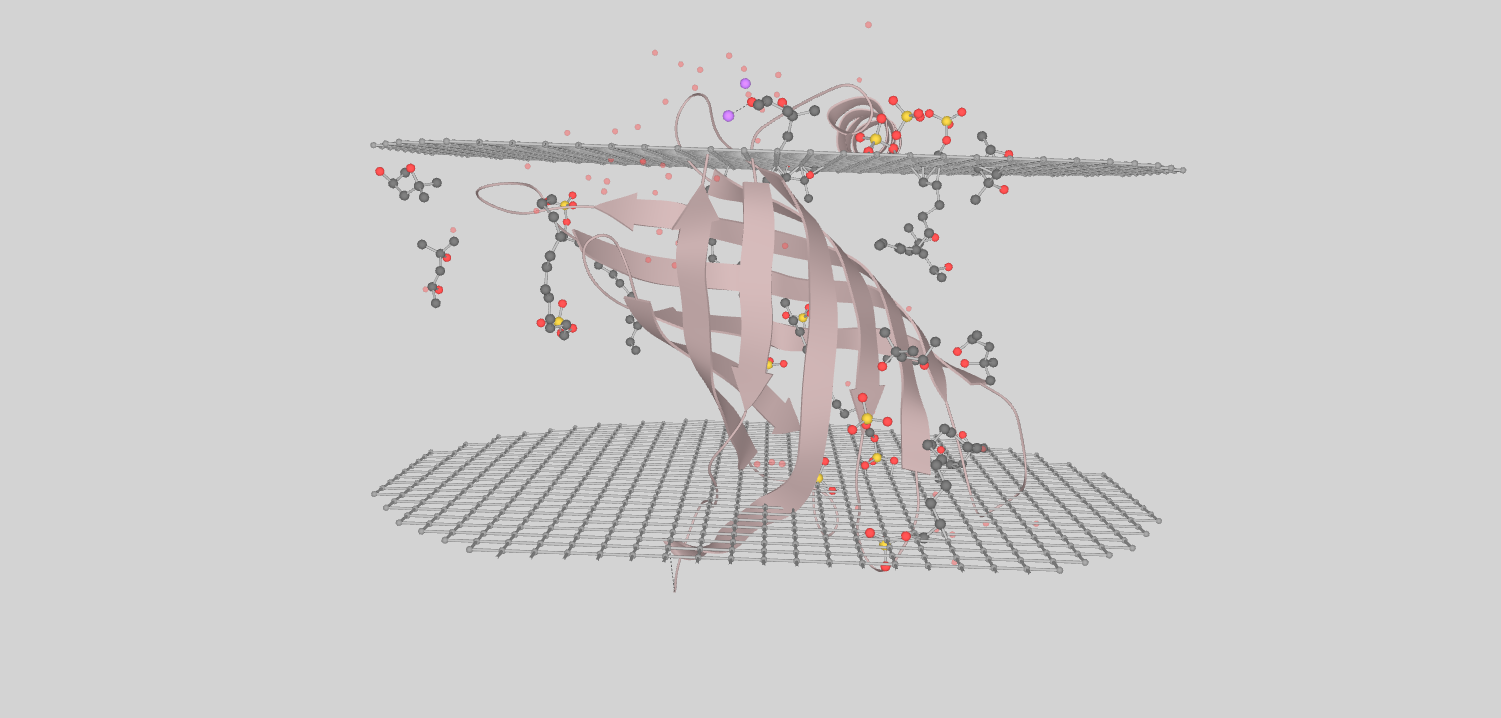
Для выполнения этого задания из базы данных OPM был выбран ацилаза липида A (Lipid A acylase PagP), принадлежащая Escherichia coli. Этот белок относится к классу трамсмембранных белков, имеющих структуру β-бочонка (Рис.1) и участвует в биосинтезе липида A. Также известно, что он может вызывать невосприимчивость к катионным антимикробным пептидам хозяина.
ацилаза липида A Escherichia coli локализована во внешней мембране. Согласно информации из баз данных, в толщину гидрофобная часть белка составляет 24.7 Å, что подтверждается измерением растояния с помощью инструмента Measure distance в программе Jmol.
Согласно базе данных, белок имеет следующие трансмембранные участки: 1(22-30),2(53-58),3(65-74),4(81-94),5(101-114),6(121-133),7(136-144),8(153-161). Средняя длинна трансмембранного участка - 11 aa.
| Тип белка | Трансмембранный белок |
| Класс | бета-бочонок |
| Суперсемейство | Aцилазы липида A |
| Семейсво | Aцилазы липида A |
| Организм | Escherichia coli |
| Локализация | Внешняя мембрана Грам-отрицательной бактерии |
| Uniprot ID | PAGP_ECOLI |
| PDB | 3gp6 |
| Толщина гидрофобного слоя | 24.7 |
| Число трансмембранных структур | 8 |
| Средняя длина β-листа | 11 aa |
2. DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
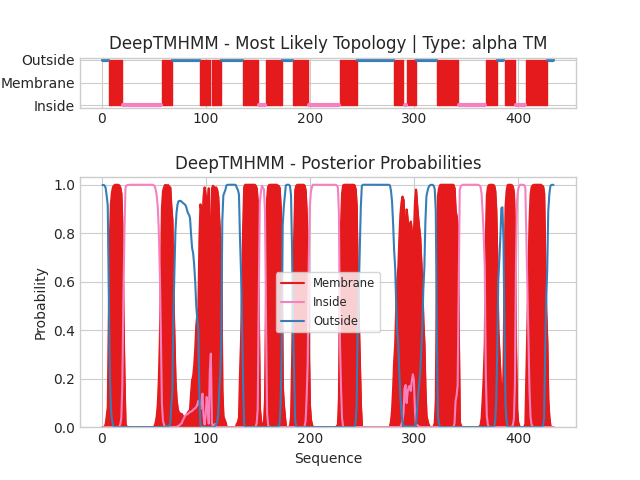
На нижней схеме представленны вероятности пренадлежности аминокислотного остатка к одной из групп: трансмембранная α-спираль (красный), локализация во внешней среде (синий), локализация в цитоплазме (розовый), a на вехней наиболее вероятная топология
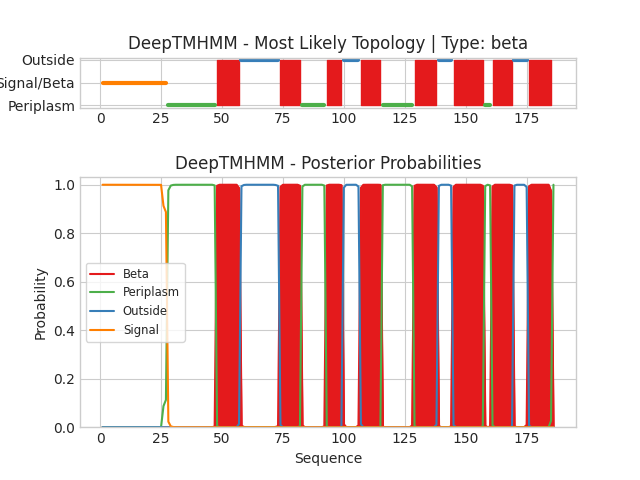
На нижней схеме представленны вероятности пренадлежности аминокислотного остатка к одной из групп: сигнал локализации на внешней мембране (оранжевый), β-листовой трансмембранный участок (красный), локализация во внешней среде (синий), локализация в периплазме (зеленый), а на верхней наиболее вероятная топология.
Предсказания трансмембранных элементов с помощью DeepTMHMM проводились для α-спирального белка YHFT_ECOLI и β-листового PAGP_ECOLI. На вход алгоритму были поданы соответсвующие fasta-файлы YHFT_ECOLI.fasta и PAGP_ECOLI.fasta, на выходе были получены следующие файлы: YHFT_ECOLI_results.md и PAGP_ECOLI_results.md.
Для PAGP_ECOLI было предсказано 8 трансмембранных участков со средней протяженностью 9.6 aa, что значительно меньше, чем средняя протяженность, посчитанная по данным из OPM, тем не менее число трансмембранных участков предсказано верно и их координаты примерно совпадают с указанныим в OPM, если учесть, что в OPM белок был без сигнальной последовательности. Если сравнить предсказание с аннотацией трансмембранных участков из UniProt, можно сделать вывод, что DeepTMHMM довольно точно может предсказать их положение, однако длины этих участков могут слегка не соотвествовать достоверным значениям.
Для YHFT_ECOLI было предсказано 14 трансмембранных участков протяженностью от 11 до 21 aa. В тоже время в UniProt указаны 12 трансмембранных участков с длинной 21. При этом в результатах DeepTMHMM вызывают подозрения 2 пары кототких трансмембранных участков, между которыми есть области с небольшой вероятностью (0.1-0.3)цитоплазматической локализации. Также, кажется DeepTMHMM неправильно предсказал мембрану: т.к. E. coli грамотрицательная бактерия, какая-то часть участков белка должна находиться в периплазме, тем не менее на схеме выданной DeepTMHMM есть только цитоплазма и внеклеточная среда. В базе данных BioCyc указано, что белок находится на внутренней мембране, из чего можно предположить, что внешняя среда на схеме соответствует периплазме.
3. PPM: Предсказание положения выданного белка в мембране
Для предсказания положение белка в мембране с помощью PPM был взят выданный белок YHFT_ECOLI - неохарактеризованный белок YhfT, он принадлежит E. coli, для которого известно что он лежит на внутренней мембране. PPM был запущен со следующими параметрами:
| Number of Membranes | 1 |
| Type of membrane | Gram-negative bacteria inner membrane |
| Allow curvature | no |
| Topology (N-ter) | out (из предсказания DeepTMHMM) |
| Include heteroatoms, excluding water and detergents, for positioning in membrane: | no |
На основе данной структуры, предсказанной с помощью AlphaFold было получено предсказание расположения данного белка в мембране и определенны некоторые параметры
| Тип белка | Трансмембранный белок |
| Организм | Escherichia coli |
| Локализация | Внутренняя мембрана Грам-отрицательной бактерии |
| Uniprot ID | YHFT_ECOLI |
| Толщина гидрофобного слоя | 28.7 ± 0.6 Å |
| Число трансмембранных структур | 14 |
| Средняя длина трансмембранного участка | 19.3 aa |
Координаты трансмембранных участков: 1( 3- 21), 2( 53- 72), 3( 77- 88), 4( 94- 116), 5( 126- 151), 6( 155- 173), 7( 179- 196), 8( 228- 245), 9( 274- 294),10( 310- 317),11( 321- 339),12( 358- 384),13( 385- 403),14( 404- 425)
.png)
4. Сравнение алгоритмов предсказания трансмембранных спиралей
Предсказания для PAGP_ECOLI:
Для удобства сравнения из данных DeepTMHMM вычтена длянна сигнала
DeepTMHMMТрансмембранные участки: 1(24-33), 2(50-58), 3(69-75), 4(83-91), 5(105-124), 6(121-133), 7(137-145), 8(152-161)
OPMТрансмембранные участки: 1(22-30), 2(53-58), 3(65-74), 4(81-94), 5(101-114), 6(121-133), 7(136-144), 8(153-161)
Из сопоставления видно, что DeepTMHMM смог найти все 8 трансмембранных участков в нужных местах, хотя и с небольшими расхождениями в 1-4 остатка.
Предсказания для YHFT_ECOLI:
DeepTMHMMТрансмембранные участки: 1(7-20), 2(58-68), 3(95-104), 4(106-115), 5(136-150), 6(158-173), 7(184-198), 8(229-245), 9(281-290), 10(293-302), 11(322-342), 12(369-380), 13(387-397), 14(408-428))
OPMТрансмембранные участки: 1(3-21), 2(53-72), 3(77-88), 4(94-116), 5(126-151), 6(155-173), 7(179-196), 8(228-245), 9(274-294), 10(310-317), 11(321-339), 12(358-384), 13(385-403), 14(404-425))
Из сопоставления видно, что DeepTMHMM и PMM нашли одинаковое количество участков, однако не все из них соответсвуют друг другу. Так, например, участок 3(77-88) PPM не имеет соответсвия в результатах DeepTMHMM, а участок 4(94-116) PPM соответсвует 2 участкам DeepTMHMM: 3(95-104), 4(106-115),а участки 10 тоже не перекрываются. Остальные участки более менее соответсвуют друг другу, с отклонениями 0-10 aa.
5. База данных TCDB
PAGP_ECOLI отсутствует в TCDB: в UniProt был найден его AC и вставлен в строку поиска TCDB.
YHFT_ECOLI найден в TCDB, его код 9.B.118.1.1, он означает слудующее
- 9 - Неполностью охарактеризованные транспортные системы
- B - Предпологаемые транспортные системы
- 118 - Семейство белков YhfT, содержащих 11 или 12 трансмембранных участков.
- 1.1 - YhfT, белок c 11 или 12 трансмембранными участками