Ортологи и паралоги, визуализация
Задание 1. Составление списка гомологичных белков, включающих паралоги
В таблице ниже представлены отобранные мною бактерии
| Название | Мнемоника |
| Lactococcus lactis subsp. cremoris | LACLA |
| Lactobacillus acidophilus | LACAC |
| Lactobacillus delbrueckii subsp. bulgaricus | LACDA |
| Listeria monocytogenes serovar 1/2a | LISM0 |
| Geobacillus kaustophilus | GEOKA |
| Bacillus anthracis | BACAN |
| Bacillus subtilis | BACSU |
Чтобы найти в своих бактериях достоверные гомологи белка CLPX_ECOLI:
1)Я объединила файлы с полными протеомами бактерий из таблицы в один файл командой cat *.fasta > proteomes.fasta
2)Далее создала базу данных для BLAST makeblastdb -dbtype prot -in proteomes.fasta -out proteomes
После выполнения команды в соответсвующей директории повились три файла с разными расщирениями.
3)Непосредственно поиск гомологов белка CLPX_ECOLI по базе данных, созданной в пункте 2), осуществлялся командой
blastp -query CLPX_ECOLI.fasta -db proteomes -evalue 0.001 -out CLPX_ECOLI.blastp
Файл с находками из выдачи BLAST
Задание 2. Реконструкция и визуализация
Чтобы реконструировать дерево найденных гомологов:
1) Я зашла на Uniprot, где открыла меню retrieve/ID mapping, в пустое окошко вставила идентификаторы.
В результате у меня получилось загрузить последовательности всех найденных гомологов белка CLPX_ECOLI в формате fasta.
2) Из полученного фаста-файла я удалила описания последовательностей, оставив только названия. (Ctrl + K)
Это было сделано, чтобы подписи к листьям дерева, построенного в программе MEGA, не были громоздкими
3) В программе MEGA последовательности были выровняны программой muscle. Далее я построила филогенетическое дерево методом Neighbor-Joining.
Реконструкция дерева в Newick-формате.
Считая дерево реконструированным верно, я могу указать три пары паралогов: CLPX_GEOKA и CLPX_BACAN, HSLU_LACAC и HSLU_LACDA, CLPE BACSU и CLPE LACLA, а также 3 пары ортологов: CLPX_LISMO и HSLU_LISMO, Q5FKR6_LACAC и Q5FMA3_LACAC, FTSH_BACSU и CLPY_BACSU.
Ниже изображены разные ортологичные группы то есть наборы попарно ортологичных белков
(группы, содержащие три и менее последовательностей, были оставлены чёрными):
На следующем изображении можно наблюдать "схлопнутые" ортологичные группы:
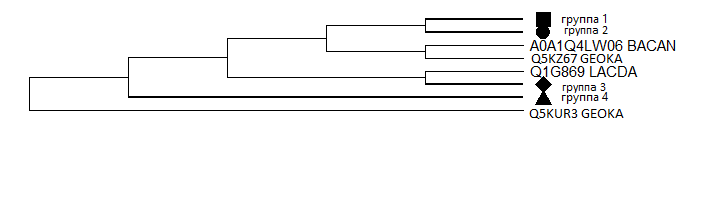
Группа 1 включает в себя 3 белка (CLPX, Q5FKR6, Q1GAP8) из всех бактерий. Реконстриурованная филогения соответствует филогении бактерий.
Группа 2 включает 2 белка (HSLU, CLPY) из 6 бактерий (GEOKA, BACAN, BACSU, LISMO, LACAC, LACDA). Реконстриурованная филогения не соответствует филогении бактерий.
Группа 3 включает в себя 5 белков (Q5FHW6, Q5L436, CLPE, Q8Y8B1, Q1GB74) из 6 бактерий (GEOKA, LACLA, BACSU, LISMO, LACAC, LACDA). Реконстриурованная филогения не соответствует филогении бактерий.
Группа 4 включает 6 белков (Q1GBN8, Q5FMA3, FTSH, Q8YAC6, A0A347ZXP1, Q5L3T1) из всех бактерий.
соответствует ли реконструированная филогения белков филогении бактерий). Реконстриурованная филогения не соответствует филогении бактерий.