Обзор протеома Lactobacillus futsaii.
Машкова София Дмитриевна.
Факультет биоинженерии и биоинформатики, Московский государственный университет имени М. В. Ломоносова, Ленинские горы д. 1 стр. 73, 119234, Москва, Россия.
Резюме.
Получены сведения для общего ознакомления с протеомом lactobacillus futsaii при помощи сервиса Google Sheets. Результаты анализа представлены в удобной для визуализации форме таблиц и гистограмм, содержащих информацию о длинах белковых и нуклеотидных последовательностей, а также о характере и закономерностях их распределения.
Ключевые слова:
lactobacillus futsaii, lactobacillus, протеом, геном, ДНК.1.Введение.
Lactobacillus futsaii - грамположительная аспорогенная факультативно анаэробная бактерия, впервые описанная группой китайских ученых Shiou-Huei Chao, Yuko Kudo, Ying-Chieh Tsai, Koichi Watanabe в 2012 году. Колония представлена неподвижными клетками палочковидной формы, встречающимися поодиночке, парами или короткими цепочками.[1] Род Lactobacillus принадлежит семейству Lactobacillaceae и является одним из важнейших в группе молочнокислых бактерий, большинство членов которой превращает углеводы в молочную кислоту. Они, как правило, не патогенны и выполняют положительную роль в питании человека.[2] Ценность представленной группы бактерий вызывает особый интерес к изучению описанного в работе вида, так как он, вероятно, может иметь практическую значимость для человека, например, в пищевой промышленности. Поэтому с целью более детального ознакомления с Lactobacillus futsaii ниже приведен мини-обзор ее протеома для выявления основных закономерностей и особенностей жизнедеятельности.
2. Методы.
Для приведения информации о содержании транскриптома lactobacillus futsaii, взятой из открытого банка NCBI, в удобный для анализа вид, а также обработки полученных данных составлены сопроводительные материалы с помощью программы Google Sheets. Для создания плоских таблиц использованы различные поисковые и математические функции (ВПР (VLOOKUP), CУММ, СЧЁТЕСЛИ, СТЬЮДРАСП), методы фильтрации и сортировки с целью копирования и переноса необходимых данных, относительные и абсолютные ссылки, специальная вставка, связь таблиц, распространение формул и составление гистограмм. Далее полученные таблицы были приведены к презентабельному виду путем условного форматирования.
3. Результаты и обсуждение.
| DNA type | length | sum |
|---|---|---|
| chromosome | 2558218 | 2648769 | plasmid | 37880 | plasmid | 27087 | plasmid | 25584 |
3.1 Описание длин ДНК, составляющих геном.[1]
Геном Lactobacillus futsaii представлен единственной хромосомой и тремя плазмидами (соответствующие длины указаны в таблице 1). Плазмиды, как правило, содержат гены, повышающие приспособленность организмов к неблагоприятным факторам окружающей среды (резистентность к антибиотикам) и являются средством горизонтального переноса генов между соседними клетками. Наличие плазмид- достаточно распространенный признак для рода lactobacillus. Примерно 38% видов данного рода содержат плазмиды, причем их количество варьирует от 1 до 10[3]. Согласно табличным данным длина хромосомной ДНК входит в диапазон средних длин геномов прокариот (от 106 до 107 пар нуклеотидов [4]). Размер плазмид, напротив, относительно мал, так как крупные плазмиды могут достигать длины 600 тыс. пар оснований [5].
| Genome length | 2648769 |
| Proteins | 2548 |
| Pseudogenes | 57 |
| tRNA | 55 |
| rRNA | 12 |
| tmRNA | 1 |
| SRP_RNA | 1 |
| RNase_P_RNA | 1 |
| ncRNA | 0 |
3.2 Описание встречаемости генов различных типов.[2]
В ходе работы был проведен анализ встречаемости генов различных типов (таблица 2).Результаты показывают, что в геноме бактерии присутствует 57 псевдогенов. Псевдогенами именуются гены, которые не транскрибируются вследствие сдвига или нарушения рамки считывания, а также появления преждевременных стоп-кодонов [6]. В геноме наблюдается тенденция организации тРНК-генов в кластеры. Практически половина тРНК-генов сконцентрирована на одном участке обратной цепи ДНК(информацию можно найти в сопроводительных материалах, лист tRNA_genes). Гены рибосомных РНК также расположены кластерами, причем прослеживается следующая закономерность: обнаружено 4 кластерных участка, каждый из которых содержит по три гена рРНК (лист rRNA_genes). В дальнейшем можно заняться более детальным изучением распределения генов РНК, поиском тандемных повторов и промоторных участков с целью определения точного характера транскрипции. Оставшиеся гены представлены в единичном экземпляре и выполняют важные регуляторные функции в клетке: расщепление РНК, терминация синтеза белка, разрушение дефектных пептидов и т.д.
3.3 Нуклеотидный состав ДНК генома.[3],[4]
| A | 853934 |
| T | 847504 |
| G | 476134 |
| C | 471197 |
| Sum | 2648769 |
С помощью готовых команд bash проведен анализ количества нуклеотидов. Результат анализа (таблица 3) показывает, что суммарно число представленных ниже четырех нуклеотидов в точности совпадает с упомянутым ранее размером генома, следовательно, ДНК состоит только из аденина, гуанина, цитозина и тимина, что подтверждает свойство универсальности генетического кода. Можно полагать, что количество аденина совпадает с количеством тимина, а количество гуанина - с количеством цитозина, что полностью согласуется с правилом Чаргаффа.
Согласно данным из таблицы 4 частота встречаемости пар AT выше, чем GC, что вполне ожидаемо. Высокая концентрация CG-пар повышает устойчивость молекулы ДНК, что характерно для термофильных бактерий, каковой не является lactobacillus futsaii.
| sequence | GC | AT |
|---|---|---|
| NZ_CP040736.1 | 0,36 | 0,64 |
| NZ_CP040737.1 | 0,39 | 0,61 |
| NZ_CP040738.1 | 0,39 | 0,61 |
| NZ_CP040739.1 | 0,35 | 0,65 |
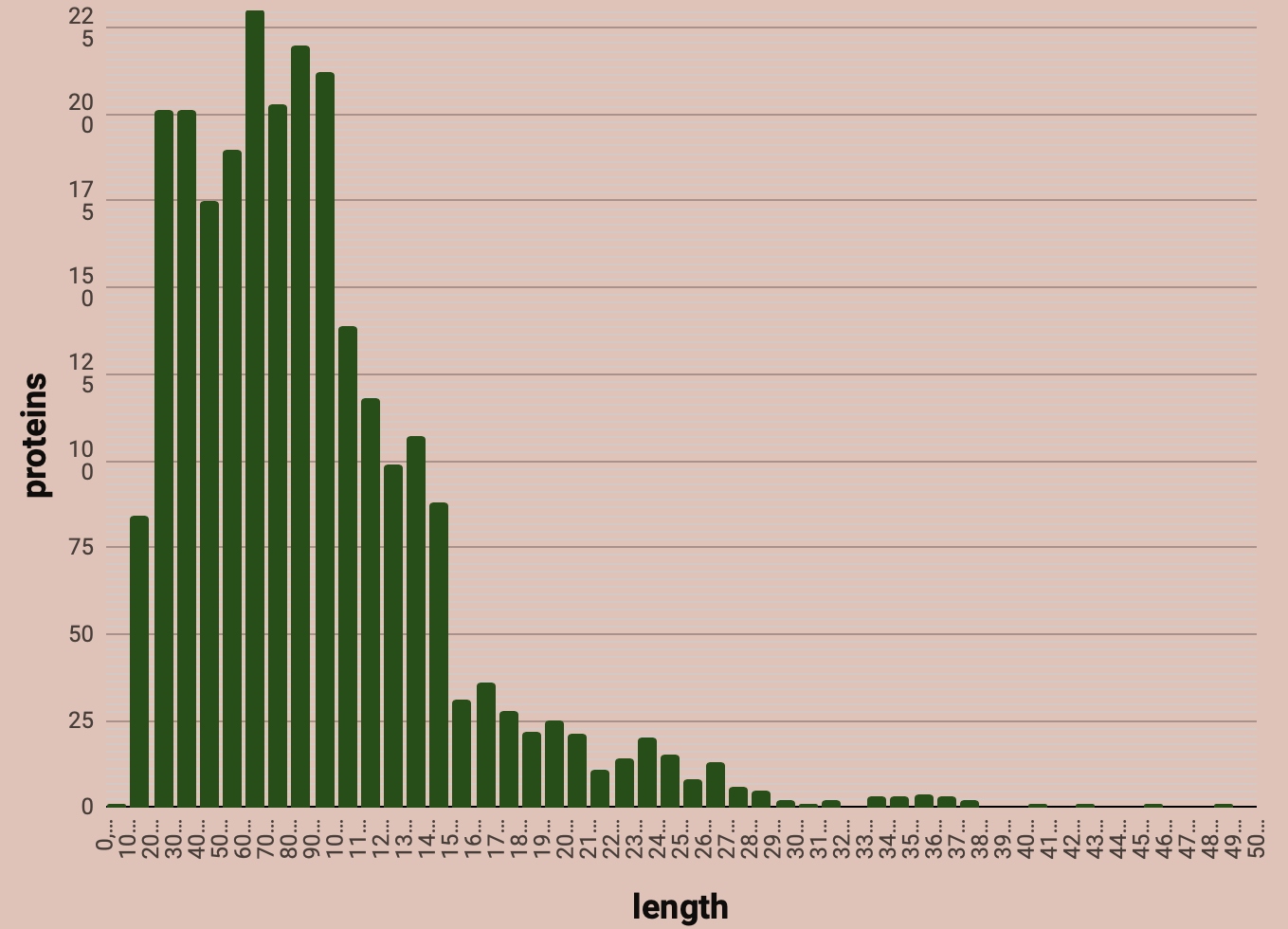
3.4. Анализ белковых последовательностей.[5],[6]
В процессе работы произведен анализ длин последовательностей белков (рис. 1). Результаты показывают, что наиболее распространены белки длиной от 200 до 900 аминокислотных остатков, причем максимальное количество приходится на диапазон длин от 600 до 700. Такие величины характерны для регуляторных белков и ферментов, вовлеченных в основные биохимические циклы и процессы белкового синтеза.Согласно данным из таблицы 5 гипотетические белки составляют примерно 16% процентов от всего протеома, что говорит о наличии скудных данных и недостаточной изученности lactobacillus futsaii.
| amount of hypothetical proteins | all proteins | percentage of hypothetical proteins |
|---|---|---|
| 409 | 2548 | 16,05180534 |
Заключение.
Lactobacillus futsaii является типичным представителем рода lactobacillus. В ходе исследования выявлены некоторые закономерности, требующие более детального изучения. Также необходимо проверить некоторую информацию на достоверность, чтобы по возможности идентифицировать гипотетические белки и выявить их функциональные особенности.
Благодарности.
Автор искренне выражает благодарность преподавателям курса биоинформатики за терпение и передачу бесценных знаний, а также Ренате Бетеньковой, Олесе Богомаз и Ренату Винникову за взаимный обмен опытом.
Конфликты.
Исследование проведено исключительно в мире и согласии
Сопроводительные материалы.
Ссылка на электронную таблицуСписок использованной литературы.
[1] Shiou-Huei Chao, Yuko Kudo, Ying-Chieh Tsai, Koichi Watanabe. (2012) Lactobacillus futsaii sp. nov., isolated from fu-tsai and suan-tsai, traditional Taiwanese fermented mustard products // international journal of systematic and evolutionary microbiology.- 2012. - Т.62. - выпуск 3.
[2] Википедия, русская, страница с информацией о плазмидах лактобактерий https://ru.wikipedia.org/wiki/Лактобациллы (была доступна 19.11.2020 )
[3] Tsung-Tsan Wang, Byong H. Lee. (published online: 27 Sep 2008) Plasmids in Lactobacillus // Critical Reviews in Biotechnology. volume 17, 1997 - issue 3
[4] Википедия, русская, страница с информацией средних размерах прокариотического генома. https://ru.wikipedia.org/wiki/Геном#Размер_и_структура_генома
[5] Википедия, русская, страница с информацией о размерах плазмид. https://ru.wikipedia.org/wiki/Плазмиды#Размеры_и_численность
[6] Yang Liu, Paul M Harrison, Victor Kunin, Mark Gerstein.(Published: 24 August 2004) comprehensive analysis of pseudogenes in prokaryotes: widespread gene decay and failure of putative horizontally transferred genes // Genome Biology 5, Article number: R64 (2004).