


Цель практикума: определить экзон-экзонную структкру фрагмента эукариотического генома.
Исследуемая последовательность была подана на вход программе GENSCAN. Выбраны настройки для позвоночных (Vertebrate). В результате были предсказаны следующие экзоны (см. табл.1.). Были исключены записи, не соответствующие экзонам: PlyA (сайт полиаденилирования) и Prom (промотор).
| Тип экзона | Цепь | Начало | Конец |
| Начальный | + | 5108 | 5173 |
| Внутренни й | + | 8202 | 8253 |
| Внутренни й | + | 11330 | 11526 |
| Внутренни й | + | 12321 | 12397 |
| Внутренни й | + | 12750 | 12779 |
| Внутренни й | + | 14593 | 14672 |
| Терминальный | + | 18402 | 18571 |
| Терминальный | - | 19179 | 19151 |
| Внутренни й | - | 19443 | 19314 |
| Внутренни й | - | 20581 | 20490 |
Табл. 1. Результаты работы программы GenScan. Координаты предсказанных экзонов.
Далее была запущена программа BLAT на BLAT, которая аналогично BLAST позволяет искать последовательности в геноме с учетом возможной фрагментированности генома.
Был получен список найденных фрагментов генома. Так как в списке было больше одной записи, то я выбрала ту, которая имеет наибольшее сходство с исследуемой мной последовательнотей - имеющую наибольший Score (24404) и 100% identity. Список наилучших находок вы можете просмотреть на рисунке 1.
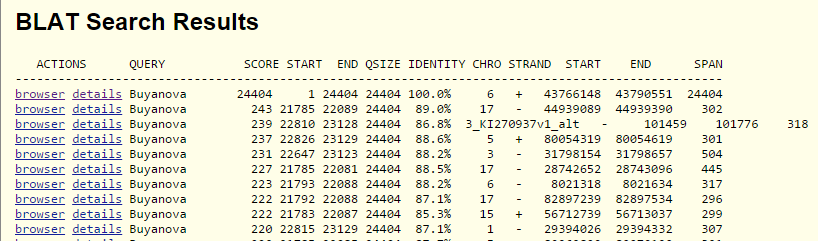
Рис. 1
Переключатели Blat Sequence в группе Mapping and Sequencing Tracks, а также переключатели Human mRNAs и Spliced ESTs в группе mRNA and EST Tracks, были поставленны на pack. Остальные на hide. Это неободимо, так как в задании ислледуется именно сплайсинг. Остальные переключатели поставьте на hide. Полученная кртинка представлена на рисунке 2.
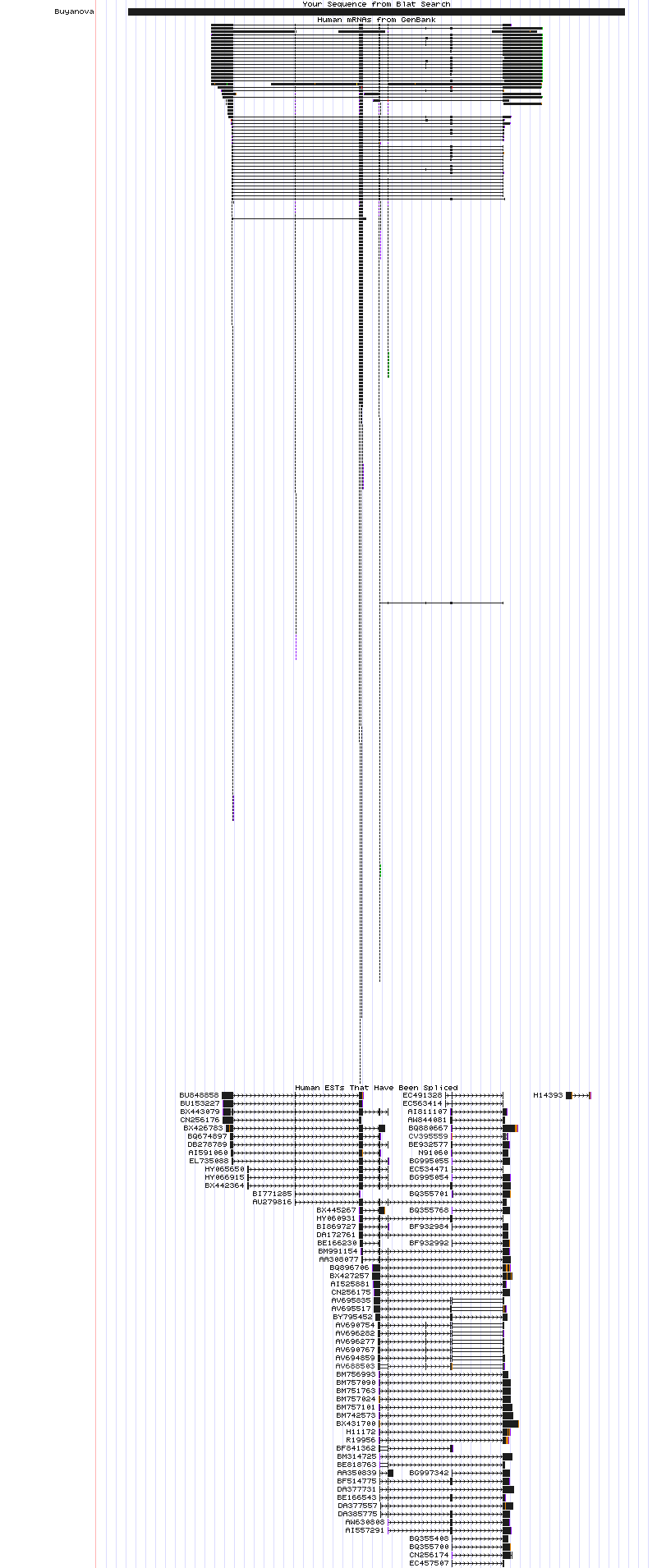
Рис. 2 Выравнивание, визуалтзтрованное Genome Browser
К сожалению, на полученной картинке видно крайне мало информации, из-за большой длины фрагмента. Я отдельно выделила фрагмент выравнивания на рис.3, для того, чтобы наглядно показать некоторые примеры альтернативного сплайсинга.

Рис. 3. Фрагмент выравнивания. Альтернативный донорный сайт между M2781 и S85192 выделен зеленым, кассетные экзоны (например между JA810025 и FW406918) красным.
Предсказание генов и экзон-интронной структуры проводилось для фрагмента ДНК киви (Actinidia chinensis). Геном киви не является хорошо аннотированным (в отличие от генома человека), поэтому для предсказания генов фрагмента генома этого растения невозможно использовать GENSCAN или Genome Browser. Поэтому разметка экзон-интронной структуры генов этого фрагмента производилась с помощью BLASTX (поиск гомологичных последовательностей белков, соответствующих участкам исследуемого фрагмента ДНК).
Был исключен (Exclude) поиск по моделям и пробам среды (Models (XM/XP) и Uncultured/environmental sample sequences) и выбрана база данных Swiss-Prot, т.к. по уже указанным причинам нужно стараться использовать не предсказанные, а экспериментально наблюдавшиеся белки. Сначала был запущен поиск по всем таксонам, и так вышло, что на геном киви выравниваются нуклеотидные последовательности белков из самых разных таксонов, как например серин/треониновая киназа Dictyostelium discoideum (слизевик) и Arabidopsis thaliana (высшее растение), белок, ассоциированный с микротрубочками Arabidopsis thaliana (высшее растение), GATA транскрипционный фактор Arabidopsis thaliana (высшее растение), Валил-тРНК синтетаза из генома Brucella melitensis (бактерия), Транспозон Ty1 из из генома Saccharomyces cerevisiae (дрожжи).
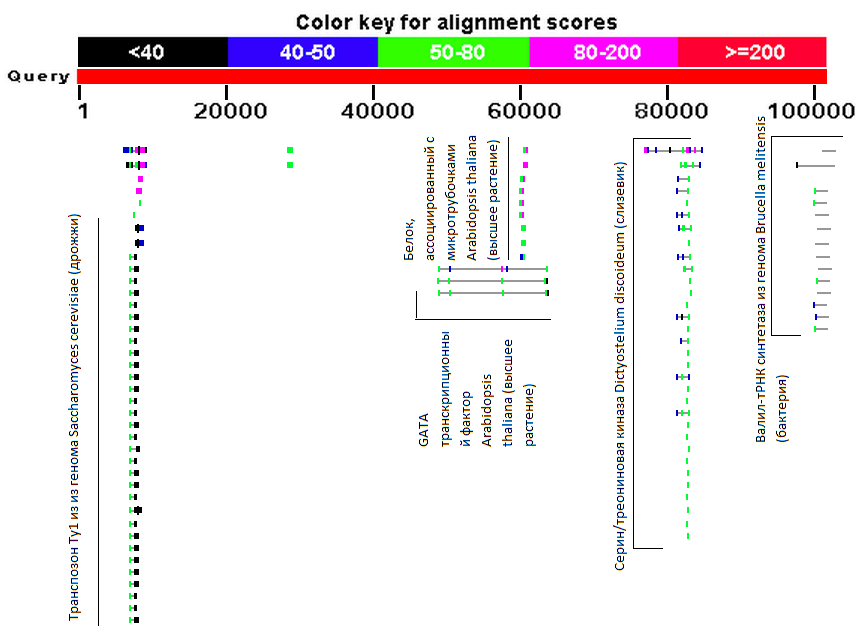
Рис. 4 Визуализированный результат работы BlastX без ограничения выравнивания только по последовательностям высших растений
Далее был запущен BLASTX только по высшим растениям, без учета генома винограда.
Было выявлено 3 чего-то похожего на гены. Интересно, что все найденные предполагаемые гены Киви картируются на геном Резуховидки Таля. Известно, что они пренадлежат к эвдикотам - группе двудольных растений неопределённого ранга, которая используется в системах классификации Покрытосеменных.
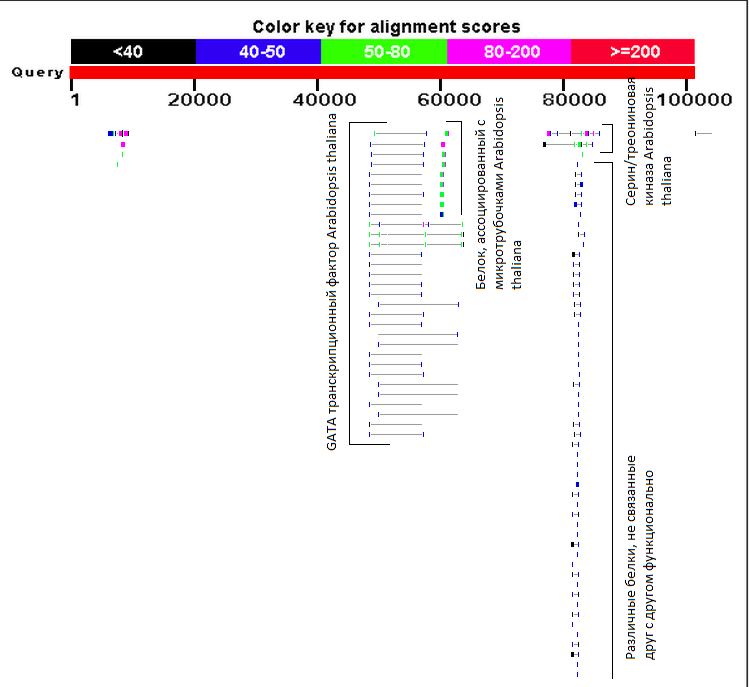
Рис. 5 Визуализированный результат работы BlastX с выравниваниями только по последовательностям высших растений
Предполагаемые экзоны предсказанных генов киви вы можете найти в таблице:
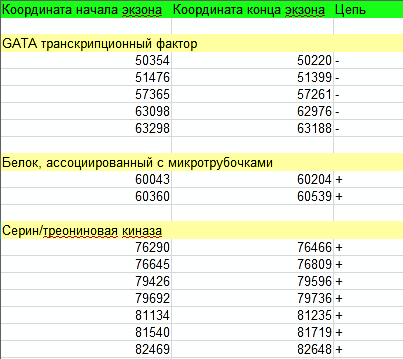
Табл. 2. Координаты экзонов, предсказанные с помощью BlastX.