


Первым заданием было определить, из какой бактерии взята заданная последовательность с помощью программы Megablast на сайте NCBI. В качестве банка был выбран "refseq_genomic". Поиск был ограничен археями и бактериями. В разделе Filters and Masking была убрана галочка с параметра Low complexity regions.
Точное совпадение было участком хромосомы M7 в Methanocaldococcus vulcanius M7. AC записи RefSeq: NC_013407.1, координаты данного фрагмента в записи 1145..1444, является кодирующей последовательностью, кодирует белок CRISPR-associated protein Cas1 с идентификатором GenBank: YP_003246357.1.
Задание 2
В нем было необходимо найти гомологов человеческого белка, начинающегося с максимального количества совпадающих с моей фамилией букв, в геноме африканского слона (Loxodonta africana).
Для начала я нашла белки человека, начинающиеся с bu с помощью следующего запроса:
infoseq sw:bu*_human -only -name -desc -out myproteins
Файл с белками, начинающимися с сочетания BU
Я выбрала белок BUB1B_HUMAN (Mitotic checkpoint serine/threonine-protein kinase BUB1 beta). Это серин-треониновая киназа BUB1 beta митотической контрольной точки. Скорее всего этот важный белок пронаблюдается и в последовательности африканского слона (Loxodonta africana).
На сайте ENA был запущен поиск гомологов. Результаты представлены на рис.1
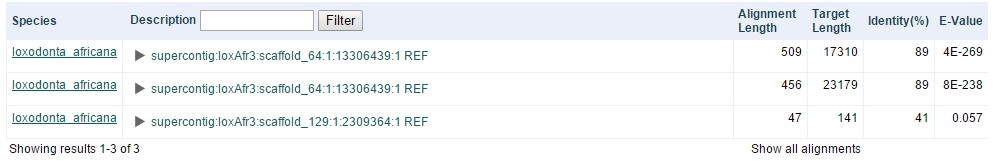
Рис.1. E-value лучшей находки 4E-269, identity 89%, длина выравнивания 509 пар нуклеотидов.
Задание 3
Была выбрана первая последовательность тРНК из файла, содержащего последовательности тРНК организма Pyrobaculum aerophilum str. IM2, в свою очередь взятого с ftp сервера NCBI.
>ref|NC_003364|:21776-21867|His tRNA| [locus_tag=PAEtR42] GCCGCCGTAGTCTAGCGGTTAGGATGGCGGGTTGTGGTCCCGTTGCGGCGAGCCGCGGGAGGACCCGGGT TCAAATCCCGGCGGCGGCCCCA
Был выполнен поиск гомологов данной последовательности с помощью трех разных алгоритмов: megablast, blastn с параметрами по умолчанию, blastn с длиной слова = 7, match/mismatch = 1/-1.
Результаты поиска приведены на рис.2.1 -2.3
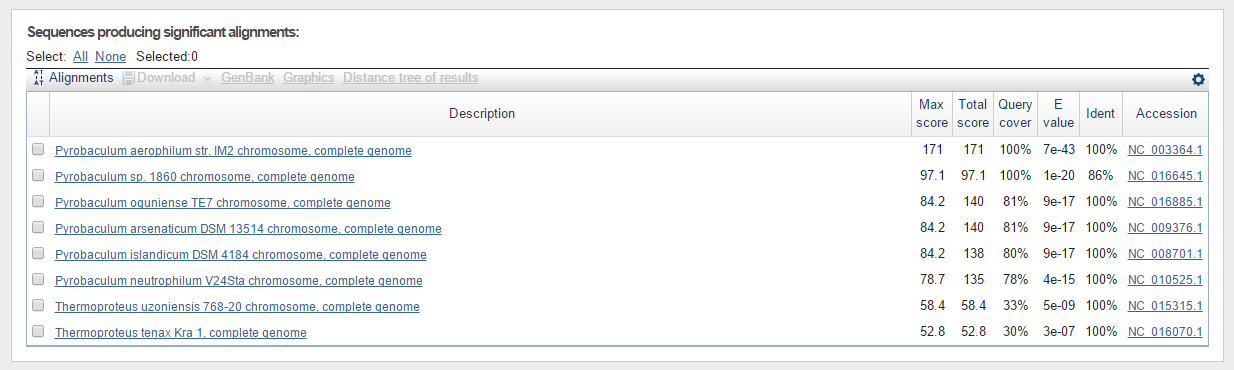
Рис. 1. Поиск гомологов последовательности с помощью megablast. Число находок с e-value < 0,001: 8. Первая запись - это собственно та запись, из которой была взята исследуемая последовательность tRNA.
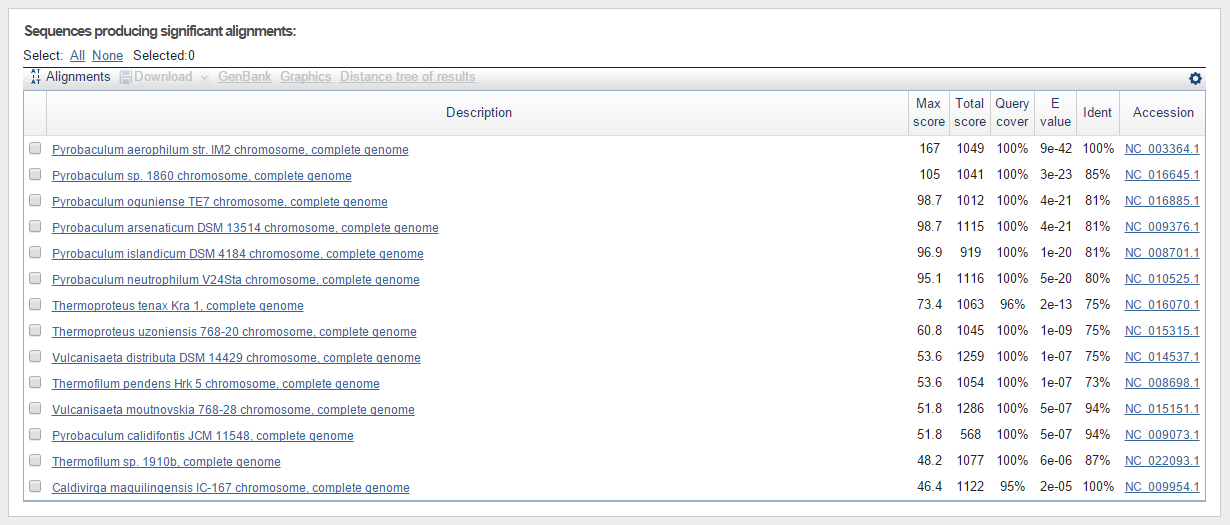
Рис. 1. Поиск гомологов последовательности с помощью blastn с параметрами по умолчанию. Число находок с e-value < 0,001: 14. Первая запись - это собственно та запись, из которой была взята исследуемая последовательность tRNA.
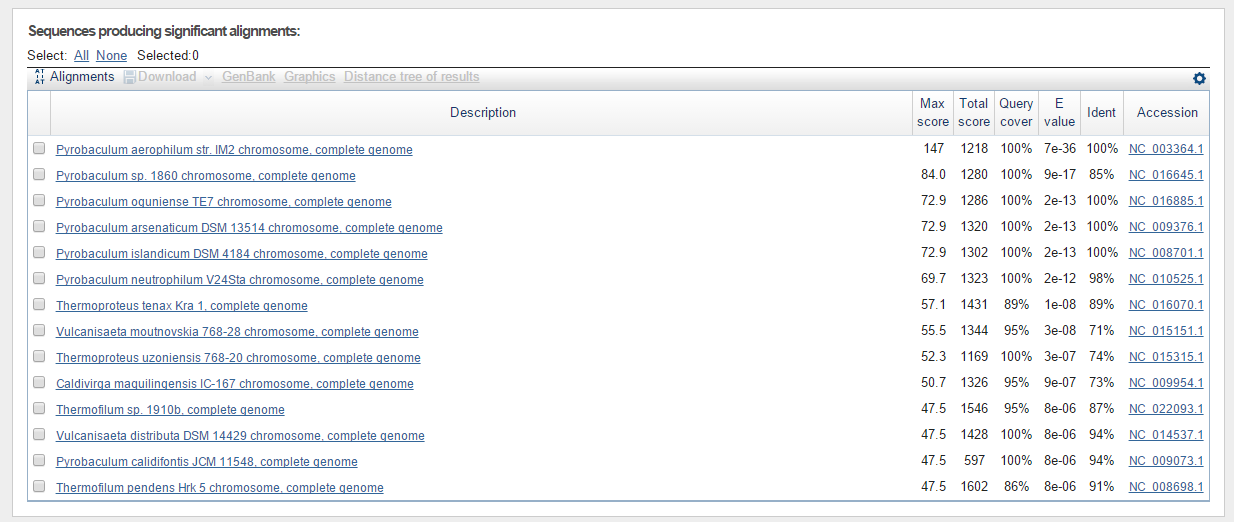
Рис. 1. Поиск гомологов последовательности с помощью blastn с измененными параметрами. Число находок с e-value < 0,001: 14. Первая запись - это собственно та запись, из которой была взята исследуемая последовательность tRNA.
В первом приближении, мне показалось очень странным, что гистидиновая тРНК, которая исследовалась мной в задании имеет настолько мало гомологов в других организмах того же порядка Thermoproteales. Предполагаю, что это связано с немночисленностью видов, а также с небольшим количеством отсеквенированных геномов, так как археи этого порядка - экстремофилы (гипертермофилы) и поэтому являются достаточно специфическими объектами исследования.
Стоит отметить, что megablast нашел меньше последовательностей, но с наиболее значимыми e value, score и identity, тогда как blastn больше последовательностей, но с менее значительными указвнными параметрами. Identity всех, кроме одного, было 100%. Это объясняется принципом работы и предназначением разных алгоритмов. Megablast был создан и используется для поиска наиболее точно совпадающих длинных выравниваний с искомой последовательностью. Этот алгоритм может быть удобен для определения, к какому организму принадлежит полученный сиквенс. Алгоритм же blastn разбивает последовательность на короткие слова и ищет выравнивания этих слов, делая этот алоритм более чувствительным к похожим, но не в точности совпадающим коротким последовательностям. Это удобно для поиска похожих последовательностей в родственных организмах.