


1) Поиск однонуклеотидных полиморфизмов и инделей
Однонуклеотидный полиморфизм (ОНП, англ. Single nucleotide polymorphism, SNP, произносится как снип) — отличия последовательности ДНК размером в один нуклеотид (A, T, G или C) в геноме (или в другой сравниваемой последовательности) представителей одного вида или между гомологичными участками гомологичных хромосом. [1] Под инделями подразумеваются делеции (потеря учатка хромосомы) и инсерции (вставка в нуклеотидной последовательности).
В конце практикума "Картирование на референсный геном" был получен файл в формате .bam, содержащий информацию о ридах, откартированных на геном митохондрии и хлоропласта резуховидки. Чтобы сравнить референсные геномы с откартированными ридами, файл bwa_align_sorted.bam (содержащий откартированные, отсортированные и проиндерсированные риды) вместе с файлом, содержащим исследуемые геномы, был подан на вход программе samtools (Опции: -g для файла в формате bcf -u в несжатом виде, -f для входного файла референсного генома в формате fasta.):
samtools mpileup -u -g -f arabidipsis_chl_mito.fasta bwa_align_sorted.bam > arabidopsis_compare.bcf
Далее с помощью программы bcftools был получен список инделей и однонуклеотидных полиморфизмов:
bcftools view -v -c -g arabidopsis_compare.bcf > arabidopsis_ind_snp.vcf
Подсчитано их количество:
grep 'INDEL;' arabidopsis_ind_snp.vcf| wc -l
grep 'DP=' arabidopsis_ind_snp.vcf| wc -l
Риды содержат 284 индель и 666 однонуклеотидных полиморфизмов.
Сборка хлоропласта и митохондрии
Для сборки генома был использован пакет Velvet, который осуществляет сборку генома из ридов на основе графа де Брёйна. Пакет состоит из друх программ. Первая - velveth - создаёт на основе входных ридов банк из заданного количества k-меров (последовательностей из k нуклеотидов). Вторая - velvetg - производит сборку контигов на основе графа де Брёйна из этих k-меров.
Значение N50, выдаваемое программой при сборке генома позволяет оценить качество сборки. Если суммарная длина всех контигов L, то N50 называется длина такого контига, что суммарная длина всех контигов больше выбранного равна L/2. В задании было предложено начать с 35, затем варьировать до 15 в меньшую сторону или до 97 в большую. При запуске Velvet выдал "Velvet can't handle k-mers as long as 35! We'll stick to 31 if you don't mind." Поэтому поиск наилучшего значения k-mer осуществлялся путем уменьшения значения k-mer. Наилучшее значение N50 было достигнуто при длине k-mer равной 25
Программвы пакета velvet были запущены несколько раз, оптимальное значение k-mer: 25 при максимальном N50: 247
velveth velveth_dir31 31 -fastq Ath_tae_CTTGTA_L003_R1_002_trimmed.fastq velvetg velveth_dir31 -cov_cutoff auto Final graph has 336516 nodes and n50 of 216, max 8091, total 40643248, using 0/3837711 reads
velveth velveth_dir30 30 -fastq Ath_tae_CTTGTA_L003_R1_002_trimmed.fastq velvetg velveth_dir30 -cov_cutoff auto Velvet can't work with even length k-mers, such as 30. We'll use 29 instead, if you don't mind. Final graph has 351159 nodes and n50 of 226, max 7655, total 42669603, using 0/3837711 reads
velveth velveth_dir27 27 -fastq Ath_tae_CTTGTA_L003_R1_002_trimmed.fastq velvetg velveth_dir27 -cov_cutoff auto Final graph has 370660 nodes and n50 of 235, max 7653, total 44724666, using 0/3837711 reads
velveth velveth_dir25 25 -fastq Ath_tae_CTTGTA_L003_R1_002_trimmed.fastq velvetg velveth_dir25 -cov_cutoff auto Final graph has 429328 nodes and n50 of 247, max 7649, total 48419674, using 0/3837711 reads
velveth velveth_dir23 23 -fastq Ath_tae_CTTGTA_L003_R1_002_trimmed.fastq velvetg velveth_dir23 -cov_cutoff auto Final graph has 394854 nodes and n50 of 243, max 7651, total 46682011, using 0/3837711 reads
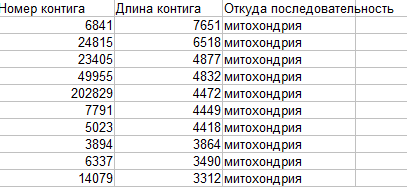
В результате запуска пакета velvet получена папка velveth_dir_25, содержащая несколько файлов, среди которых файл с последовательностями полученных контигов (contigs.fa) и файл со статистикой, в том числе о длине и номере соотвутствующих контигов (stats.txt). Были определены номера 10-ти самых длинных контигов, скопированы в longest_contigs.fa и с помощью локального blast был произведен поиск этих контигов в геномах митохондрии и хлоропласта.
makeblastdb -in arabidipsis_chl_mito.fasta -dbtype nucl
blastn -task megablast -query longest_contigs.fa -db arabidipsis_chl_mito.fasta -out blast_contigs.txt -outfmt 7
Выдачу blast можно увидеть здесь. Инофрмацию о 10 самых длинных контигов, найденных Velvet можно найти в табл. 1. и в файле
Таблица 1. Информация о 10 самых длинных контигах, полученных в результате сборки геномов хлоропласта и митохондрии резуховидки.
Ссылки
[1] https://ru.wikipedia.org/wiki/Однонуклеотидный_полиморфизм