


В первом задании было предложено построить модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA. Полученные структуры A и B форм ДНК (5 повторов GATC) и структура Z формы ДНК (10 повторов GC) вы можете найти по приведенным ссылкам A форма, B форма, Z форма.

Рис. 1.1 Изображение структуры A формы ДНК, полученное с помощью PDB

Рис. 1.2 Изображение структуры B формы ДНК, полученное с помощью PDB

Рис. 1.3 Изображение структуры Z формы ДНК, полученное с помощью PDB
В задании 2 было необходимо выделить некоторые части А формы ДНК. Результат выполнения задания см. на рис.2.
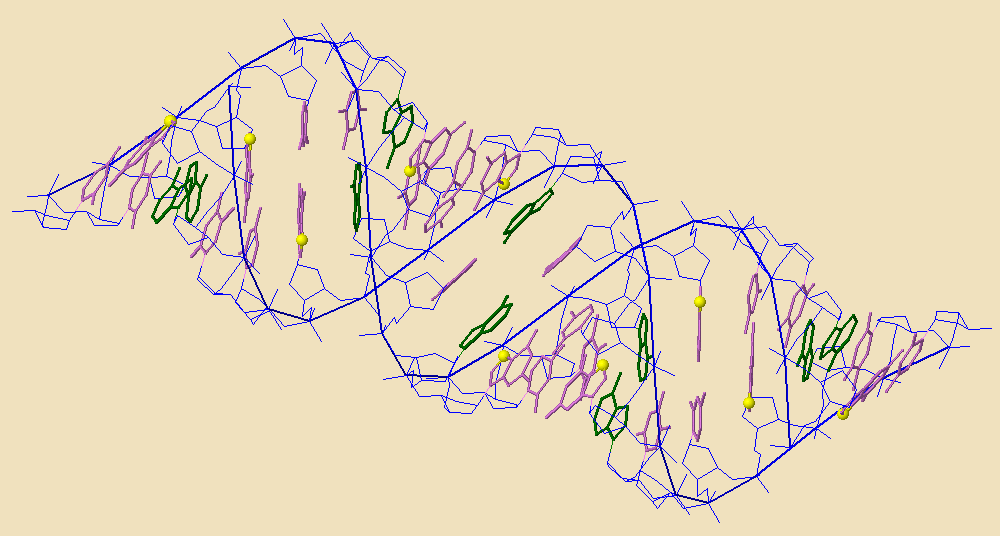
Рис. 2 Сахарофосфатный остов ДНК выделен синим, нуклеотиды выделены фиолетовым, все аденины выделены зеленым, атом N7 во всех гуанинах выделен желтым в шариковой модели.
Следующим заданием было исследовать структуры ДНК-белковых комплексов и тРНК из таблицы на наличие разрывов. В структуре с идентификатором PDB 1HDD разрывов найдено не было (см. рис. 3.1). В структуре тРНК имелись разрывы, даже при тщательном рассмотрении в различных моделях (см. рис. 3.2)
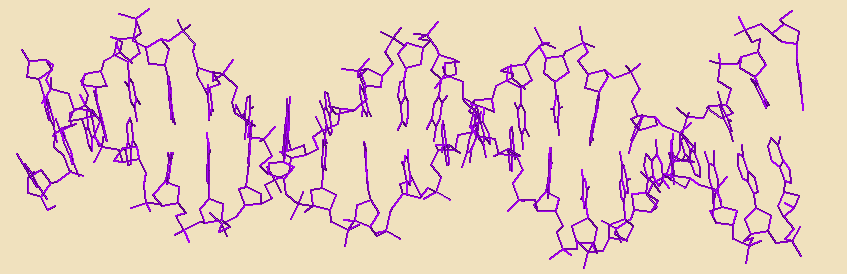
Рис. 3.1 Проволочная модель трехмерной структуры ДНК из 1HDD. Разрывов не наблюдается
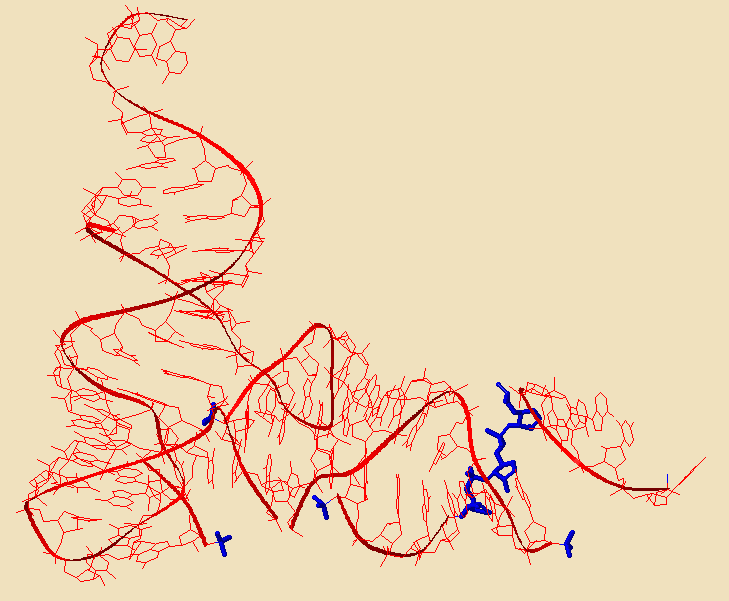
Рис. 3.2 Трехмерная структура тРНК в проволочной модели и дополнительно остов в ленточной модели. Синим выделены места, в котороых при использовании комманд select backbone -> wireframe были видны разрывы. Оказалось, что нуклеотиды 539, 540, 541 модифицированные (см. рис. 3.3), на позициях 546, 516, 531, 533 нуклеотидов отражены только некоторые атомы, из чего можно заключить, что это ошибки ренгеноструктурного анализа. На позициях 517, 532 нуклеотидов не оказалось, я предполагаю, что там есть разрыв, либо это опять ошибка рентгеноструктурных данных.
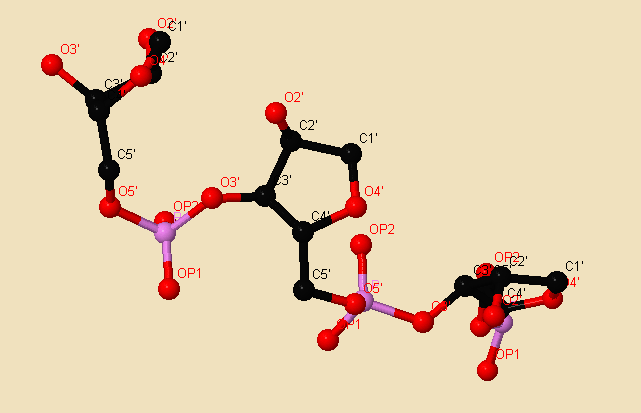
Рис. 3.3 Модифицированные нуклеотиды 539-541
В следующем задании было необходимо найти и исследовать большие и малые бороздки ДНК во всех трех формах ДНК. Для начала рассмотрим структуру B формы ДНК.
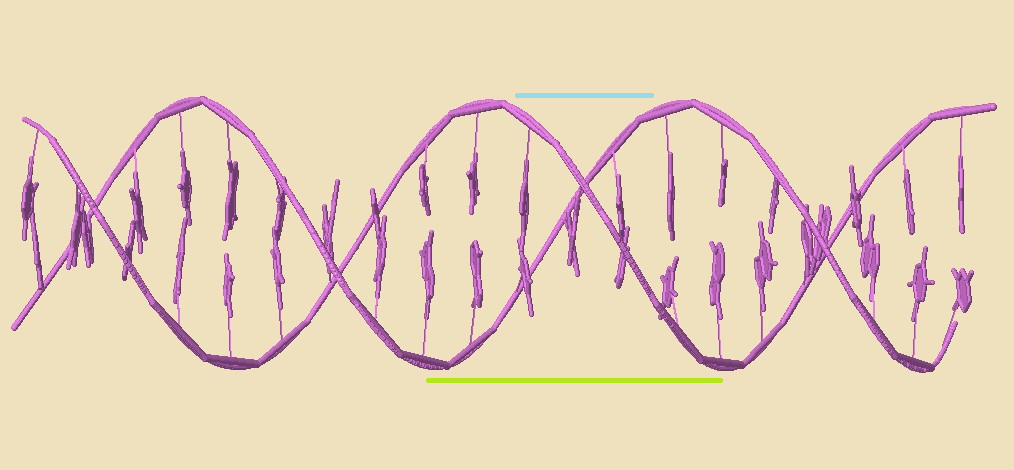
Рис. 4.1 Зеленым отмечена большая бороздка, голубым малая бороздка.
При изучении ориентации атомов тимина по отношению к малой и большой бороздкам была получена следующая информация (подробнее см. на рис. 4.2 и рис. 4.3):
В сторону малой бороздки обращены - N1, C2, O2.
В сторону малой бороздки обращены - N1, C2, O2.
В сторону большой бороздки обращены - С4, С5M, С6, С5М, О4
Остальные атомы - N3
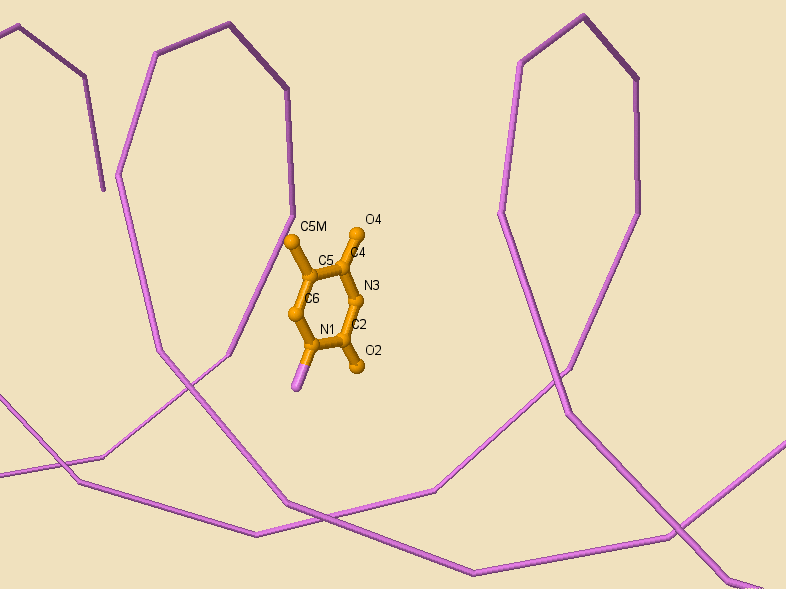
Рис. 4.2 Положение тимина относительно бороздок. Пространственная структура.
Рис. 4.3 Положение тимина относительно бороздок. Красным выделены атомы, обращенные к большой бороздке, синим - к малой.
Изучение А формы ДНК проводилось аналогично. В A-форме атомы тимина обращены ровно в противоположенные стороны с атомами в В-форме: в сторону большой бороздки обращены атомы: С2, N1, O2. В сторону малой бороздки обращены атомы: С4, С5, O4, С5M. В Z-форме тиминов нет.
Дальнейшее изучение исследуемых структур ДНК позволило выявить информацию о ширине бороздок (см. рис. 5.1-3 и табл.1)
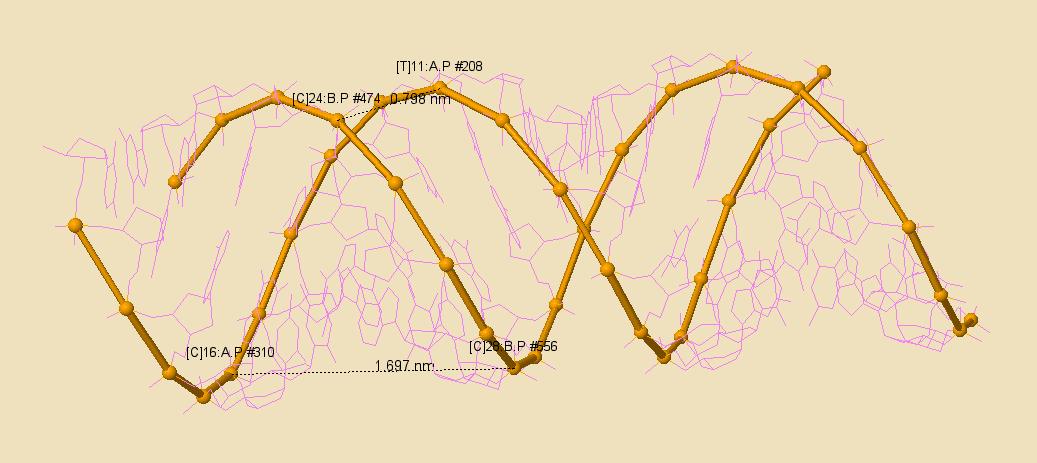
Рис. 5.1 Изучение большой и малой бороздок ДНК в А форме
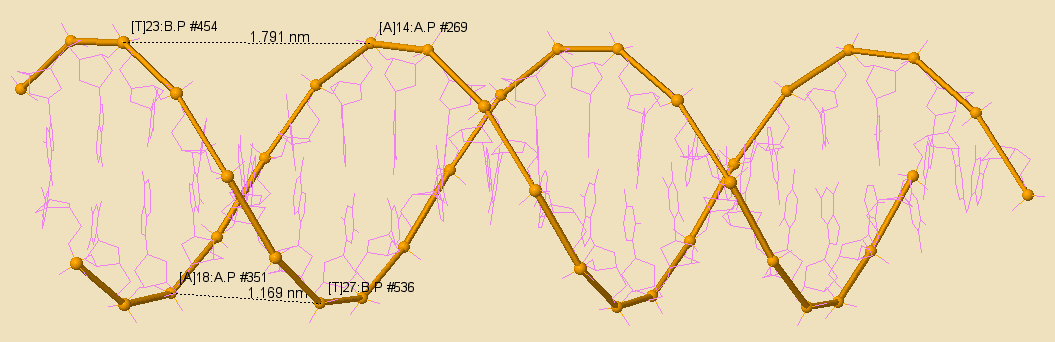
Рис. 5.2 Изучение большой и малой бороздок ДНК в B форме
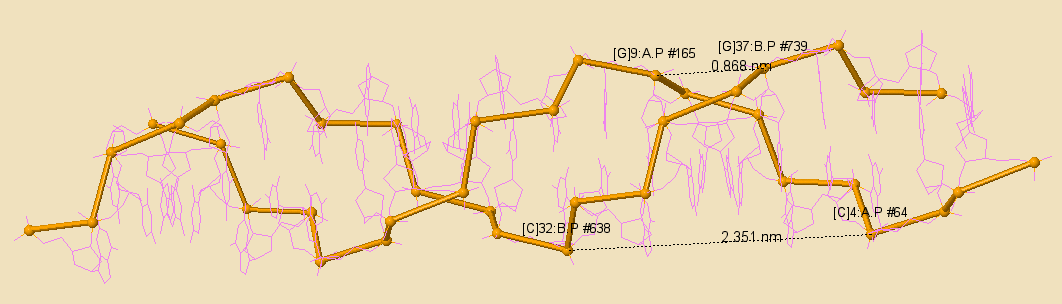
Рис. 5.3 Изучение большой и малой бороздок ДНК в Z форме
Таблица 1
| A-форма | B-форма | *Z-форма | |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (нм) | 2.803 | 3.091 | 4.350 |
| Число оснований на виток | 12 | 10 | 13 |
| Ширина большой бороздки (нм) | 1.697 | 1.791 | 2.351 |
| Ширина малой бороздки (нм) | 0.798 | 1.169 | 0.868 |
Затем мной были измерены торсионные углы выбранного ранее нуклеотида, а именно 31 тимина и сопоставлены со значениями из презентации . Результат моей работы представлен на рис. 6.1, 6.2 и в табл. 2.
Торсионные углы:
alpha(O3'-P-O5'-C5')
beta (P-O5'-C5'-C4')
gamma (O5'-C5'-C4'-C3')
delta (C5'-C4'-C3'-O3')
epsilon (C4'-C3'-O3-P')
xi (C3'-O3-P'-O5')
chi (C2-N1-C4'-O4')
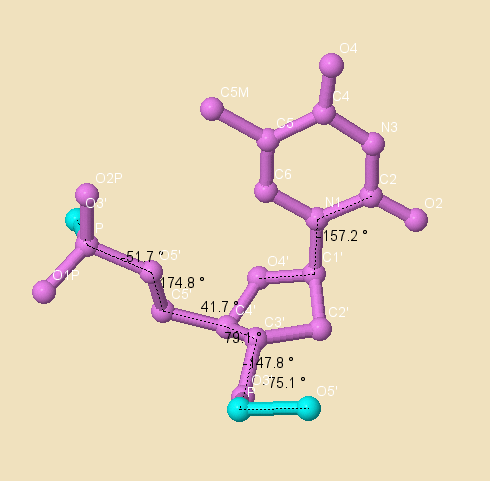
Рис. 6.1 Торсионные углы тимина в А форме
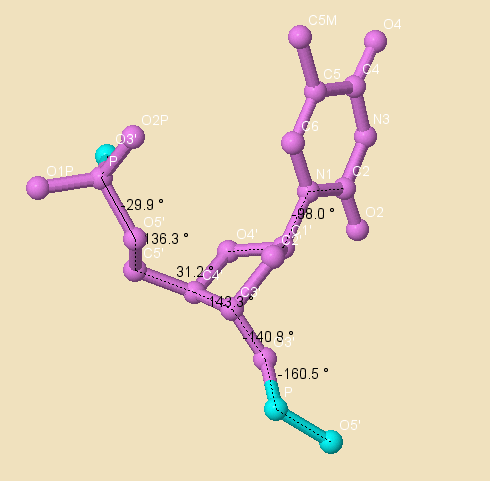
Рис. 6.2 Торсионные углы тимина в B форме
| alpha | beta | gamma | delta | epsilon | xi | chi | |
| A-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| A-форма (из презентации) | 62 | 173 | 52 | 88/3 | 178 | -50 | -160 |
| B-форма | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
| B-форма (из презентации) | 63 | 171 | 54 | 123/131 | 155 | -90 | -117 |
В следующем задании был применен пакет 3DNA, а именно PDB файлы были переведены в старый формат и проанализированы с помощью следующих комманд:
remediator --old ''1HDD.pdb'' > ''1HDD_old.pdb remediator --old ''2DLC.pdb'' > ''2DLC_old.pdb find_pair -t 1HDD_old.pdb stdout | analyze find_pair -t 2DLC_old.pdb stdout | analyze
В результате были получены выходные файлы, в том числе файлы 1HDD_old.out и 2DLC_old.out.
Из полученных файлов были выявлены значения торсионных углов для обоих файлов, а также с помощью программы LibreOffice Calc были вычислены средние значения для торсионных углов обоих цепей. Из полученных данных (см. рис. 7.1 и рис. 7.2) можно заключить, что исследуемые структуры не похожи ни на одну из ранее исследуемых, а также то, что значения торсионных углов очень вариабельны для каждого нуклеотида, из чего нельзя сделать вывод о какой-либо структуре.
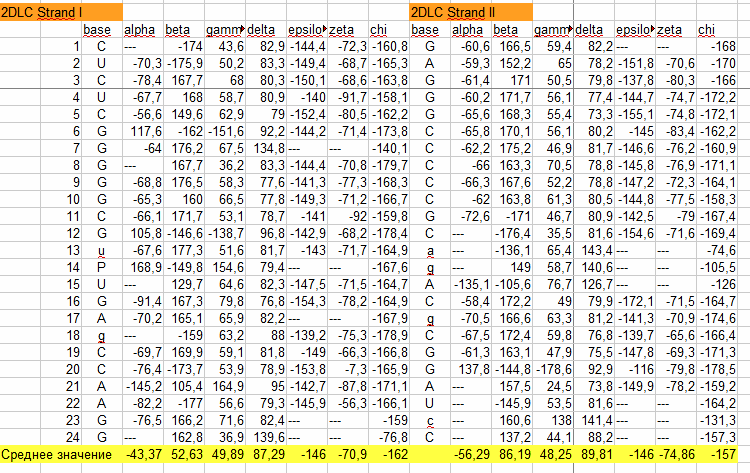
Рис. 7.1 Значения торсионных углов для 2DLC и средние значения.
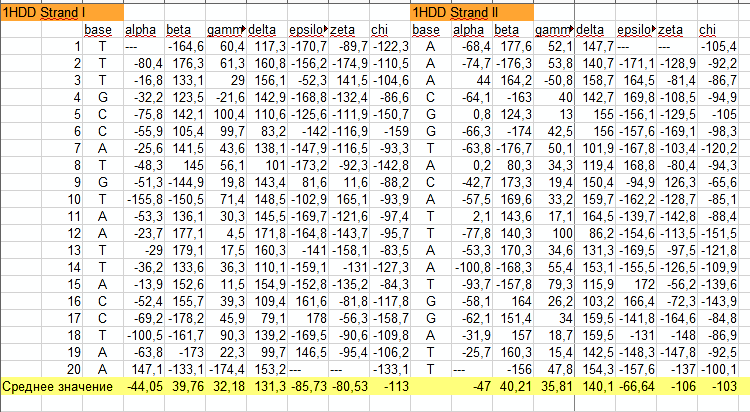
Рис. 7.2 Значения торсионных углов для 1HDD и средние значения.
В следующем задании было предложено изучить водородные связи тРНК. С помощью рис.8.1 из статьи "СТРОЕНИЕ ТРАНСПОРТНЫХ РНК И ИХ ФУНКЦИЯ НА ПЕРВОМ (ПРЕДРИБОСОМНОМ) ЭТАПЕ БИОСИНТЕЗА БЕЛКОВ" О.О.Фаворовой.
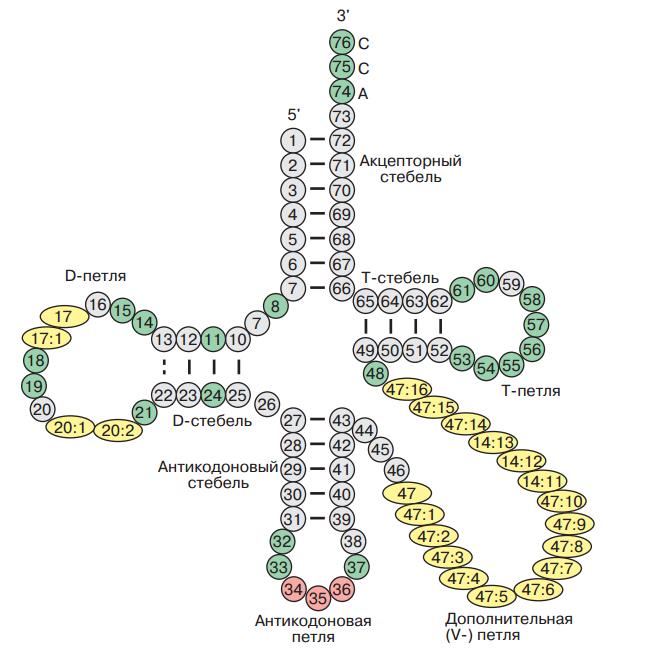
Рис.8.1 Рисунок, показывающий структуру тРНК из статьи Фаворовой О.О.
На следующей информации представлена часть выдачи find_pair и стебли тРНК выделены цветом. Выделение 4х стеблей в данной тРНК. Красный - акцепторный стебель, синий - T-стебель, желтый - антикодоновый стебель, зеленый - Д-стебель.
Strand I Strand II Helix
1 (0.007) Y:.501_:[..C]C-----G[..G]:.572_:Y (0.014) |
2 (0.009) Y:.502_:[..U]U-----A[..A]:.571_:Y (0.007) |
3 (0.006) Y:.503_:[..C]C-----G[..G]:.570_:Y (0.010) |
4 (0.009) Y:.504_:[..U]U-*---G[..G]:.569_:Y (0.008) |
5 (0.007) Y:.505_:[..C]C-----G[..G]:.568_:Y (0.008) |
6 (0.008) Y:.506_:[..G]G-----C[..C]:.567_:Y (0.012) |
7 (0.013) Y:.507_:[..G]Gx----C[..C]:.566_:Y (0.014) |
8 (0.010) Y:.549_:[..G]G-----C[..C]:.565_:Y (0.011) |
9 (0.009) Y:.550_:[..G]G-----C[..C]:.564_:Y (0.007) |
10 (0.008) Y:.551_:[..G]G-----C[..C]:.563_:Y (0.009) |
11 (0.008) Y:.552_:[..C]C-----G[..G]:.562_:Y (0.009) |
12 (0.009) Y:.553_:[..G]G----xC[..C]:.561_:Y (0.008) |
13 (0.010) Y:.554_:[5MU]u-**-xa[1MA]:.558_:Y (0.006) |
14 (0.039) Y:.555_:[PSU]Px**+xg[OMG]:.518_:Y (0.009) x
15 (0.011) Y:.542_:[..U]U-*---A[..A]:.528_:Y (0.009) |
16 (0.010) Y:.543_:[..G]G-----C[..C]:.527_:Y (0.007) |
17 (0.009) Y:.544_:[..A]Ax*---g[M2G]:.526_:Y (0.012) |
18 (0.012) Y:.510_:[2MG]g-----C[..C]:.525_:Y (0.007) |
19 (0.008) Y:.511_:[..C]C-----G[..G]:.524_:Y (0.012) |
20 (0.007) Y:.512_:[..C]C-----G[..G]:.523_:Y (0.010) |
21 (0.008) Y:.513_:[..A]A-*--xA[..A]:.522_:Y (0.007) |
22 (0.007) Y:.514_:[..A]A-**-xU[..U]:.508_:Y (0.009) |
23 (0.010) Y:.515_:[..G]Gx**+xc[5MC]:.548_:Y (0.006) x
24 (0.009) Y:.519_:[..G]G-----C[..C]:.556_:Y (0.008) +
В составе тРНК присутствует 8 пар азотистых оснований с неканоническим типом взаимодействий. Они отмечены звёздочками (4 U-G, 13 5MU-1MA, 14 PSU-0MG, 15 U-A, 17 A-2MG, 21 A-A, 22 A-U, 23 G-5MC).
Дополнительно в стабилизации структуры тРНК водородными связями участвуют пары 12 G-C, 13 5MU-1MA (подтверждение того, что эта пара взаимодействует неканонически вы можете найти на рис.8.2), 14 PSU-0MG, 17 A-2MG, 22 A-U, 23 G-5MC, 24 G-C.
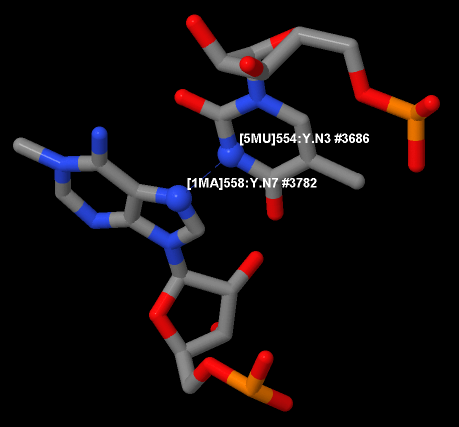
Рис.8.2 Неканоническое взаимодействие пары 5MU-1MA. Водородные связи отмечены синим пунктиром. Взаимодействующие атомы в шариковой модели
Далее в задании было предложено научиться находить возможные стекинг-взаимодействия в струкуре тРНК. Информация, полученная из файла 2DLC_old.out представлена ниже:
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 CU/AG 0.00( 0.00) 0.00( 0.00) 0.54( 0.00) 1.84( 1.76) 2.37( 1.76)
2 UC/GA 0.70( 0.00) 0.00( 0.00) 0.00( 0.00) 4.10( 2.88) 4.80( 2.88)
3 CU/GG 3.25( 1.18) 0.00( 0.00) 0.00( 0.00) 3.88( 2.45) 7.13( 3.63)
4 UC/GG 0.30( 0.00) 0.00( 0.00) 0.21( 0.00) 2.07( 0.63) 2.58( 0.63)
5 CG/CG 0.02( 0.00) 0.00( 0.00) 4.74( 1.89) 0.00( 0.00) 4.76( 1.89)
6 GG/CC 3.33( 1.80) 0.00( 0.00) 0.18( 0.00) 0.17( 0.00) 3.69( 1.80)
7 GG/CC 1.99( 0.56) 0.00( 0.00) 0.99( 0.00) 0.00( 0.00) 2.98( 0.56)
8 GG/CC 2.69( 1.16) 0.00( 0.00) 0.74( 0.00) 0.16( 0.04) 3.59( 1.20)
9 GG/CC 3.83( 2.61) 0.00( 0.00) 0.18( 0.00) 0.58( 0.00) 4.59( 2.61)
10 GC/GC 6.99( 4.11) 0.00( 0.00) 0.00( 0.00) 5.96( 2.88) 12.95( 6.99)
11 CG/CG 0.00( 0.00) 0.00( 0.00) 4.73( 1.85) 0.00( 0.00) 4.73( 1.85)
12 Gu/aC 7.94( 2.62) 0.00( 0.00) 0.00( 0.00) 3.98( 1.07) 11.92( 3.69)
13 uP/ga 5.54( 1.57) 0.00( 0.00) 0.00( 0.00) 8.47( 3.54) 14.01( 5.11)
14 PU/Ag 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
15 UG/CA 0.03( 0.00) 0.00( 0.00) 3.13( 0.52) 0.00( 0.00) 3.16( 0.52)
16 GA/gC 1.49( 0.21) 0.00( 0.00) 0.00( 0.00) 3.72( 1.58) 5.21( 1.79)
17 Ag/Cg 0.00( 0.00) 0.00( 0.00) 0.10( 0.00) 0.93( 0.44) 1.03( 0.44)
18 gC/GC 5.56( 2.65) 0.00( 0.00) 0.00( 0.00) 6.09( 3.06) 11.66( 5.71)
19 CC/GG 0.00( 0.00) 0.00( 0.00) 0.98( 0.00) 2.71( 1.19) 3.69( 1.19)
20 CA/AG 2.37( 0.38) 0.00( 0.00) 0.00( 0.00) 2.84( 0.82) 5.20( 1.20)
21 AA/UA 0.00( 0.00) 3.18( 0.47) 0.25( 0.04) 0.00( 0.00) 3.42( 0.51)
22 AG/cU 2.74( 0.69) 0.00( 0.00) 0.01( 0.00) 0.00( 0.00) 2.75( 0.69)
23 GG/Cc 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Из полученной информации можно заключить, что наибольшее перекрывание, а следовательно и наибольшее стекинг взаимодействие, наблюдается для пар 13 uP/ga (u - это 5-метилуридин, a - это 6-гидро-1-метиладенозин, P - это псевдоуридин, а g - это О2'-метилгуанозин) . Для того, чтобы получить изображение, необходимо выполнить команды:
ex_str -13 stacking.pdb step13.pdb
stack2img -cdolt step13.pdb step13.ps
Полученный файл step13.ps был визуализирован с помощью онлайн сервиса http://view.samurajdata.se/ (Спасибо за идею Оле Васюткиной). См. рис.9.1.
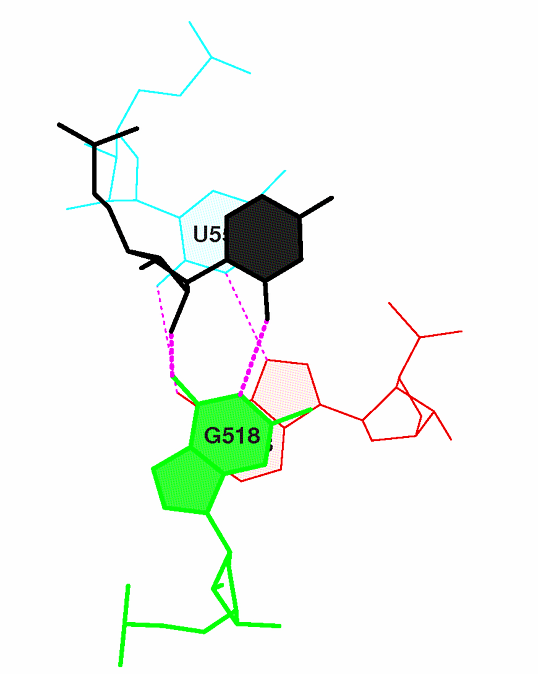
Рис. 9.1. Визуализация стэкинг-взаимодействия для пары с наибольшим перекрыванием с помощью http://view.samurajdata.se/.
Для визуализации стекинг взаимодействия с помощью Jmol был создан отдельный .pdb файл из исходного с координатами только участвующих в стэкинг-взаимодействии атомами.
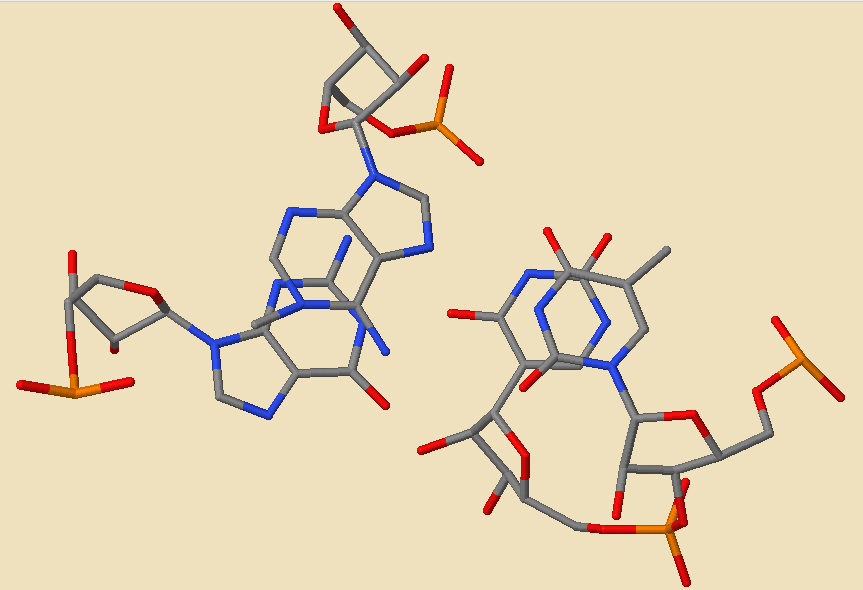
Рис. 9.2. Визуализация стэкинг-взаимодействия для пары с наибольшим перекрыванием с помощью Jmol.