


Для поиска инвертированных повторов использовалась программа einverted из пакета EMBOSS. При значении "Minimum score threshold", равном 50 (по умолчанию), ничего найдено не было. Порог был уменьшен до 15, обнаружилось 2 участка.
einverted -sequence 2DLC.fasta
< a href = "sequence.inv">Здесь вы можете найти файл sequence.inv, результат работы einverted, в котором показаны следующие инвертированные повторы:
SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps
27 caaga 31
|||||
43 gttct 39
SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps
49 gggcg 53
|||||
65 cccgc 61
С помощью онлайн версии программы mfold было построено изображение предположительной вторичной стурктуры тРНК. Программа была запущена несколько раз до получения структуры, похожей на тРНК. При этом изменялся параметр P (Percent suboptimality) от 5% по умолччанию до 15% (на сколько процентов вычисленная энергия предсказанной структуры может отличаться от оптимальной) При P=15% было получено пять стурктур, из них была выбрана одна наиболее подходящая. Визуализированный результат вы можете увидеть на рис.1
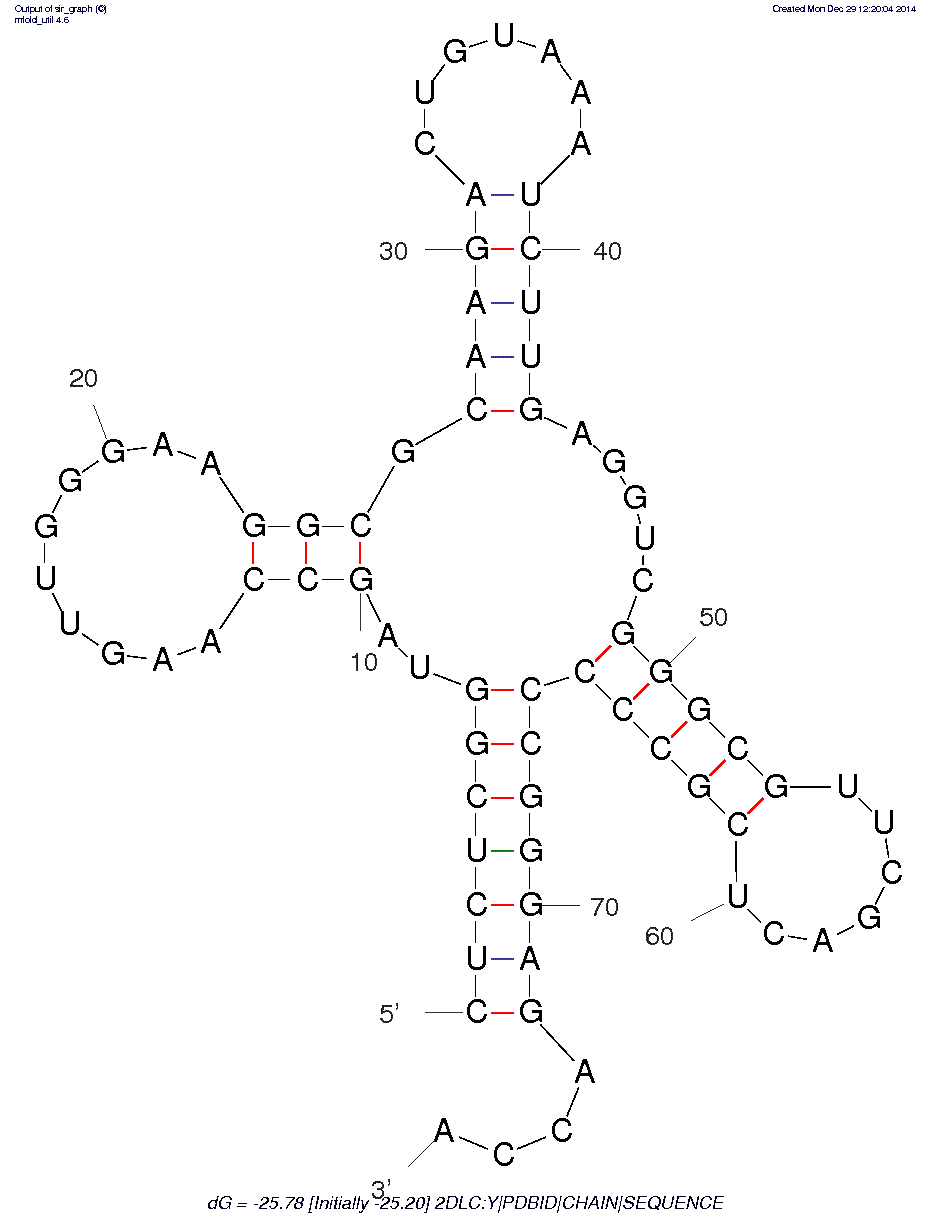
Рис.1
Таблица 1.
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-501-507-3' 5'-566-572-3' всего 7 пар | не найден | 7 пар из 7 реальных |
| D-стебель | 5'-510-513-3' 5'-522-525-3' всего 4 пары | не найден | 3 пары из 3-4 реальных (по статье Фаворовой) |
| T-стебель | 5'-549-553-3' 5'-561-565-3' всего 5 пар | 5 пар из 5 реальных | 5 пар из 5 реальных |
| Антикодоновый стебель | 5'-526-528-3' 5'-542-544-3' всего 5 пар | 5 пар из 5 реальных | 5 пар из 5 реальных |
| Общее число канонических пар нуклеотидов | 21 | 10 | 20 |
По результатам таблицы можно заключить, что с помощью алгоритма Зукера можно получить результат близкий к реальному. Предсказание с помощью инвертированных повторов (программа einverted) не дало хороших результатов.
Поиск ДНК-белковых контактов в заданной структуре
Было предложено создать написать скрипт-файл с определением множеств атомов кислорода 2'-дезоксирибозы (set1), атомов кислорода в остатке фосфорной кислоты (set2), множество атомов азота в азотистых основаниях (set3). Файл с результатом А так же скрипт-файл, вызов которого в JMol даст последовательное изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов set1 (окрашиваются в оранжевый), затем set2 (окрашиваются в фиолетовый) и set3 (окрашиваются в зеленый). Файл с результатом
Поиск ДНК-белковых контактов производился в помощью программы Jmol.
Будем считать полярными атомы кислорода и азота, а неполярными атомы углерода, фосфора и серы. Для выполнения задания и заполнения таблицы 2 определим следующие множества атомов:
полярные атомы дезоксирибозы
неполярные атомы дезоксирибозы
полярные атомы остатков форфорной кислоты
неполярные атомы остатков форфорной кислоты
полярные атомы остатков азотистых оснований со стороны большой бороздки
полярные атомы остатков азотистых оснований со стороны большой бороздки
полярные атомы остатков азотистых оснований со стороны малой бороздки
полярные атомы остатков азотистых оснований со стороны малой бороздки
полярные атомы белка
неполярные атомы белка
Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5 ангстрем. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5 ангстрем. На основании вышеперечисленных утверждений был написан скрипт, последовательно показывающий контакты разных групп атомов нуклеиновой кислоты с белком. Результаты приведены в таблице 2.
Таблица 2. Количество контактов ДНК и белка.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 4 | 16 | 20 |
| остатками фосфорной кислоты | 7 | 10 | 17 |
| остатками азотистых оснований со стороны большой бороздки | 2 | 13 | 15 |
| остатками азотистых оснований со стороны малой бороздки | 1 | 1 | 2 |
Следующим заданием было получить схему ДНК-белковых контактов с помощью программы nucplot. Это было выполнено при помощи команд:
remediator --old ''1HDD.pdb'' > ''1HDD_old.pdb
nucplot 1HDD_old.pdb
Полученный на выходе файл nucplot.ps вы можете скачать здесь. Визуализированная схема контактов представлена на рис. 2.1-3
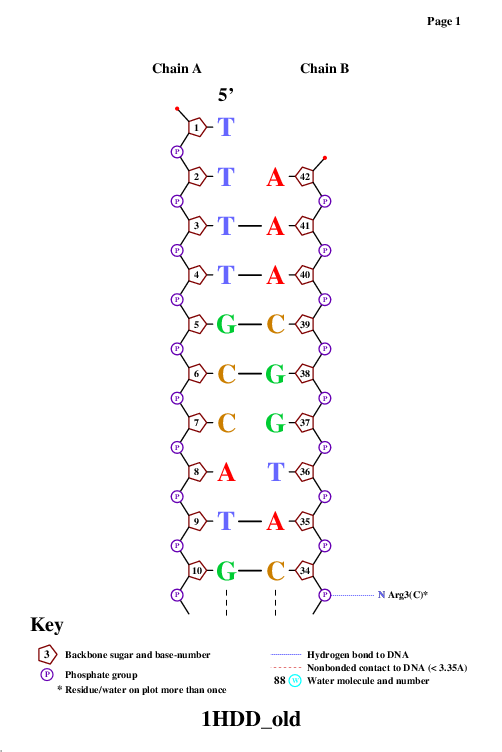
Рис.2.1
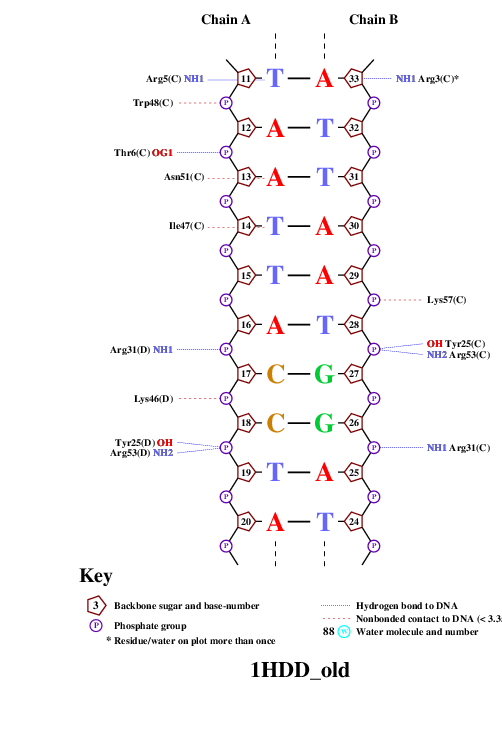
Рис.2.2
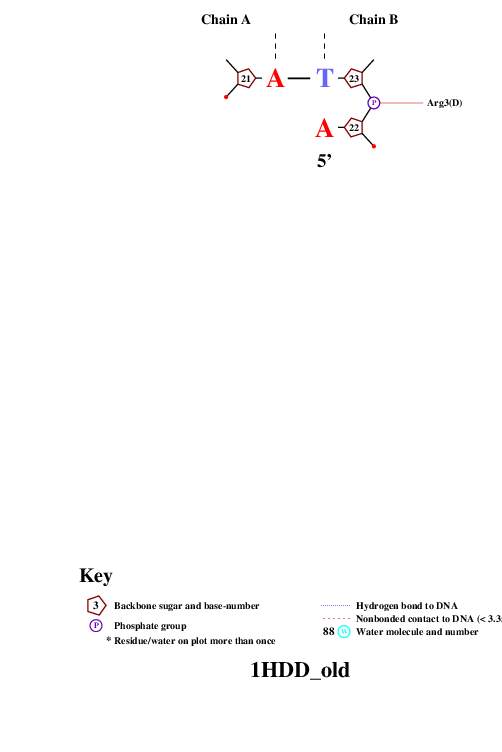
Рис.2.3
Из рис.2.1-3 видно, что Arg3 цепи C образует два контакта с ДНК, что является наибольшим количеством контактов. Наиболее важными для распознавания последовательности ДНК являются те нуклеотиды, образующие водородные связи с азотисттыми основаниями, такие как Arg3(C), Arg5(C). На рис.3.1-2 вы можете наблюдать визуальзацию данных взаимоействий в Jmol.
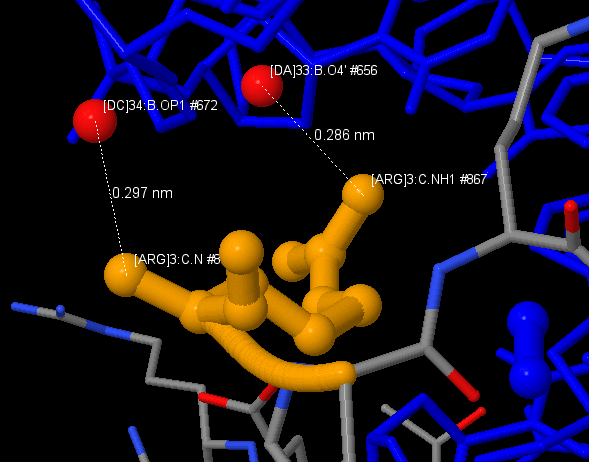
Рис. 3.1
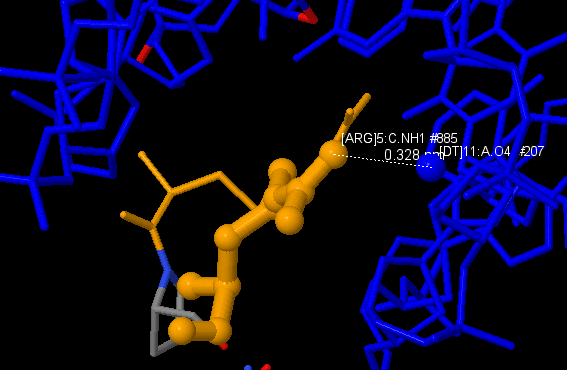
Рис. 3.2