


В данном практикуме было необходимо проаннотировать фрагмент (контиг) бактериального микробиома сумчатого животного - Болотного валлаби (Wallabia bicolor).
Для начала я воспользовалась программой getorf пакета EMBOSS со следующими параметрами: бактериальный генетический код, рамка считывания не менее 180 нуклеотидов, трансляция между старт и стоп кодонами (-find 1) .
getorf -sequence contig.fasta -outseq Wallabi_bicolor.orf -table 11 -minsize 180 -find 1
Нашлось 14 рамок считывания. Их вы можете посмтореть здесь.
Далее я запустила blastp на сервере NCBI . Выбрала базу данных UniProtKB/Swiss-Prot в параметрах поиска, а также выбрала в поле Organism: bacteria (taxid: 2). Последнее показалось мне оправданным, так как исходно исследуется микробиом желудка Wallabi.
Результат, выданный программой, показался мне очень странным. Ни одна из находок не имела даже близкого к хорошему evalue, ни нужного покрытия, хотя была найдена последовательность генома как раз симбиотической бактерии кишечника. Предполагаю, что это связано с тем, что я пыталась проаннотировать гены, подав на вход сразу все рамки считывания.
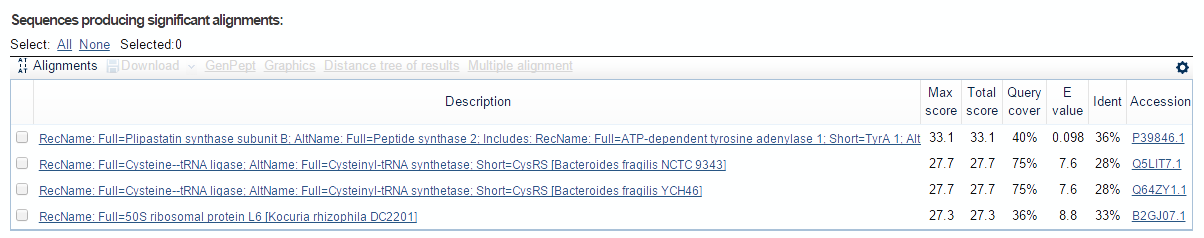
Рис. 1 Результат первого запуска blast. Непонятные значения.
После этого, я запустила BLAST для каждой найденной рамки в отдельности. Значительные соответствия были найденны в 5, 7, 13 и 14 рамках считывания. Информацию о полученных результатах (координатах найденных рамок считывания, возможных генов, закодированных вв последовательности) вы можете найти в таблице 1.
| 2279 | 2572 | 98 | + | Чувствительный к меди ререссор транскрипции. Стоит отметить, что найденные соответствия принадлежат к суперсемейству CsoR-like_DUF156, что было подчеркнуто в результатах blastp. Это суперсемейство включает в себя транскрипционные регулятороы, которые реагируют на стресс-включая Cu (I), Ni (I), сульфит, и формальдегида. Содержатся как раз в симбиотитических кишечных бактериях. |
| 2588 | 2914 | 109 | + | Cu зависимая АТФаза типа P. Стоит отметить, что найденные белки принаджлежат к HMA cуперсемейтву. Белки этого семейства содержат консервативный домен, ответственный за транспортировку и детоксикацию тяжелых металлов. Находки были обнаружены в золотистом стафилококке, Bacilis Subtilis, Helicobacter Pylori. |
| 1625 | 846 | 260 | - | Найденные соответсвия принадлежат к суперсемейству prsW протеаз. Это распространенное семейство мембранных белков, которые вызывают стресс клеточной оболочки, при контакте с чужеродными пептидами. |
| 47 | 826 | 260 | - | Были найдены белки суперсемейства дегалогеназ (HAD_like superfamily). |
Далее мной была запущена программа GeneMark (эвристический алгоритм 1999), которая тоже используется для предсказания генов в контиге. На вход программе был подан исходный контиг , который использовался ранее для предсказания рамок считывания. График кодирующего потенциала в формате pdf вы можете найти здесь Фрагмент графика из файла вы можете найти на рисунке 2.
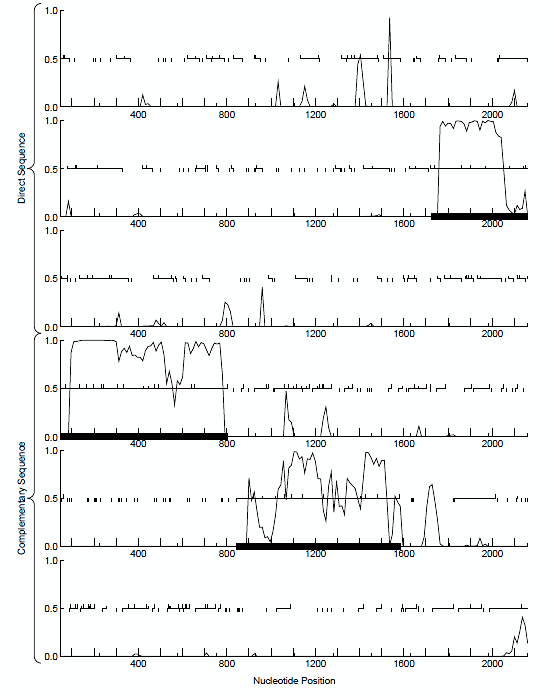
Рис. 2. Фрагмент отчета работы программы GeneMark.
Также из одного из выходных файлов можно узнать точные координаты предсказанных генов.
| Номер предсказания | Цепь | Левый конец | Правый конец | Длина гена | |
| 1 | - | 44 | 802 | 759 | |
| 2 | - | 843 | 1583 | 741 | |
| 3 | + | 1724 | 2200 | 477 | |
| 4 | + | 2279 | 2575 | 297 | |
| 5 | + | 2594 | 2914 | 321 |
Как видно из полученных данных, предсказанные GeneMark и getorf довольно хорошо соответствуют. Различие состоит только в небольшом сдвиге координат предполагаемых генов и наличия в результатах работы GeneMark предполагаемого гена (номер 3), не обнаружевшего соответствий при работе blastp.