


Построение филогенетического дерева восьми выбранных бактерий отдела Firmicutes проводилось на основе нуклеотидных последовательностей РНК малой субъединицы рибосомы (16S rRNA).
Последовательности 16S рибосомальной РНК каждой бактерии были скачаны из базы полных геномов NCBI (для каждой бактерии выбиралась одна из последовательностей 16S РНК, имеющихся в файле с расширением .frn). Все последовательности были записаны в файл, в названиях последовательностей были оставлены только соответствующие мнемоники бактерий.
Далее последовательности были импортированы в MEGA, выравнены с помощью ClustalW выравнивание сохранено и открыто для анализа (Analyze). Дерево было реконструировано методом Maximum likelihood. Графическое изображение дерева можно увидеть на рис. 1.
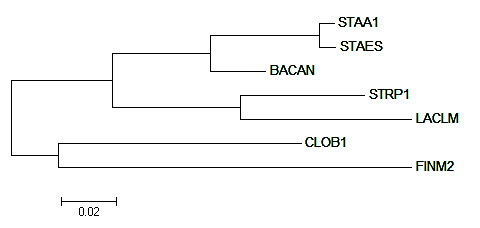
Рис. 1. Филогенетическое дерево нескольких видов бактерий отдела Firmicutes, построенное на основе выравнивания нуклеотидных последовательностей РНК малой субъединицы рибосомы (16S rRNA). Дерево построено методом Maximum likelihood.
Полученное дерево полность совпадает с предыдущим с указанным правильным деревом, построенным на основе филогении (см. предыдущий практикум), несмотря на то, что выравнивания нуклеотидных последовательностей сами по себе менее достоверны, чем выравнивания соответствующих белков (из-за того что нуклеотидные последовательности состоят только из 4-ёх букв, вероятность случайного совпадения позиций намного больше, чем для белковых последовательностей). Тем не менее, выравнивание, построенное на основе 16s РНК получилось абсолютно точным, что доказывает применимость построения филогенетических деревьев на основе 16s РНК.
Построение и анализ дерева, содержащего паралоги
Проводился поиск гомологов белка CLPX_BACSU среди белков семии выбранных бактерий.
Протеомы бактерий были скачаны из директории P:\y13\term4\Proteomes и объединены в общий файл proteomes.fasta. cat bacan.fasta >> proteomes.fasta cat staa1.fasta >> proteomes.fasta cat staes.fasta >> proteomes.fasta cat strp1.fasta >> proteomes.fasta cat laclm.fasta >> proteomes.fasta cat clob1.fasta >> proteomes.fasta cat finm2.fasta >> proteomes.fasta Файл с последовательностью исследуемого белка был скачан из базы данных Swissprot.
Поиск гомологов проводился с помощью blastp:
makeblastdb -in proteomes.fasta -dbtype prot
blastp -query CLPX_BACSU.fasta -db proteomes.fasta -evalue 0.001 -outfmt 7 -out blastp.out
На основе полученного файла, содержащего информацию о гомологах, был создан файл-список
cat blastp.out | egrep -v '#' | awk ' {print $2}' | awk ' BEGIN {FS = "|"} {print "fasta::proteomes.fasta:" $3}' > homol_id.bs
Затем из этого списка были удалены "дублирующиеся" белки (могут появиться из-за того, что один белок может иметь несколько участков совпадений с исходным). Получившийся список homol_id.bs был подан на вход программе seqret:
seqret @homol_id.bs
Таким образом был получен файл homologs.fasta, содержащий аминокислотные последовательности гомологов белка CLPX_BACSU, которые были выравнены с помощью ClustalW. На основании выравнивания с помощью MEGA построено дерево реконструировано методом Maximum likelihood. Графическое изображение получившегося дерева можно увидеть на рис. 2.
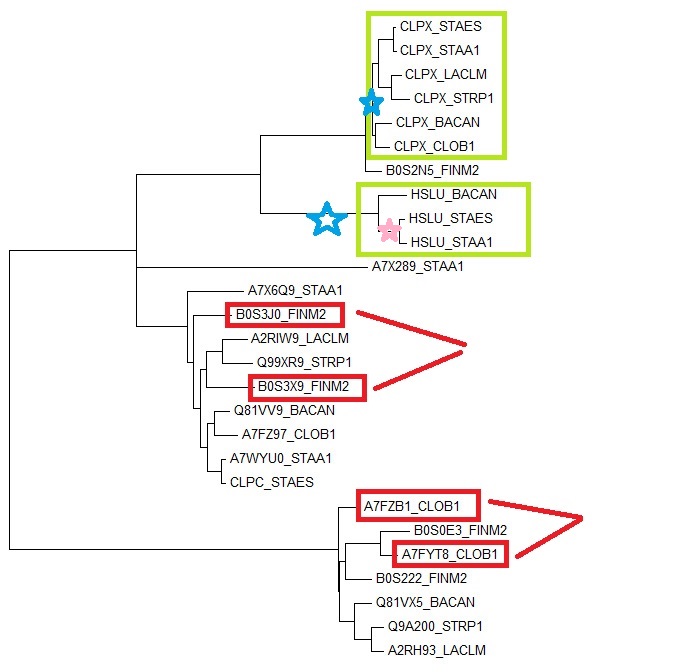
Рис. 2. Филогенетическое дерево гомологов белка CLPX_BACSU среди белков восьми бактерий отдела Firmicutes. Построено методом Maximum likelihood. Зелёными рамочками выделены группы попарно ортологичных белков, красными - паралоги. Зелёными звёздочками помечены примеры разделения путей эволюции белков в результате видообразования, красной звёздочкой - пример дупликации гена.
Два гомологичных белка будем называть ортологами, если они: а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. Два гомологичных белка из одного организма будем называть паралогами.
На рис. 2 отмечены две группы попарно ортологичных белков (в зелёных рамочках), две пары паралогов (выделены красными рамочками), пример дупликации гена (розовая звёздочка) и два примера разделения путей эволюции белков в результате видообразования (синие звёздочки).