


Целью данного задания было проверить, являются ли члены разных ортологических рядов KEGG гомологичными белками.
Для исследования в рамках данной работы была выбрана реакция с EC-номером 3.1.2.21 из биохимического пути cинтеза жирных кислот, карту которого можно увидеть здесь, ее изображение приведено на рис. 1
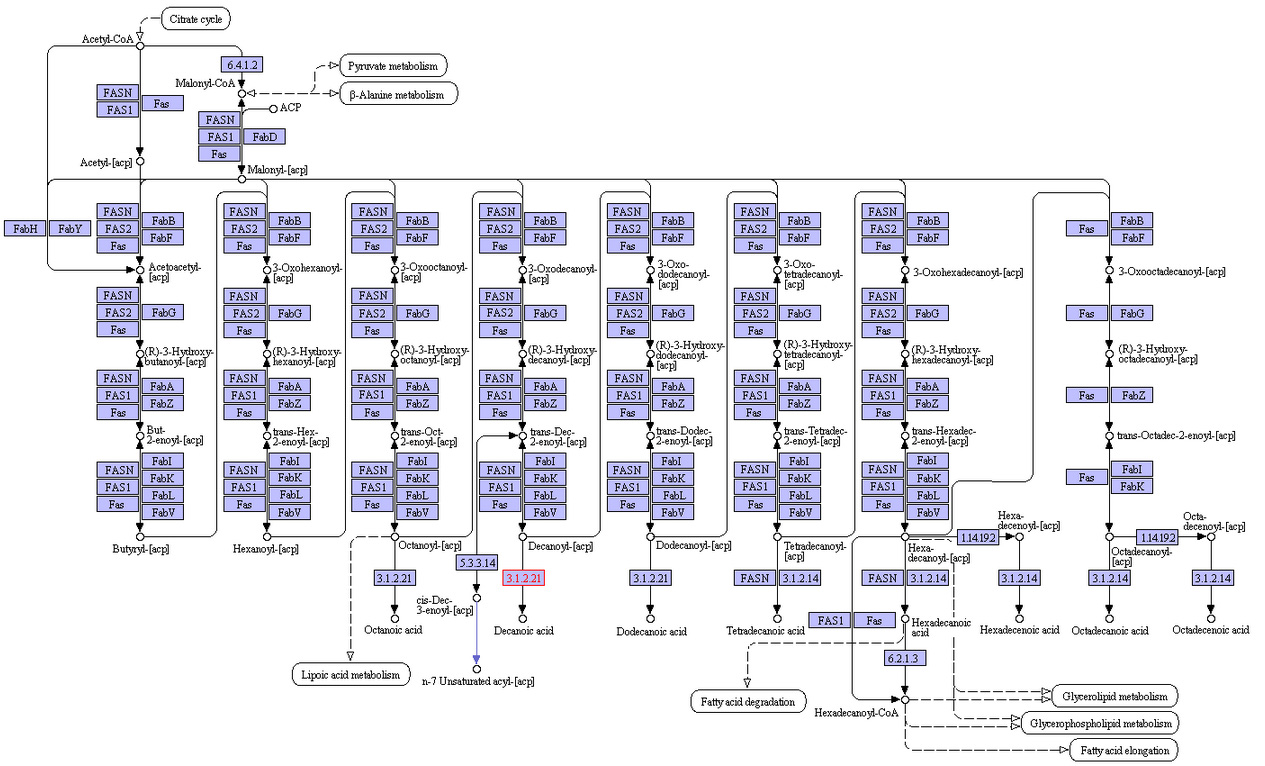
Рис.1. Карта метаболического пути синтеза жирных кислот из базы данных KEGG. Выбранная реакция покрашена в красный цвет.
Изображение реакции приведено на рис.2. Данную реакцию катализируют два ортологических ряда белков - K01071 гидролазы ацила средней цепи (medium-chain acyl-[acyl-carrier-protein] hydrolase), K10781 тиоэстеразы жирных кислот (fatty acyl-ACP thioesterase B).
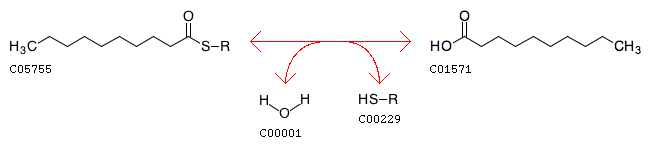
Рис.2 Схема рассматриваемой реакции
Далее были получены идентификаторы белков каждого ряда из таблицы идентификаторов (чтобы открыть эту таблицу надо нажать на кнопку "Uniprot" на странице с описанием нужного ряда). Список идентификаторов был подан на вход сервису "Retrieve/ID mapping" базы данных Uniprot. В результате были получены файлы с fasta-последовательностями нужных белков. Названия последовательностей были отредактированы с помощью скрипта Python, все последовательности были объединены в общий файл и выравнены с помощью Muscle (muscle -in all_orthology.fasta -out all_orthology_aligned.fasta). Файл с выравниванием вы можете скачать здесь.
Полученное выравнивание было открыто в JalView и покрашено раскраской Clustalx. Белки, сильно отличающиеся по составу и не включающие большинство доменов были исключены.Изображение выравнивания приведено на рис.3. Проект JalView align_clustalx.jvp

Рис. 3. Выравнивание белков из ортологических рядов
Белки, сильно отличающиеся по составу и не включающие большинство доменов были исключены. С помощью программы MEGA методом Neighbour-Joining с 100 бустреп-репликами была осуществлена попытка построения филогенетического дерева выбранных белков. К сожалению, построить дерево программе не удалось, MEGA6 выдавала ошибку на рис.4. Так же не удалось построить дерево при выравнвании ClustalW, встроенным в MEGA6, а так же ни одним из имеющихся филогенетических тестов, встроенных в MEGA6. (Maximum likelihood и Minimum evulution)
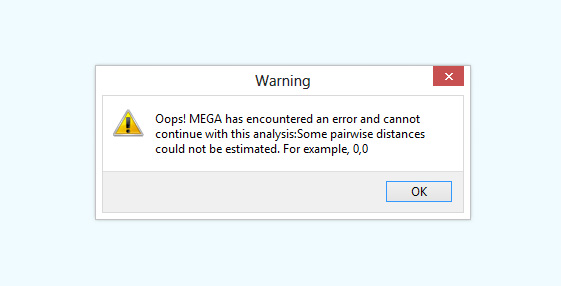
Рис. 4 Ошибка MEGA
Так как подходящие реакции не были мной найдены в метаболическом пути жирных кислот, я решила проделать то же самое для пентозофосфатного пути (Pentose phosphate pathway). Выбранная реакция с EC-номером 1.2.99.8. Схема пути приведена на рис. 5. Схема реакции на рис 6. Полученное выравнивание (проект Jalview) на рис. 7
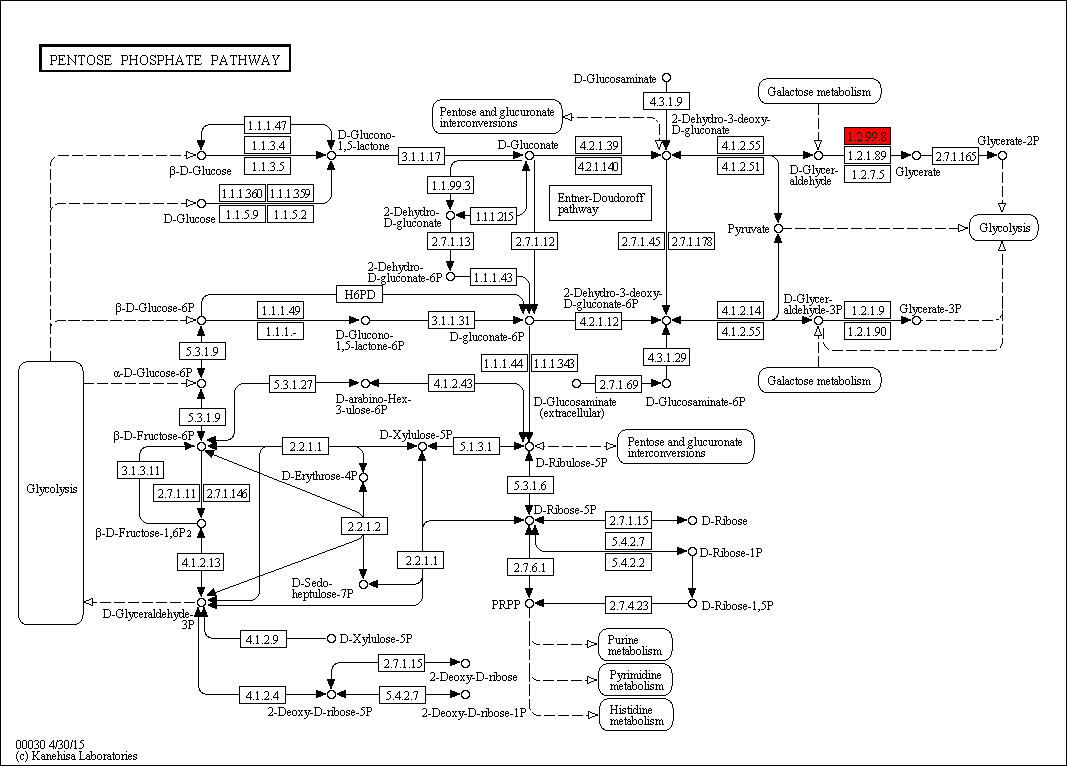
Рис. 5 Карта пентозофосфатного пути
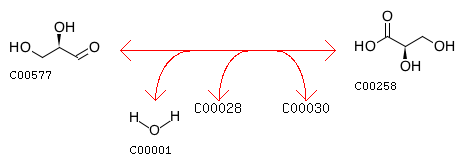
Рис.6 Схема реакции. Ракцию катализируют: K18020 (большая субъединица глицеральдегид дегидрогеназы; 20 белков Uniprot), K18021 (средняя субъединица глицеральдегид дегидрогеназы; 19 белков) и K18022 (малая субъединица глицеральдегид дегидрогеназы; 18 белков).
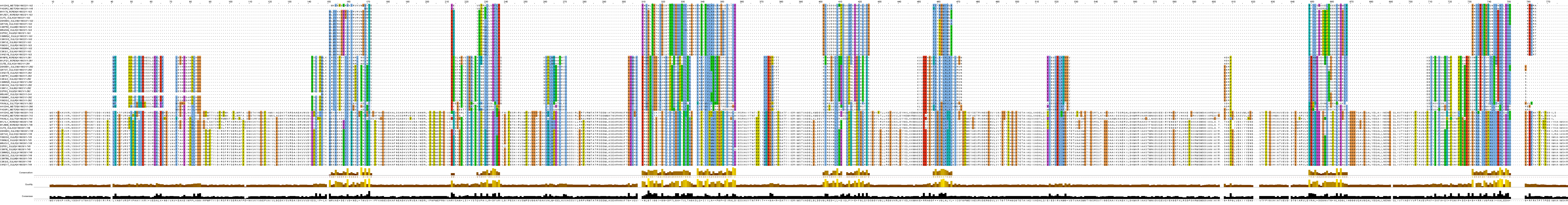
Рис. 7 Выравнивание последовательностей белков ортологических рядов
На основе этих характеристик построенного дерева нельзя сделать вывод о гомологичности всех исследуемых белков, поэтому построение дерева было бы бессмысленным, так как может дать какую-то информацию для белков только с общим происхождением. Исходя из выравнивания можно заключить, что хорошо выровнены белки внутри ортологического ряда, тогда как выравнивания каждого из ортологических рядов не похожи друг на друга, к тому же, длина белков, входящих в разные ортологические ряды, сильно различается. Их длины соответствуют названиям - большой малой и средней субъединицы глицеральдегид дегидрогеназы, принадлежность ортологических рядов к трём субъединицам одного комплекса, также вероятнее всего говорит о негомологичности белков.