


В данном практикуме было построено изображение электронной плотности (ЭП) вокруг полипептидной цепи и 3 отлельных аминокислотных остатков. Для работы я выбрала белок термостабильную алкогольдегидрогеназу из Pyrobaculum aerophilum, изучением которого я занималась в прошлых семестрах, с идентификатором PDB: 4JBI, разрешение структуры: 2.35 Е. Данные о нем удовлетворяют всем критериям выбора объекта изучения в этом семестре. С сервера EDS была скачана карта электронной плотности белка в формате по умолчанию (O и 2mFo-DFc).
ЭП для цепи J алькогольдегидрогеназы показана на рис. 1. Боковые цепи на рисунке не изображены. Для визуализации ЭП в PyMOL использовались следующие команды:
load 4JBI.pdb load 4jbi.omap, 4jbi_map select not (chain J) hide lines, sele hide (resn HOH) isomesh new_map, 4jbi_map, 1.5, backbone, 2.5, carve=2.5 zoom (chain J) bg_color wheat set mesh_color, grey50
Рис.1. Изображение ЭП вокруг полипептидной цепи
Здесь:
new_map: имя нового объекта, соответствующего изображению поверхности
3ntu_map: имя объекта, соответствующего электронной плотности
1.5: уровень контура - значение электронной плотности, для которого строится поверхность
backbone: полипептидный остов в качетстве множества атомов, электронную плотности вокруг которого надо изобразить,
carve=2.5: радиус вокруг каждого атома, для которого изобразить плотность
Построим изображение ЭП для Val 305, Tyr 306, Pro 307 цепи (см. рис. 2) для значений порога ЭП 0.5, 1.5 и 2.0.
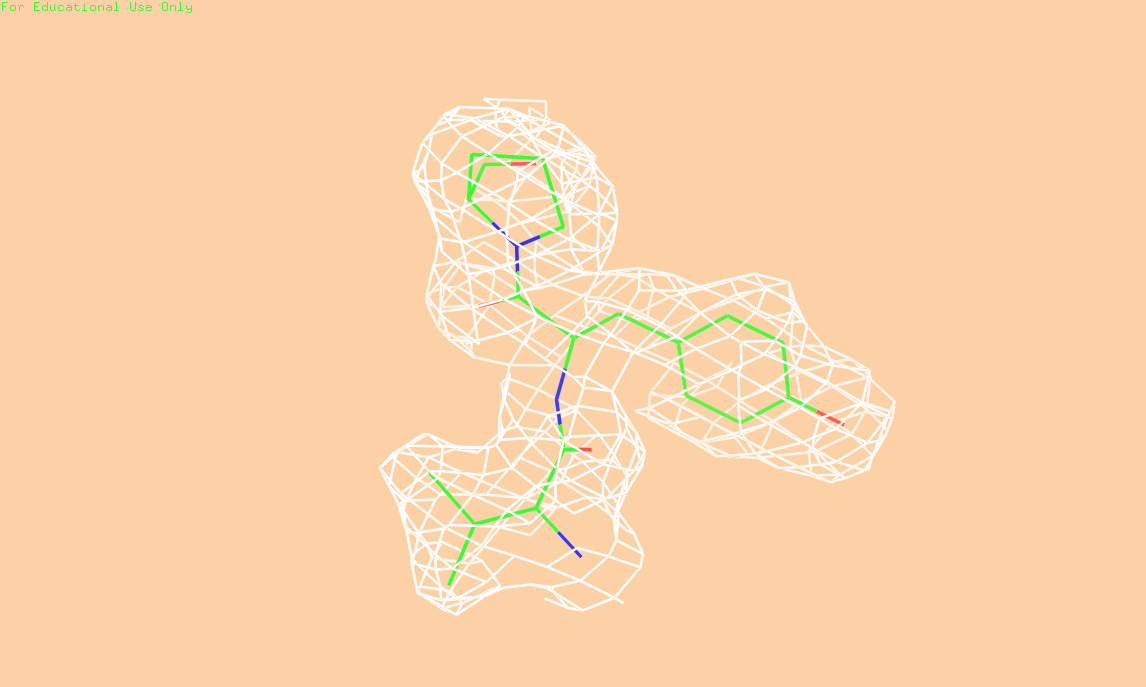
Рис. 2 (а) Изображение ЭП вокруг трех аминокислотных остатков. Порог 0.5
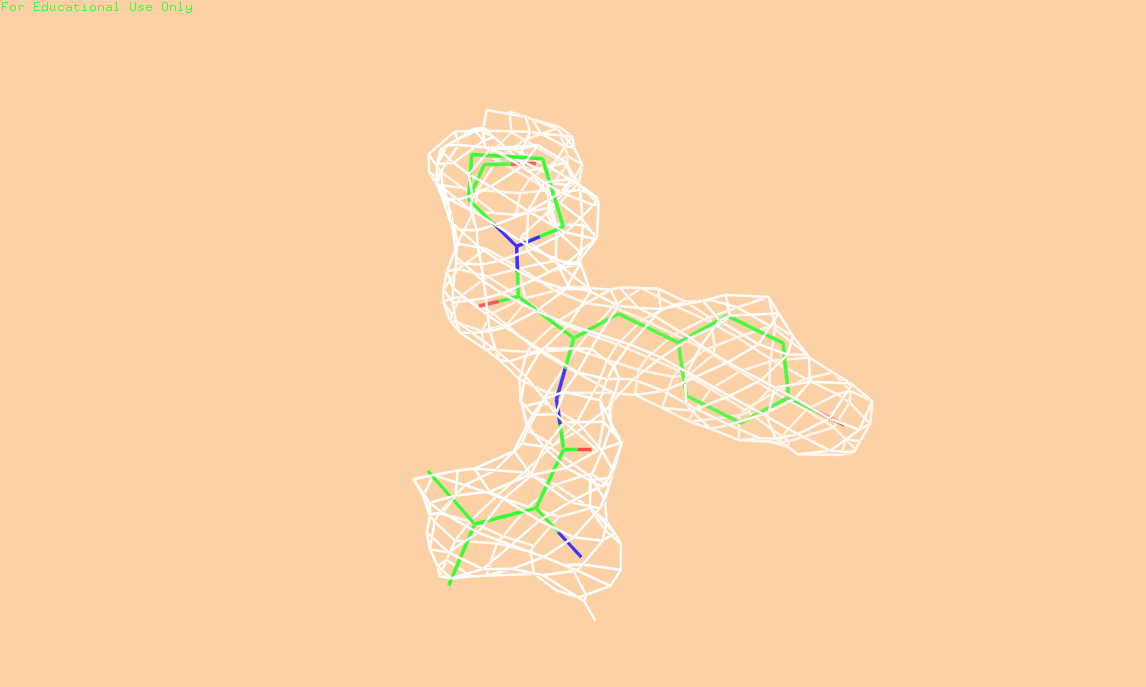
Рис. 2 (б) Изображение ЭП вокруг трех аминокислотных остатков. Порог 1.5
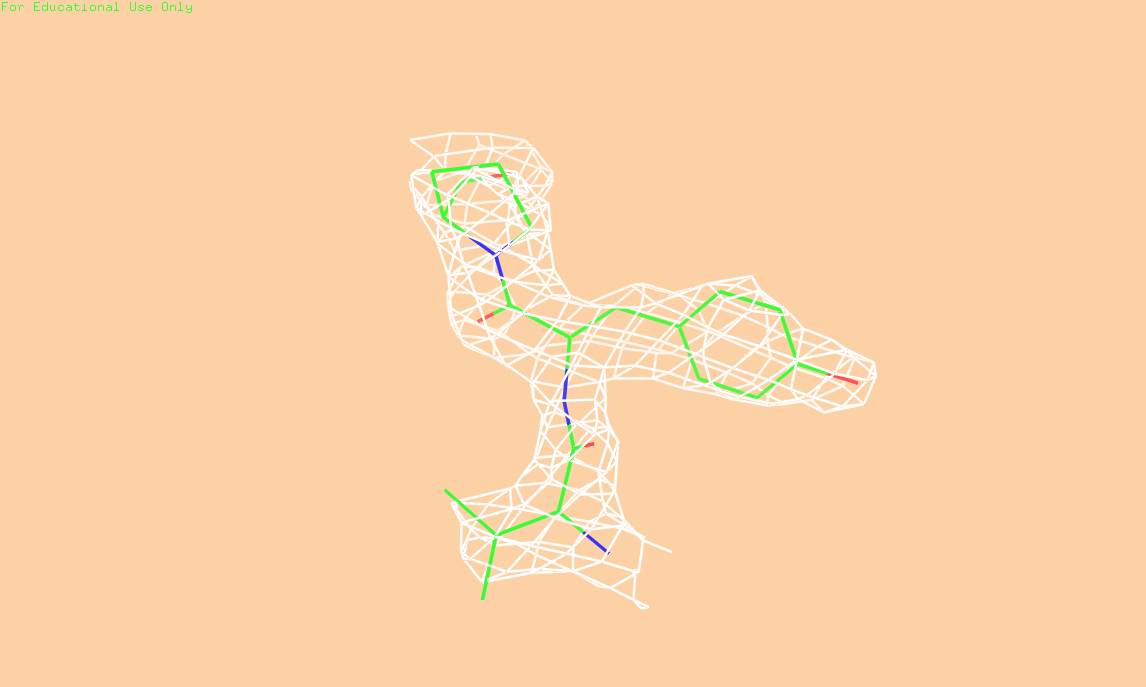
Рис. 2 (в) Изображение ЭП вокруг трех аминокислотных остатков. Порог 2.0
Исходя из полученных изображений электронной плотности можно заключить, что: разрешение структуры 4JBI не позволяет наблюдать отдельные атомы на изображении карты электронной плотности, однако оно достаточно для того, чтобы был виден ход полипептидной цепи и угадываелось положение боковых групп аминокислот.