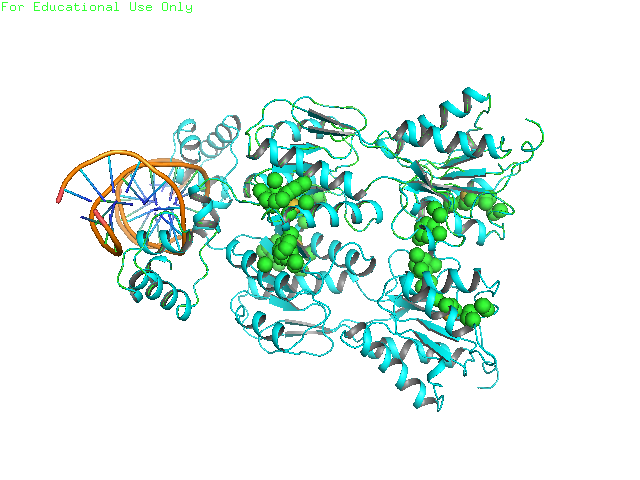
Для работы над этим практикумом был выбран пуриновый репрессор 1PNR, для который в файле PDB записана его ассиметрическая единица. В природе же он является димером, поэтому с помощью команды PyMol “symexp sym, 1PNR, 1PNR, 2” была воссоздана его биологическая единица. Изображение этого белка приведено на рис. 1.
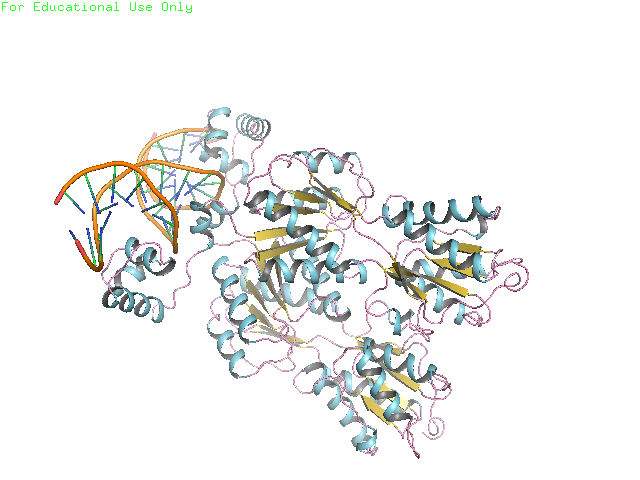
Рис. 1 Рис.1. Комплекс гомодимера пуринового репрессора с ДНК 1PNR Мономеры показаны разными цветами.
Для комплекса димера 1PNR было необходимо создать изображения:
а) поверхности контакта мономера белка с симметричным мономером на фоне остовной (ribbon) модели мономера - см. Рис 2
б) поверхности контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка, вовлечённой в контакт см. Рис 3
в) поверхности контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали - см. Рис 4
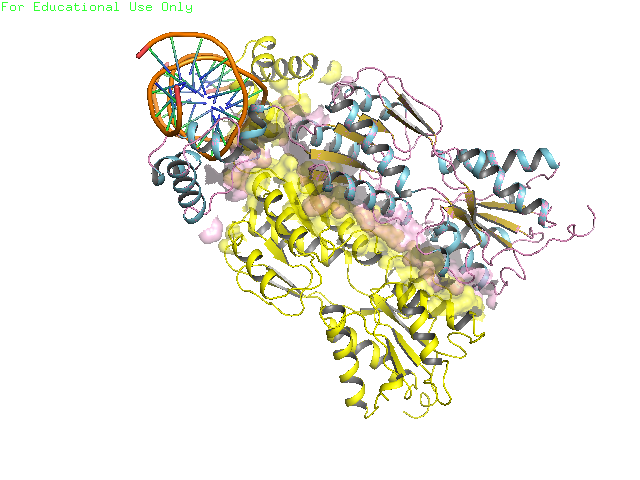
Рис.2
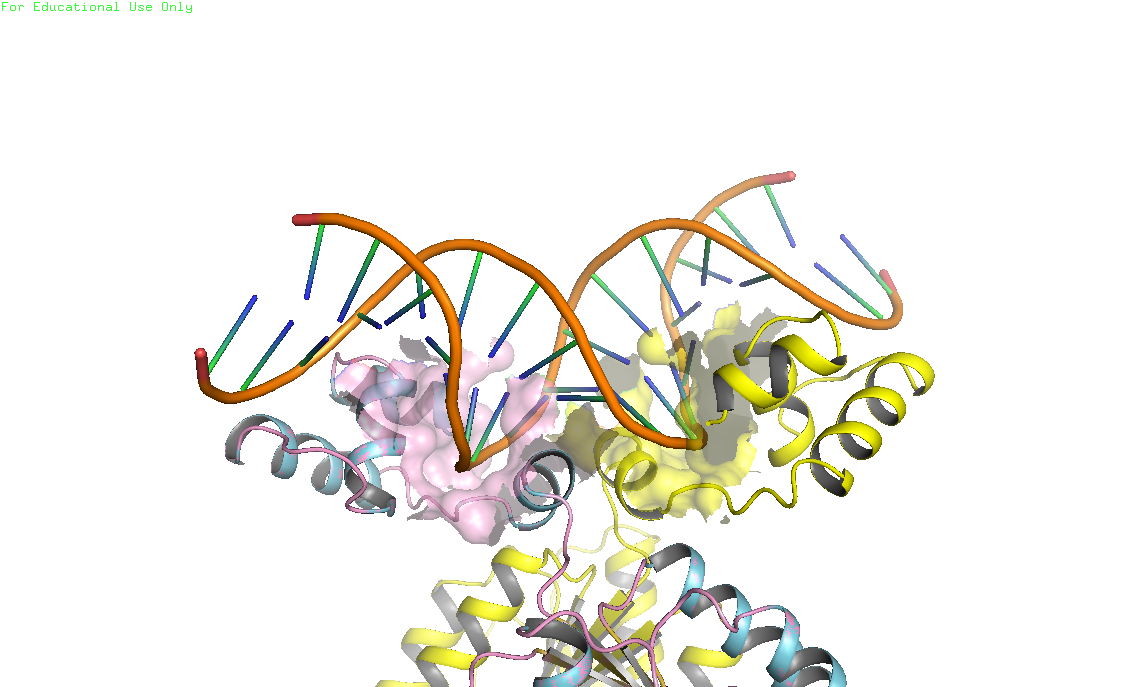
Рис.3
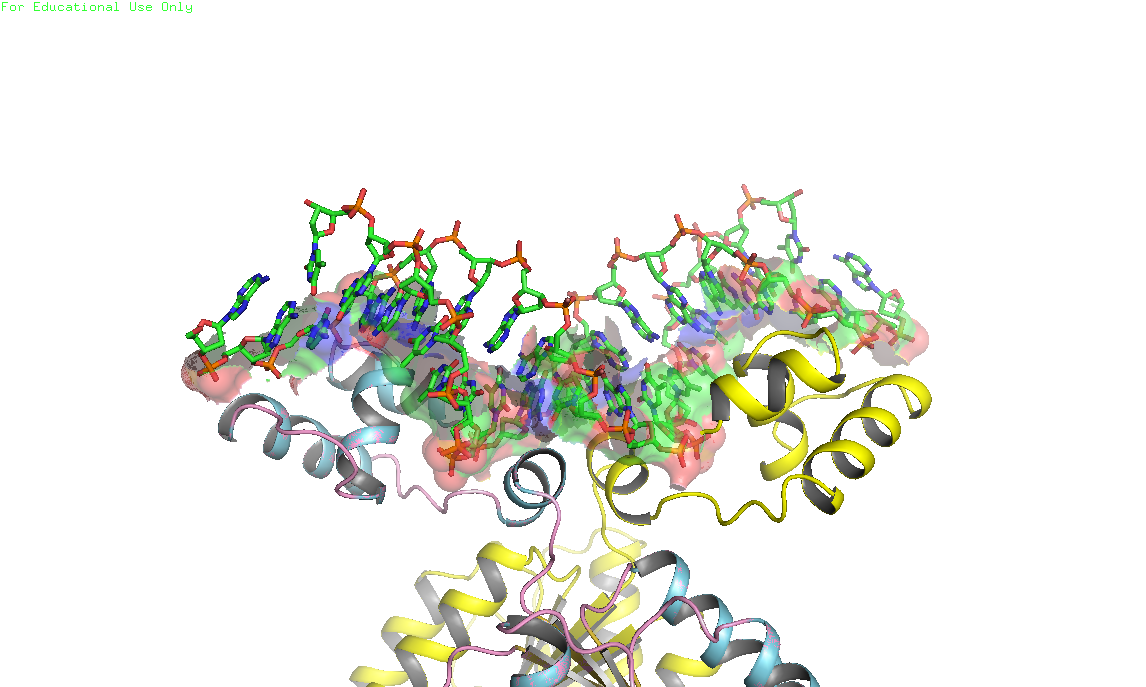
Рис.4
load 1pnr.pdb, A_1 symexp sym, A_1, A_1, 2 bg_color white set ray_opaque_background, off remove hydrogens remove solvent hide lines hide sphere hide sticks hide label hide cartoon hide surface set cartoon_highlight_color, grey40 set cartoon_oval_width, 0.2 set cartoon_oval_length, 0.8 set surface_quality, 2 show cartoon select helix, (ss h) select sheet, (ss s) select loop, (ss l+'') color argon, helix and not c. B color sulfur, sheet and not c. B color pink, loop and not c. B create B_1, A_1 alter_state 1,B_1,(x,y,z)=(x,-y,-z) save 1pnr_dimer.pdb ray 1200 png dimer.png hide cartoon, not c. B show ribbon, not c. B color yellow, A_1 and not c. B color pink, B_1 and not c. B select a_surf, (A_1 and not c. B) w. 5 of (B_1 and not c. B) select b_surf, (B_1 and not c. B) w. 5 of (A_1 and not c. B) show surface, a_surf show surface, b_surf set transparency, 0.5 ray 1200 png dimer_contacts.png hide surface select a_dna_surf, (A_1 and not c. B) w. 5 of (B_1 and c. B) select b_dna_surf, (B_1 and not c. B) w. 5 of (A_1 and c. B) show surface, a_dna_surf show surface, b_dna_surf ray 1200 png dimer_dna_contacts.png hide surface select dna, c. B hide cartoon, dna show sticks, dna select dna_surf, (dna w. 5 of (B_1 and not dna)) + (dna w. 5 of (A_1 and not dna)) show surface, dna_surf ray 1200 png dna_dimer_contacts.png
Порог расстояни 4.3 A, объём кластеров не менее 10 атомов . На странице вывода программы Clud есть файл user_structure.rsc, в котором определены все гидрофобные кластеры и атомы, входящие в них. Были выбраны кластеры, которые находятся близко к участку взаимодействия мономеров (core65, core22, core87, core44, core42):