| Input | Output | Программа | Что делает |
| SRR4240357.fastq (weight=1563M), adapters.fasta (weight=) | task1.fastq (weight=2408M) | java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240357.fastq task1.fastq ILLUMINACLIP:adapters.fasta:2:7:7 | Удаляем возможные остатки адаптеров, предварительно подготовив файл adapters.fasta с помощью
команды cat *.fa > /nfs/srv/databases/ngs/sofyabruman/adapters.fasta Ридов было: 8098979 Ридов стало: 7937658 (98.01%) Ридов убрано: 161321 (1.99%) |
| task1.fastq (weight=2408M) | task1.1.fastq (weight=3251M) | java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 task1.fastq task1.1.fastq MINLEN:30 | Удаляем чтения длины менее 30; Ридов было: 7937658 Ридов стало: 7913995 (99.70%) Ридов убрано: 23663 (0.30%) |
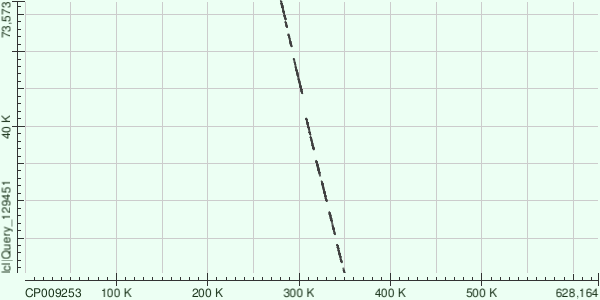
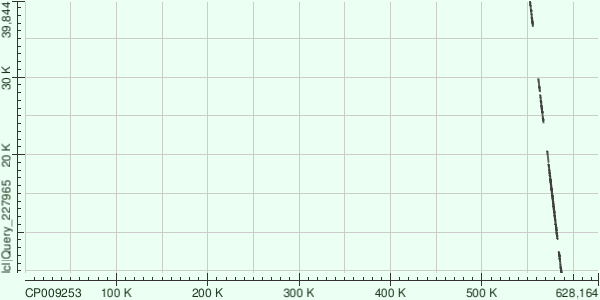
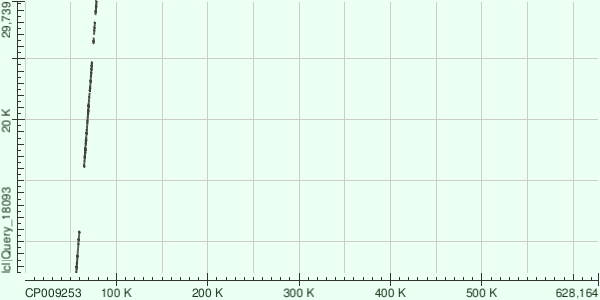
| Contig ID | Length | E-value | % Identity | Gaps / % | Chains | Chr start | Chr end | Part chr length | Coverage |
| 4 | 73570 | 0.0 | 77 | 186/7429(2%) | +/- | 283834 | 357425 | 73592 | 40.949490 |
| 3 | 39816 | 0.0 | 73 | 461/9822(4%) | +/- | 563744 | 603583 | 39840 | 45.481264 |
| 1 | 30625 | 0.0 | 74 | 275/6152(4%) | +/+ | 50035 | 80867 | 30653 | 46.855706 |