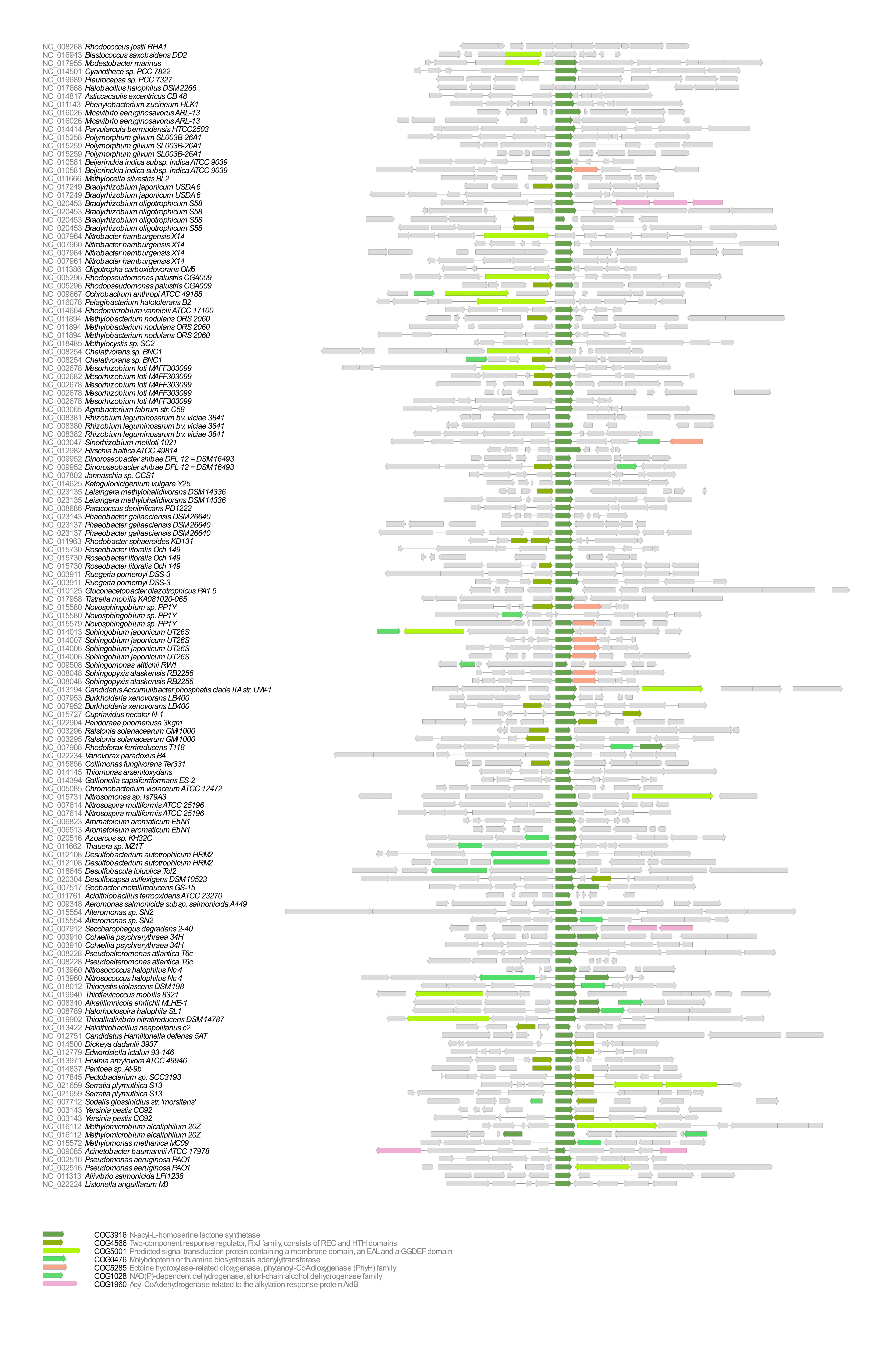
 Для определенных групп организмов характерно достаточно консервативное окружение, однако в целом окружение достаточно вариабельное. Часто рядом с COG3916 встречается COG4566 (болотно-зеленая стрелка),
причем он может быть с двух сторон от нашего гена, или с одной (если слева, то еще и в разных направлениях). Этот ген - двухкомпонентный регулятор ответа семейства FixJ, состоящий из доменов REC и HTH. Он относится к той же функциональной категории, что и наш ген, а также участвует в транскрипции, что говорит об их возможной функциональной связи.
Для определенных групп организмов характерно достаточно консервативное окружение, однако в целом окружение достаточно вариабельное. Часто рядом с COG3916 встречается COG4566 (болотно-зеленая стрелка),
причем он может быть с двух сторон от нашего гена, или с одной (если слева, то еще и в разных направлениях). Этот ген - двухкомпонентный регулятор ответа семейства FixJ, состоящий из доменов REC и HTH. Он относится к той же функциональной категории, что и наш ген, а также участвует в транскрипции, что говорит об их возможной функциональной связи.| Аспект | Идентификатор GO | Название термина | Перевод названия термина | Код типа достоверности |
| Биологический процесс (biological process) | GO:0009372 | Quorum sensing system | Чувство кворума | ISS |
| Код типа достоверности | Расшифровка кода типа достоверности | Объяснение |
| ISS | Inferred from Sequence or structural Similarity | Используется всякий раз, когда основой для аннотации служит анализ последователльности вручную; требует от кураторов создания стабильного идентификатора. Если знаение With/From является геном или его продуктом, для него должны быть экспериментальные (или IC) доказательства. То-есть, вероятно, функциональность была подтверждена экспериментальными данными и расмотрением последовательности вручную. |