Упражнение 1.
В данном упражнении нужно было получить последовательность ДНК на основании данных, полученных из капиллярного секвенатора. Составить отчёт о проблемах при чтении хроматограмм.
Проблемный нуклеотид — тот, по которому вы приняли решение, отличное от предложенного программой, или вы согласились с программой, но необходимо было проанализировать хроматограммы и принять решение. Проблемные нуклеотиды в последовательности выделяйте строчными буквами.
Полиморфизм — это нуклеотид, про который вы решили, что в секвенируемой ДНК встречаются два (или более) варианта. Полиморфизмы обозначайте кодами вырожденных нуклеотидов (ambiguity codes)
Капиллярный секвенатор выдает файлы с хроматограммой и автоматически прочтённой последовательностью в формате .ab1.
Даны два файла в формате .ab1 (WSLOX3_COI_F_D01_WSBS-Seq-1-08-15.ab1 и WSLOX3_COI_R_D02_WSBS-Seq-1-08-15.ab1), соответствующие прочтению прямой и обратной цепочки секвенируемой ДНК.
Ниже представлены произвольные участки двух хроматограмм и их характеристика.
WSLOX3_COI_F_D01_WSBS-Seq-1-08-15.ab1
WSLOX3_COI_R_D02_WSBS-Seq-1-08-15.ab1
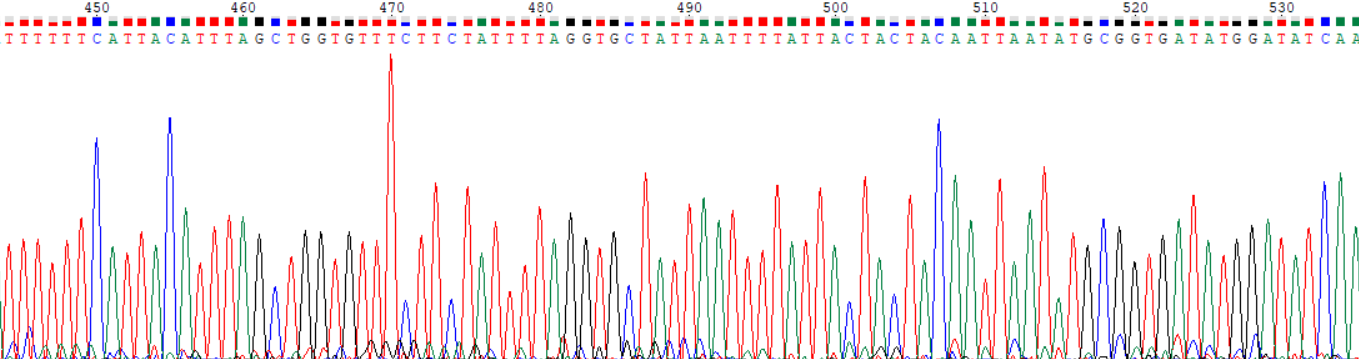
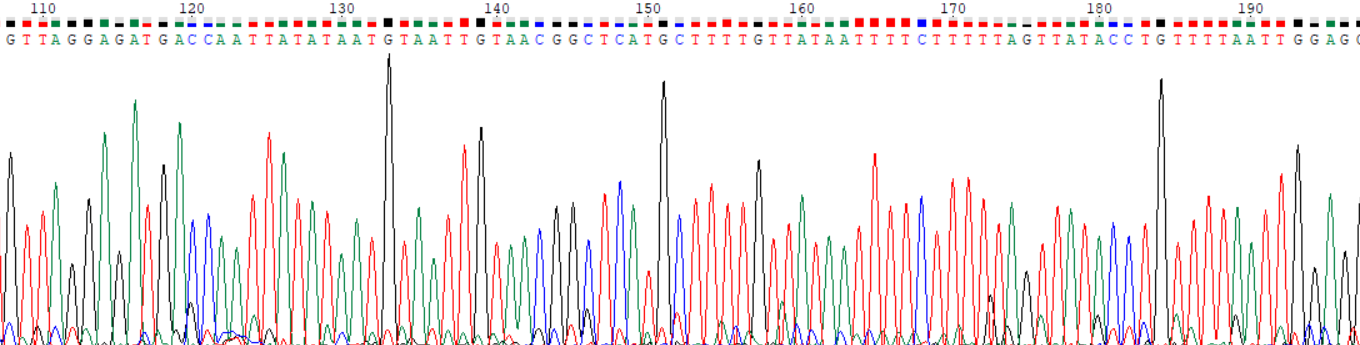
Обе хроматограммы достаточно читаемы, вторая практически идеальна - уровень шума низкий, пики хорошо различимы. Редко встречаются места с шумами уровня пиков.
Сигналы от целевой ДНК идут более или менее равномерно, с одинаковыми расстояниями между пиками и так их можно отличть от шума в спорных позициях.
Присутствует неравномерность силы сигнала и шума вдоль последовательности. Полиморфизмы очень редки.
Ссылки на исходные файлы в .ab1 формате:
WSLOX3_COI_F_D01_WSBS-Seq-1-08-15.ab1 (прямая)
WSLOX3_COI_R_D02_WSBS-Seq-1-08-15.ab1 (обратная)
Таблица 1. Границы нечитаемых 5'- и 3'-участков в каждой последовательности (обратная - Complement Reverse)
| Границы нечитаемых 5'- и 3'-участков |
5'-участок |
3'-участок |
| Прямая последовательность |
1 - 20 |
675 - 719 |
| Обратная последовательность |
1 - 21 |
673 - 720 |
Сначала редактировалась прямая последовательность. Сложные места (шум выше среднего уровня шума и почти как сигнал, пик на нетипичном расстоянии от соседей: вклинился лишний или соседние пики нетипично удалены и т.д.) проверялись по второй цепочке.
Все исправления показаны маленькими буквами. Исправления состояли в замене буквы, удалении лишней буквы, вставке буквы между предложенными софтом секвенатора и т.д. Обе последовательности выровнены программой needle, полученное в формате fasta выравнивание раскрашено по нуклеотидам в JalView.
Рассмотрим несколько интересных случаев ниже:
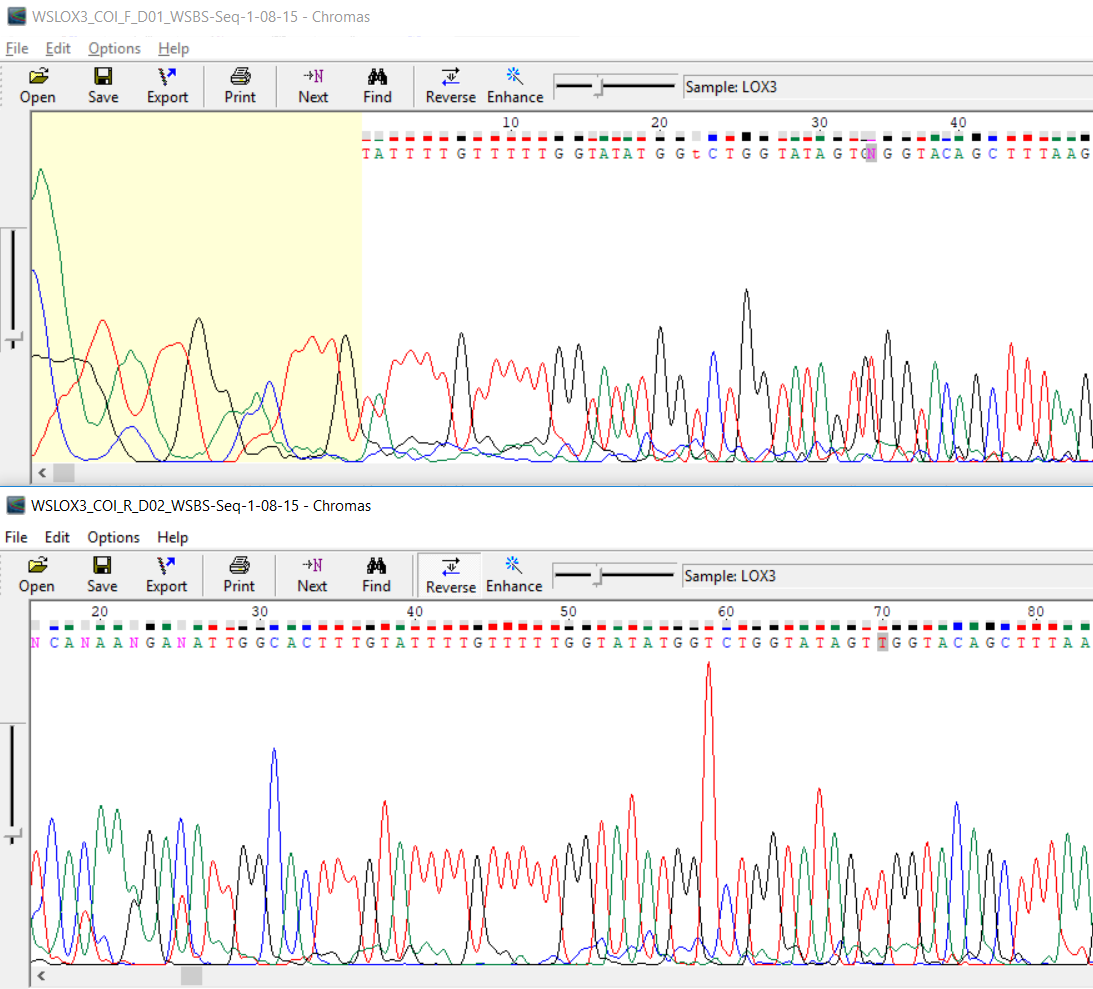
В данном случае в прямой последовательности показано одновременно смещение пиков, наложение пиков, добавление лишнего нуклеотида и смешивание с шумом. С помощью обратной последовательности
можно все интерпретировать достаточно однозначно: вместо G-33 и N-34 ставим t.
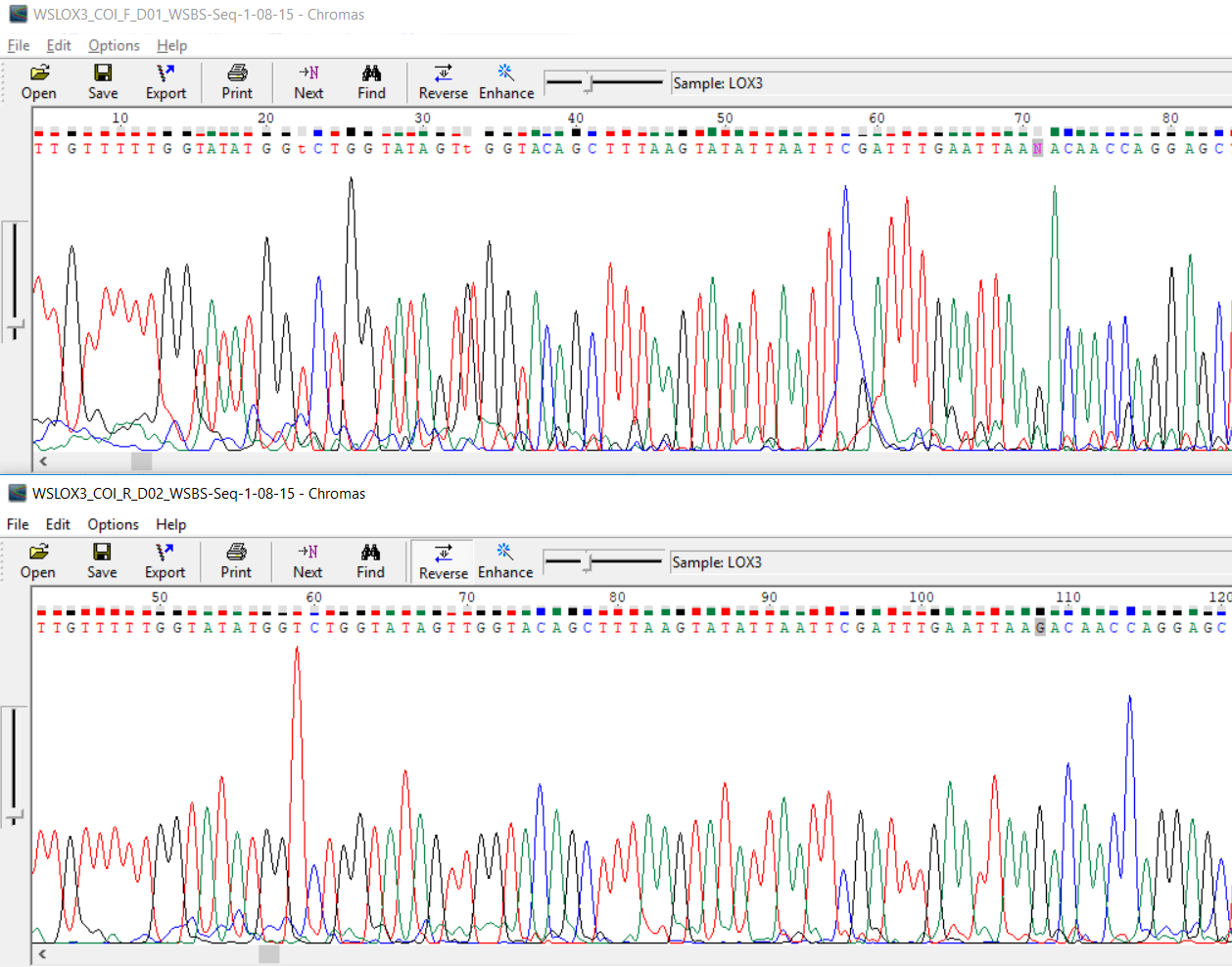
В этом случае сигнал пика оказался слишком низким и смешался с шумом. По второй последовательности можем установить, что N-71 можно заменить на g.
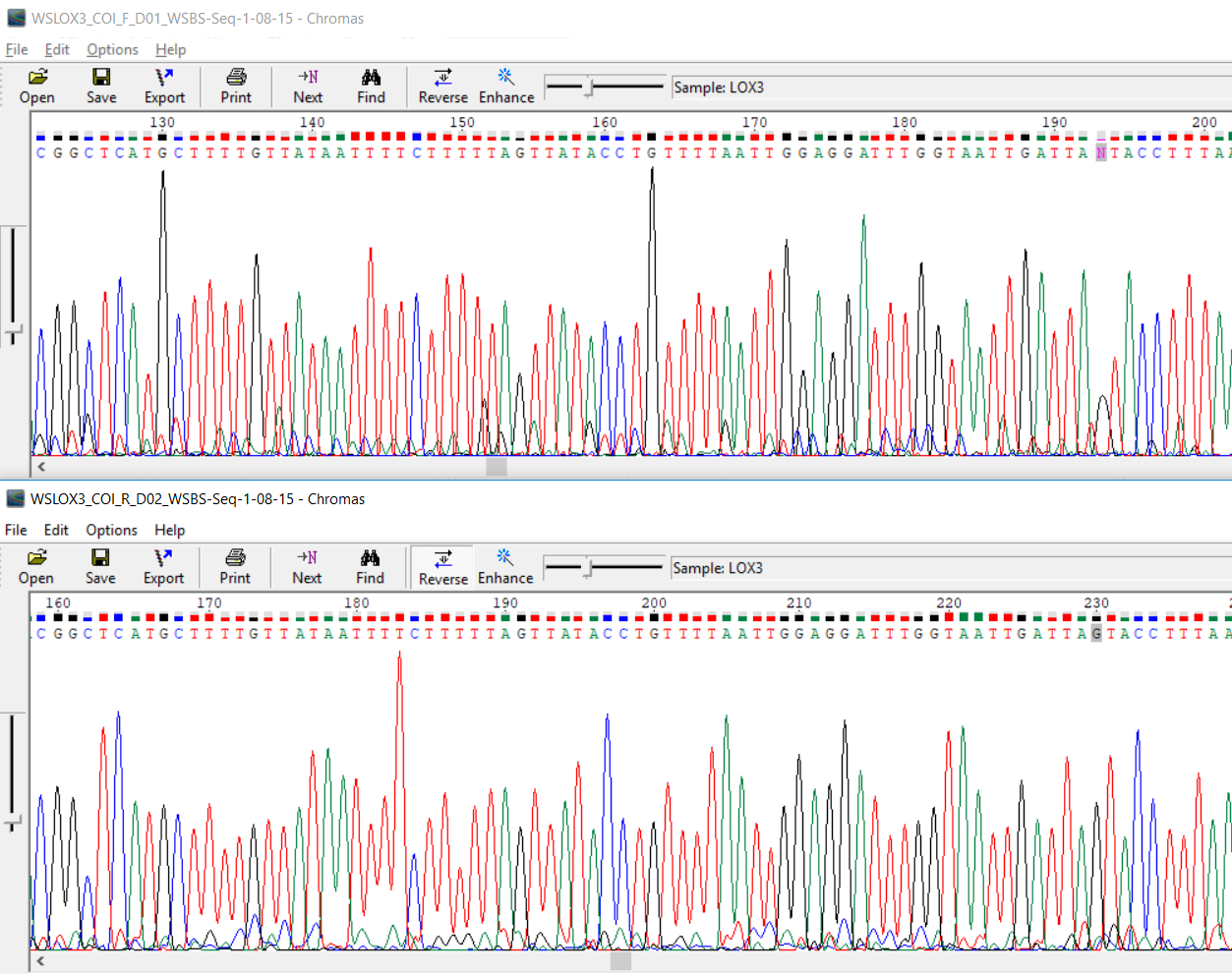
Здесь просто непонятно, что изображено на хроматограмме в данной позиции - шум или сигнал? По второй последовательности можем
однозначно установить, что N-193 можем заменить на g.
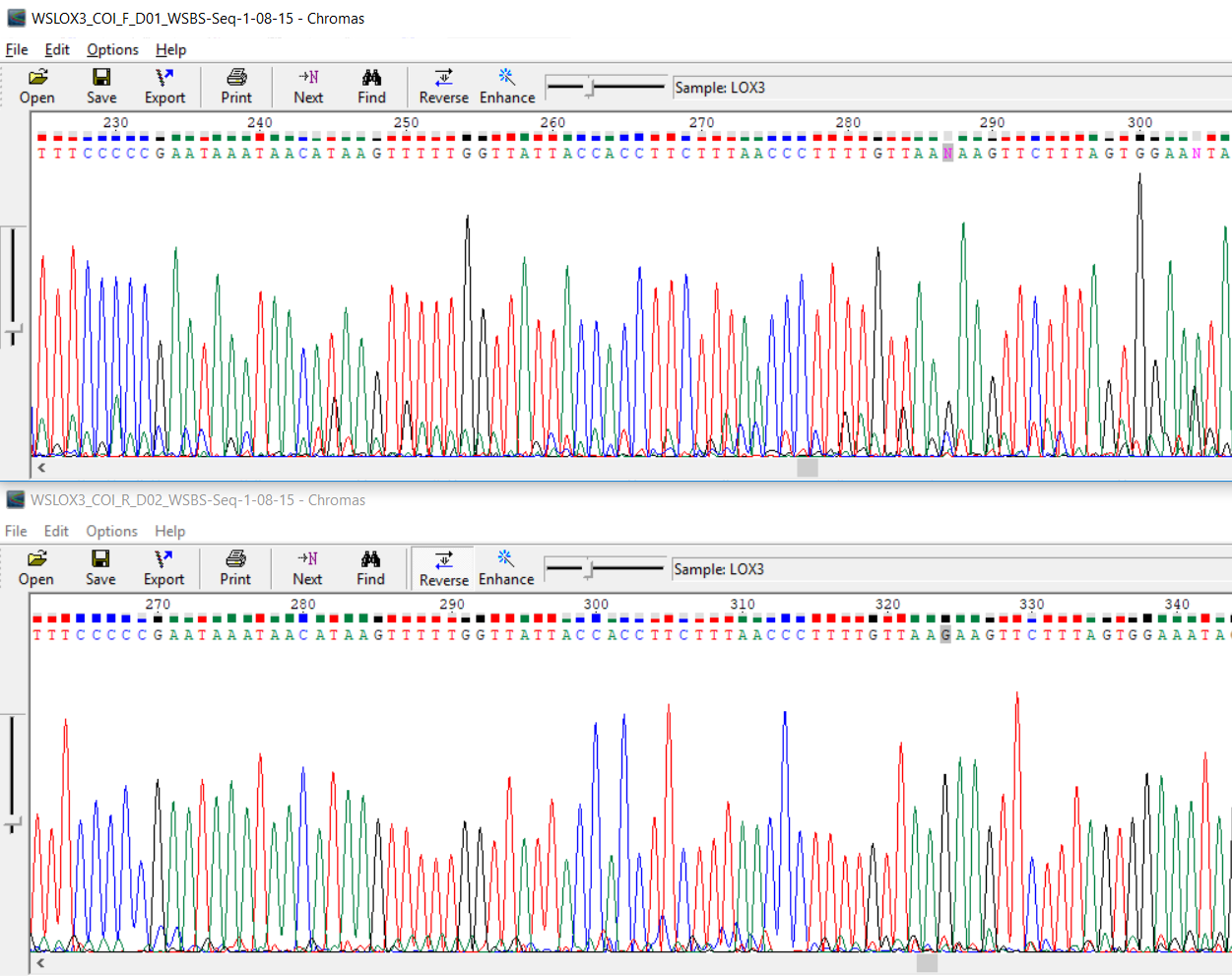
Здесь описано два случая. В первом просто происходит смешивание с шумом и N-287 можно заменить на g, а во втором происходит смешивание с шумом, который
еще и немного смещен по фазе - N-304 можем заменить на a.
Ссылки на файлы с результатом — последовательностями в fasta формате:
Прямая последовательность
Обратная последовательность
Кусок проектa в Jalview

Ссылки:
Выравнивание программой needle
Проект JalView
Упражнение 2.
В данном упражнении нужно было описать нечитаемый участок хроматограммы. Ниже приведен участок хроматограммы прямой последовательности из упражнения 1.
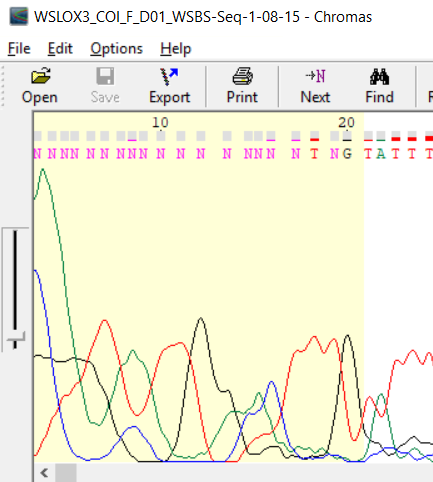
Хроматограмма нечитаема, так как пики размытые, широкие и непериодичные. Непонятно, как отличить сигнал от шума или размытой краски.
Сигналы перекрываются и находятся на одном уровне с шумами, что не позволяет распознать нуклеотиды.
© Бруман Софья, 2018







