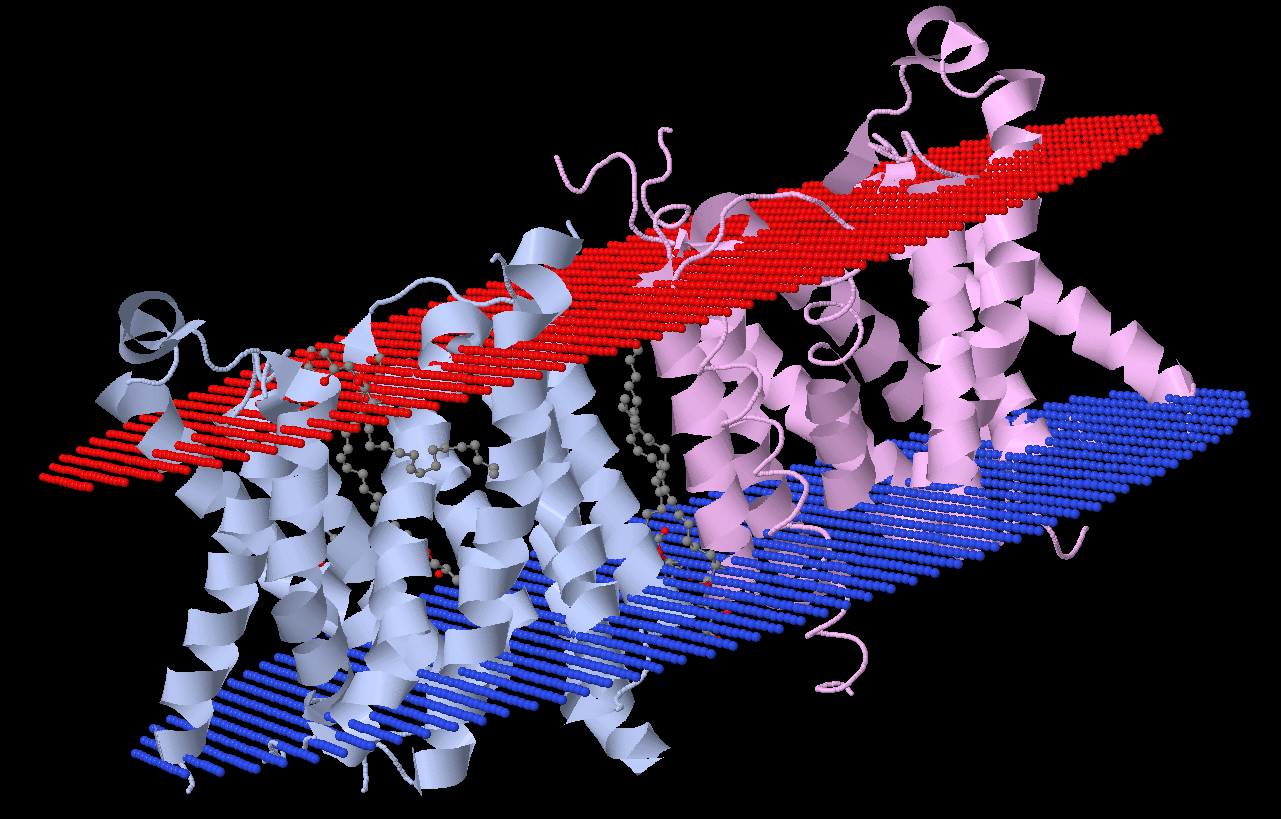

| Толщина гидрофобной части белка в мембране | 25,8 ± 0,2 Å |
| Координаты трансмембранных спиралей |
Subunits: 2 A - Tilt: 8 - TM segments: 1( 23- 40), 2( 51- 72), 3( 84- 106), 4( 111- 128), 5( 140- 158), 6( 184- 202), 7( 214- 235), 8( 252- 273), 9( 278- 299),10( 306- 323) E - Tilt: 7 - TM segments: 1( 23- 40), 2( 51- 72), 3( 84- 106), 4( 111- 128), 5( 140- 158), 6( 184- 202), 7( 214- 235), 8( 252- 273), 9( 278- 299),10( 306- 323) |
| Среднее количество остатков в одной трансмембранной спирали | 20,5 |
| Положение | Трансмембранный в аппарате Гольджи |
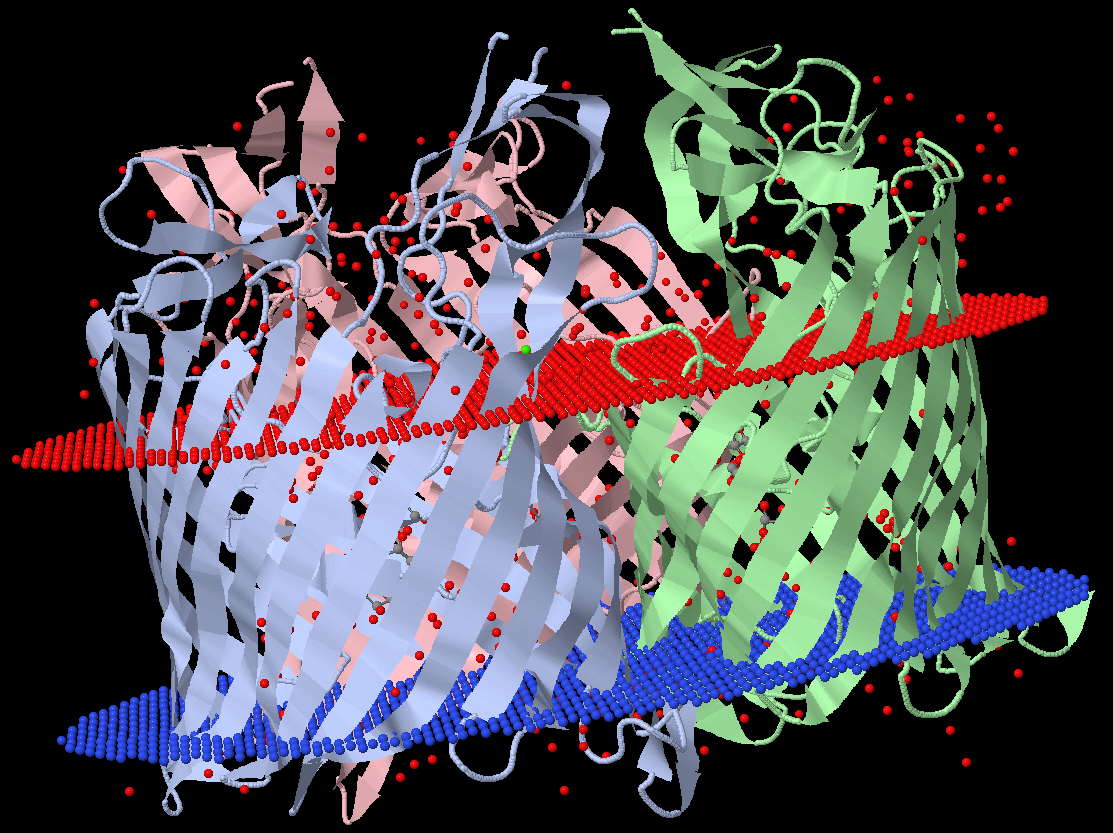
| Толщина гидрофобной части белка в мембране | 24,5 ± 1,0 Å |
| Координаты трансмембранных спиралей |
Subunits: 3 A - Tilt: 3 - TM segments: 1(3-13),2(39-48),3(59-68),4(75-88),5(99-104),6(125-132),7(138-146),8(170-179),9(186-194),10(212-221),11(227-235),12(279-288),13(294-303),14(315-324),15(330-339),16(353-362),17(371-380),18(417-426) B - Tilt: 3 - TM segments: 1(3-13),2(39-48),3(59-68),4(75-88),5(98-104),6(125-132),7(138-146),8(170-179),9(186-194),10(212-221),11(227-235),12(279-288),13(294-303),14(315-324),15(330-339),16(353-362),17(371-380),18(417-426) C - Tilt: 2 - TM segments: 1(3-13),2(39-48),3(59-68),4(75-88),5(98-104),6(125-132),7(138-146),8(170-179),9(186-194),10(213-221),11(227-235),12(279-288),13(294-303),14(315-324),15(330-339),16(353-362),17(371-380),18(417-426) |
| Среднее количество остатков в одной трансмембранной спирали | 9,78 |
| Положение | Трансмембранный бета-бочонок наружной мембраны грамотрицательной бактерии |
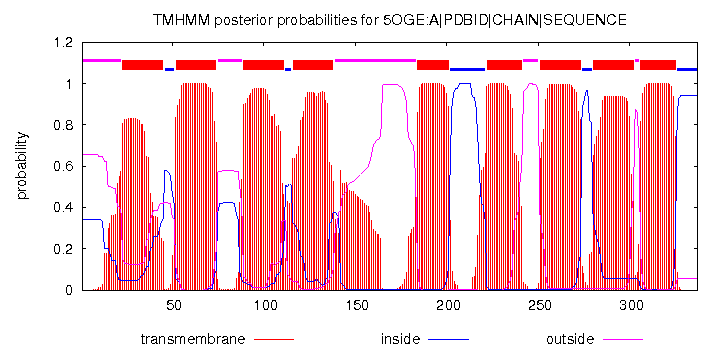
# 5OGE:A|PDBID|CHAIN|SEQUENCE Length: 337 # 5OGE:A|PDBID|CHAIN|SEQUENCE Number of predicted TMHs: 9 # 5OGE:A|PDBID|CHAIN|SEQUENCE Exp number of AAs in TMHs: 195.48946 # 5OGE:A|PDBID|CHAIN|SEQUENCE Exp number, first 60 AAs: 27.48036 # 5OGE:A|PDBID|CHAIN|SEQUENCE Total prob of N-in: 0.34273 # 5OGE:A|PDBID|CHAIN|SEQUENCE POSSIBLE N-term signal sequence 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 1 22 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 23 45 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 46 51 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 52 74 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 75 88 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 89 111 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 112 115 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 116 138 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 139 183 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 184 201 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 202 221 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 222 241 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 242 250 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 251 273 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 274 279 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 280 302 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 303 305 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 306 325 5OGE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 326 337Первая строчка несет информацию об имени и длине последовательности - А-субъединицы изучаемой структуры 5OGE. Вторая строчка - о количестве предсказанных трансмембранных альфа-спиралей (TMH - TransMembrane Helices); их у нас 9. Третья строчка говорит о предполагаемом количестве аминокислотных остатков, входящих в среднем в состав альфа-спирали. Четвертая строчка - ожидаемое количество аминокислотных остатков, входящих в состав трансмембранной альфа-спирали, из первых 60 аминокислот. В данном случае это число большое, поэтому, вероятно, N-конец является лидерным пептидом или какой-либо сигнальной последовательностью. Пятая строчка содержит инфрмацию о полной вероятности того, что N-конец белка расположен на внутренней стороне мембраны - в данном случае это 91,3%. Последующие строки сообщают о номерах аминокислотных остатков, располагающихся с наружной/внутренней стороны мембраны или непосредственно внутри.
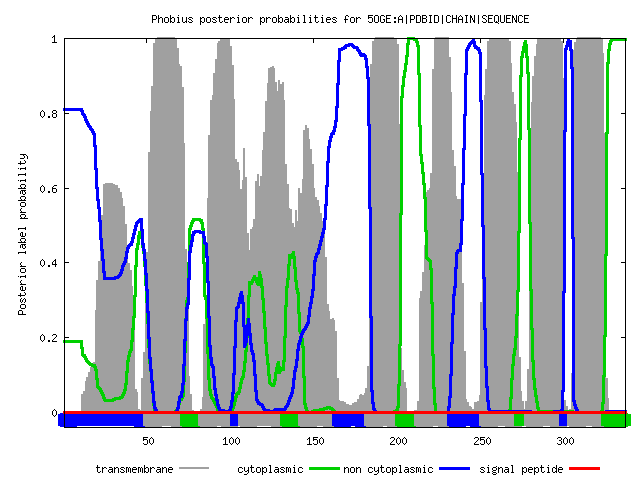
ID 5OGE:A|PDBID|CHAIN|SEQUENCE FT TOPO_DOM 1 51 NON CYTOPLASMIC. FT TRANSMEM 52 73 FT TOPO_DOM 74 84 CYTOPLASMIC. FT TRANSMEM 85 103 FT TOPO_DOM 104 108 NON CYTOPLASMIC. FT TRANSMEM 109 133 FT TOPO_DOM 134 144 CYTOPLASMIC. FT TRANSMEM 145 164 FT TOPO_DOM 165 183 NON CYTOPLASMIC. FT TRANSMEM 184 202 FT TOPO_DOM 203 213 CYTOPLASMIC. FT TRANSMEM 214 233 FT TOPO_DOM 234 252 NON CYTOPLASMIC. FT TRANSMEM 253 273 FT TOPO_DOM 274 279 CYTOPLASMIC. FT TRANSMEM 280 300 FT TOPO_DOM 301 305 NON CYTOPLASMIC. FT TRANSMEM 306 325 FT TOPO_DOM 326 337 CYTOPLASMIC.Первая строка нечет информацию об имени (идентификаторе) последовательности - А-субъединицы. Вторая и последующие строка содержит информацию о границах цитоплазматического участка белковой последовательности. Интересующие нас участки находятся в полях TRANSMEM. Точно предсказаны "границы" только участка 6( 184- 202). Участка 1( 23- 40) и даже похожего на него в данном предсказании нет совсем. "Границы" совершенно не совпадают с TMHMM, в данном алгоритме они гораздо более точные. Среднее количество АО на спираль также отличается и равно 20,7, что практически совпадает с референсом.