Поиск информации в Uniprot о белке Anti-sigma factor RsrA бактерии Streptomyces coelicolor
(strain ATCC BAA-471 / A3(2) / M145) в Uniprot
Таблица 1. Общая информациия о белке (Anti-sigma factor RsrA)
| Uniprot ID | Uniprot AC | Refseq ID | PDB ID | Длина (амин.ост.) | Молекулярная масса (Да) | Рекомендуемое Uniprot название |
| RSRA_STRCO | Q7AKG8 | NP_629364.1;
WP_003973755.1 | 5FRF;
5FRH | 105 | 11681 | Anti-sigma factor RsrA |
Комментарии к таблице 1:
Согласно данным, полученным с сайта Uniprot, исследованный белок носит название Антисигма-фактор RsrA (Anti-sigma factor RsrA). Он характерен для организма Streptomyces coelicolor (штамм ATCC BAA-471 / A3(2) / M145). Исходя из информации Uniprot, PE (степень достоверности) 1 означает, что выявлены эксперементальные доказательства существования белка. Белок был занесён в базы данных UniProtKB/Swiss-Prot 16 октября 2013 года. Длина белка составляет 105 аминокислотных остатка, молекулярная масса составляет 11681 Да. Белок выполняет функцию редокс-регулируемого антисигма-фактора для сигма-фактора с экстрацитоплазматической функцией (ECF) SigR и функцию ключевого датчика дисульфидного стресса; удерживает SigR (его родственный сигма-фактор ECF в неактивной форме), ингибируя его сигма-активность при восстановлении, но не в условиях окисления. Окисление и восстановление антисигма-фактора обратимы. Mycothiol (MSH) ответственен за восстановление RsrA, что позволяет ему связываться с SigR. В сочетании со своим родственным сигма-фактором SigR может распознавать восстановленный MSH на внутриклеточном уровне; вероятно, высвобождает SigR во время окислительного стресса.
На одну субъединицу приходится (синтезируется) один ион Zn2+. Катион цинка не требуется для связывания SigR, но он необходим для активности антисигма-фактора. Цинк делает RsrA относительно устойчивым к окислению.
Структура белка известна полностью. Белок содержит одну цепь. [1]
Таблица 2. Информация о кластерах
| Uniref | Cluster ID | Cluster name | Size |
| Uniref100 | UniRef100_Q7AKG8 | Anti-sigma factor RsrA | 3 |
| Uniref90 | UniRef90_Q7AKG8 | Anti-sigma factor RsrA | 12 |
| Uniref50 | UniRef50_Q7AKG8 | Anti-sigma factor RsrA | 68 |
Результаты сеансов поиска в Uniprot
Поиск по белку Anti-sigma factor RsrA
1) Среди всех организмов
| Текст запроса: name:"anti sigma factor rsra" |
| Сколько белков: 42 |
| Сколько из раздела Reviewed: 1 |
Ссылка |
2) В исходном организме
| Текст запроса: name:"anti sigma factor rsra" AND organism:"Streptomyces coelicolor (strain ATCC BAA-471 / A3(2) / M145) [100226]" |
| Сколько белков: 1 |
| Сколько из раздела Reviewed: 1 |
Ссылка
3) Среди семейства организма (Streptomycetaceae)
| Текст запроса: name:"anti sigma factor rsra" taxonomy:streptomycetaceae |
| Сколько белков: 14 |
| Сколько из раздела Reviewed: 1 |
Ссылка
4) Среди отдела организма (Actinobacteria)
| Текст запроса: name:"anti sigma factor rsra" taxonomy:actinobacteria |
| Сколько белков: 34 |
| Сколько из раздела Reviewed: 1 |
Ссылка
Поиск по гистонам
1) Среди всех организмов
| Текст запроса: name:histone |
| Сколько белков: 131541 |
| Сколько из раздела Reviewed: 2083 |
Ссылка
|
2) Среди Metazoa
| Текст запроса: name:histone taxonomy:"Metazoa [33208]" |
| Сколько белков: 63788 |
| Сколько из раздела Reviewed: 1040 |
Ссылка
|
3) Среди бурых водорослей
| Текст запроса: name:histone taxonomy:phaeophyceae |
| Сколько белков: 46 |
| Сколько из раздела Reviewed: 0 |
Ссылка
|
Поиск по трипсину
1) По слову трипсин
| Текст запроса: name:trypsin |
| Сколько белков: 18951 |
| Сколько из раздела Reviewed: 311 |
Ссылка
2) Ингибиторы
| Текст запроса: name:trypsin name:inhibitor |
| Сколько белков: 3914 |
| Сколько из раздела Reviewed: 210 |
Ссылка
История изменений записи Uniprot
Uniprot дает возможность просматривать статистику изменений записей о белке. Во вкладке History имеются все версии записей. Также возможно сравнивать разные версии записей. На момент изучения на Uniprot последней версией является версия под номером 37. По сравнению с первой версией произошло большое количество изменений.
К примеру, были изменены строки ID (Q7AKG8_STRCO “Unreviewed” -> RSRA_STRCO “Reviewed”) и AC (Q7AKG8 -> Q7AKG8; Q9RL96).
Также изменениям подверглись строки DT. Изначально файл содержал только одну строку:
SubName: Full=Anti-sigma factor;
В актуальной же версии строка заменена на три строки:
RecName: Full=Anti-sigma factor RsrA;
AltName: Full=Regulator of SigR;
AltName: Full=Sigma-R anti-sigma factor RsrA.
Были подкорректированы строки OC (современное положение организма):
Bacteria; Actinobacteria; Actinobacteridae; Actinomycetales;
Streptomycineae; Streptomycetaceae; Streptomyces.
Было заменено на:
Bacteria; Actinobacteria; Streptomycetales; Streptomycetaceae;
Streptomyces; Streptomyces albidoflavus group.
Также был добавлен большой комментарий с подробным описанием белка и добавлены цитирования с 3 по 8.
Было дописано обширное описание локальных особенностей последовательностей. (FT)
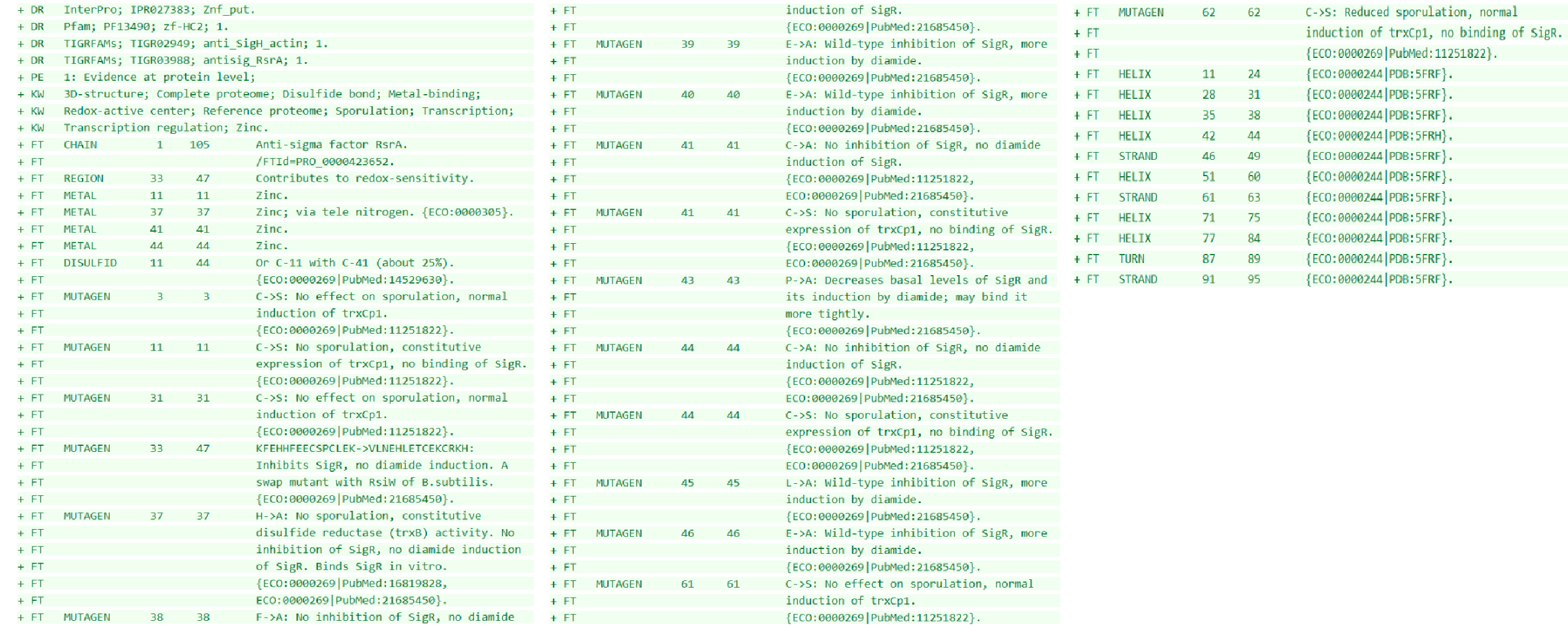
Записи характеристик участков последовательности в формате Uniprot:
Все записи характеристик участков последовательности в файле Uniprot описываются в строке FT (feature table). Ниже взят один из примеров:
- Дисульфидные связи:

Сравнение данных в базах Uniprot и RefSeq
Были сопоставлены записи о белке в RefSeq Protein [2] и Uniprot. Кардинальных различий обнаружено не было, однако стоит отметить, что в RefSeq более просто ориентироваться. Также дата обновления данных в RefSeq страрше на почти год. Из-за этого в RefSeq отмечено всего лишь 5 цитирований, тогда как в Uniprot - 8. В обеих версиях можно просмотреть последовательность белка в FASTA-формате и полное современное систематическое положение организма, содержащего белок. Недостатком RefSeq является отсутствие идентификаторов других баз данных, что ограничивает возможность получения дополнительной информации о белке.
Ссылки на источники:
© Бруман Софья, 2018
|

