9.1 Pfam
Ссылка на таблицу
Выбранная архитектура

Некоторый белок бактериального жгутика, отвечающий за моторную активность
PDB основного домена
Интересно, что когда домен встречается сам по себе он отвечает за некоторый синтез,
сопряженный со жгутиком - и напоминает АТФ-синтазу.
Таблица 1.
| номер |
ID |
AC |
название |
число последовательностей среди бактерий |
| 1 |
FliH |
PF02108 |
Flagellar assembly protein FliH |
3092 |
| 2 |
Yae1_N |
PF09811 |
Essential protein Yae1, N terminal |
75 |
| Доменная архитектура |
I0QMR4_9GAMM |
I0QMR4 |
Flagellar assembly protein H |
43 в Pfam, 243 в Uniprot |
HMM gрофиль семейства белков
1. Отбор последовательностей
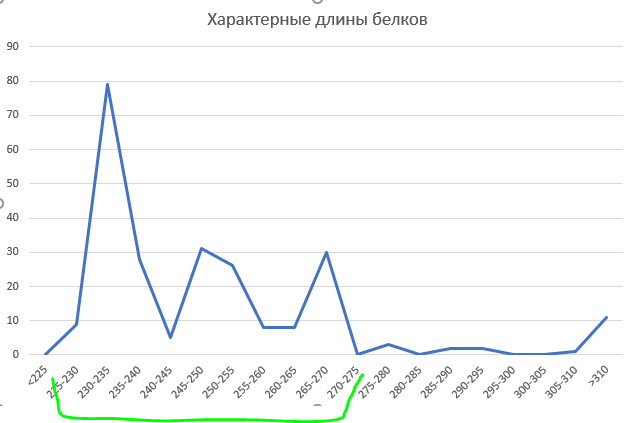
Сначала я посроила гистограмму длин белков. В ней я взяла длины с 225 до 275 аимнокислот. Конечно,
гистограмма имеет странный вид - вероятно, есть три класса белков, а может и нет.
Далее я оставила только нужные строки в таблице, убрав выбивающиеся по длине.
С помощью средств Excel (сортировка по виду, сортировка по семейству) я выбрала примерно 50
белков. (смотреть в общей аблице на листе номер 2 или в файле
)
Их AC из первой колонки я скопировала в файл.
Далее в Jalview (File -- Fetch sequences -- Uniprot -- OK -- RetrieveIDs) вставила АС, и достала последовательности.
С помощью Muscle with defaults выровняла.
Далее вруучную отредактировала выравнивание:
убрала 20 колонок с N-конца

14 колонок с C-конца

а также 7 выбивающихся из выравнивания последовательностей. Заново выровняла, сохранила в
файл с итоговым выравниванием
hmm2build build.txt aligned_2.fa
hmm2calibrate build.txt
файл с итоговым выравниванием
Я буду тестировать на последовательностях с доменом pf02108, их 15506 штук
Команда поиска
hmm2search <профиль> <файл с последовательностями>
-domE <порог E-value для доменов> -domT <порог веса T для доменов>
hmm2search -E 0.1 build.txt first_dom15000.fasta > findings.txt
Итак, в файле у нас теперь лежат все находки - те
последовательности из базы данных (в нашем случае, в учебных целях, это все белки у которых есь один из доменов),
обладающих той же архитектурой (то есть 2мя доменами )
700 tr|A0A104A7C7|A0A104A7C7_BURCE 1-Jan 18 227 .] 1 233 []
197.4 5.90E-05 0 175 0.720164609 0.034396908 0.965603092
Была построена ROC-кривая
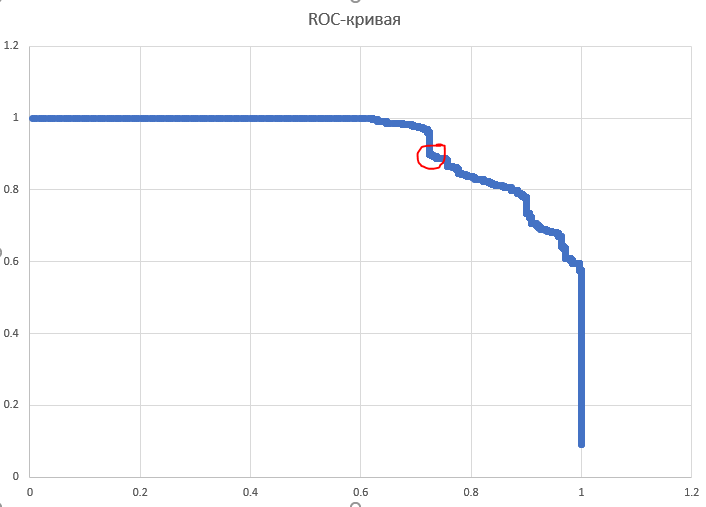
И гистограмма весов
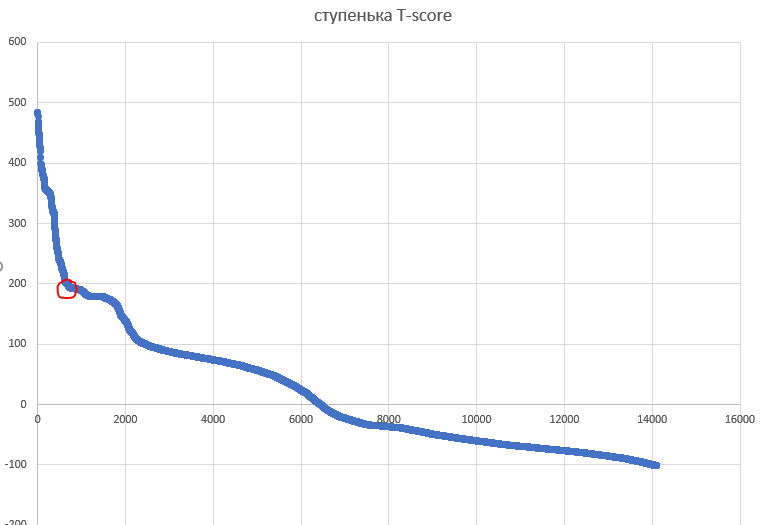
Посмотрев на них, я взяла точку, отмеченную красныч, соответствующую порогу на вес 197.4 и E-value = 5.90E-05 .
Соответствующая таблица:
Таблица 2.
|
Test positive |
Test negative |
sum |
| True positive |
175 |
68 |
243 |
| True negative |
525 |
15258 |
15263 |
| sum |
700 |
14806 |
15506 |
Ссылка на таблицу нужен лист номер 4
Как видно из таблицы, тест мой так себе - специфичность у него низковата.
