 или
или 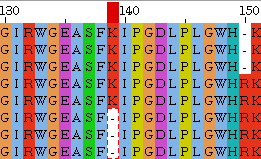
Для работы были выбраны шесть последовательностей гомологичных белков семейства HSP70 у эукариот, архей и бактерий. В таблице ниже представлены данные
| Entry | Entry name | Protein names | Length | Superkingdom | Organism |
| Q9HRY2 | DNAK_HALSA | Chaperone protein DnaK (HSP70) | 629 | Archaea | Halobacterium salinarum (strain ATCC 700922 / JCM 11081 / NRC-1) (Halobacterium halobium) |
| P0CW13 | DNAK_METMA | Chaperone protein DnaK (HSP70) | 619 | Archaea | Methanosarcina mazei (strain ATCC BAA-159 / DSM 3647 / Goe1 / Go1 / JCM 11833 / OCM 88) (Methanosarcina frisia) |
| B5Z9P0 | DNAK_HELPG | Chaperone protein DnaK (HSP70) | 620 | Bacteria | Helicobacter pylori (strain G27) |
| Q82EX9 | DNAK1_STRAW | Chaperone protein dnaK1 (HSP70-1) | 622 | Bacteria | Streptomyces avermitilis (strain ATCC 31267 / DSM 46492 / JCM 5070 / NBRC 14893 / NCIMB 12804 / NRRL 8165 / MA-4680) |
| P20163 | HSP7D_CAEEL | Heat shock 70 kDa protein D | 657 | Eukaryota | Caenorhabditis elegans |
| P29843 | HSP7A_DROME | Heat shock 70 kDa protein cognate 1 | 641 | Eukaryota | Drosophila melanogaster (Fruit fly) |
Используя опцию Tcoffee with Defaults, было смоделировано выравнивание последовательностей белков с раскраской по схеме ClustalX, при Identity Threshold = 100%. Часть выравнивания приведена ниже ссылкой на полное выравнивание. Так же предоставляю ссылку на файл, автоматически сохданный JalView в формате .jvp Помимо базовой разметки, вроде консервативности, качества выравнивания и консенсуса (достоверности подстановки конкретного остатка на данной позиции) добавлена новая строка разметки по типам консервативности, где соответствующими буквами отмечены в качестве примера некоторые остатки консервативные на 80% или более (C), абсолютно функционально консервативные (F), и позиций с гэпами (G).
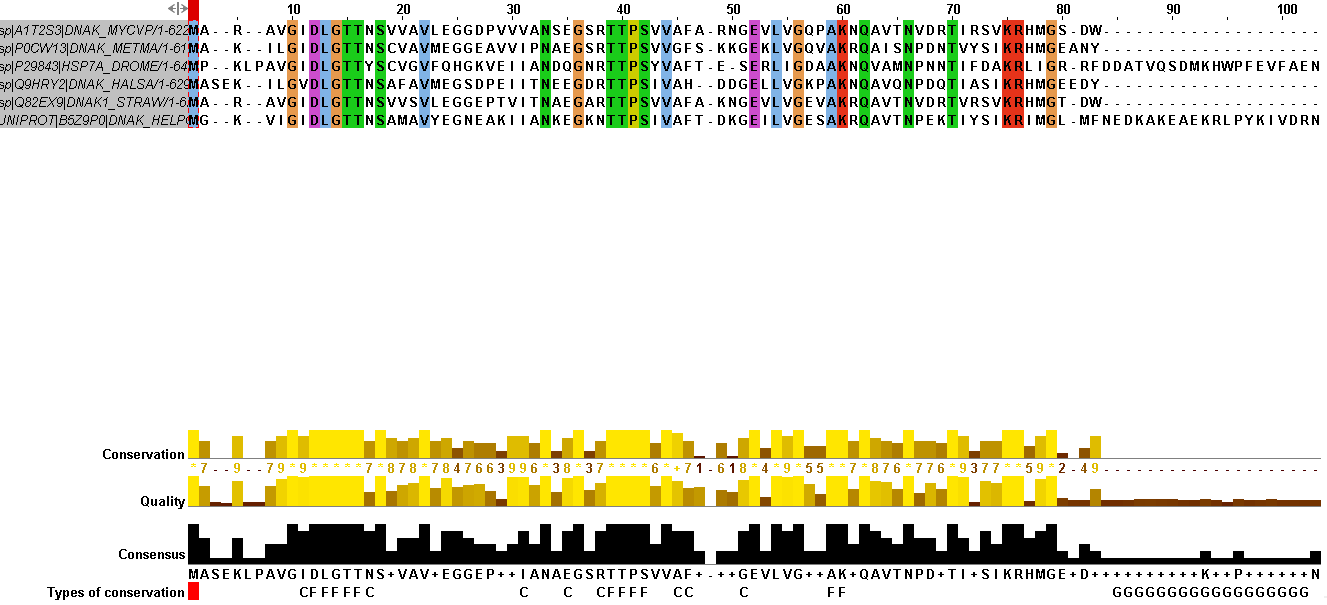
Кластер следующих таблиц отображает некоторые параметры выравнивания - каждой из исследуемых с помощью команды infoalign пакета EMBOSS последовательностей соответсвует строка таблицы следующего содержания: Name-Seqlen (имя и длина); AlignLen(длина выравнивания); GapLen,%(число и процент позиций с гэпами); Ident(число и процент идентичных позиций). Данные, полученные для консервативных и абсолютно консервативных позиций представлены в таблицах 2-4 . Функционально консервативной (с возможностью быть отмеченной F в выравнивании) считается позиция, в которой стоят те аминокислотные остатки, что обладают схожими свойствами, обусловленными схожими функциональными группами. Аминокислоты делятся на классы, с учётом их свойств и структурному составу радикала: ароматические, кислые, основные, амиды, серосодержащие и т.д. С Замена на позиции, приводящая к появлению на месте исходной аминокислоты той, что обладает схожими свойствами, характеризует консервативность данной позиции.
| Таблица 2.Консервативность 100% | Таблица 3.Консервативность 70% и выше: | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Таблица 4.Функциональная консервативность 100%: | Таблица 5.Функциональная консервативность 70% и выше: | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
Число консервативных позиций складывается из Ident и Similar. Представим сводную таблицу о выравнивании:
| Белок | Число абсолютно консервативных позиций | Доля, % | Число функционально консервативных позиций | Доля, % | Число гэпов | Доля, % | Число позиций, консервативных на 70% | Доля, % |
| DNAK_MYCVP | 181 | 26 | 298 | 43.6 | 61 | 9 | 371 | 54.3 |
| DNAK_METMA | 181 | 26 | 298 | 43.6 | 64 | 9.3 | 371 | 54.3 |
| HSP7A_DROME | 181 | 26 | 298 | 43.6 | 42 | 6.1 | 371 | 54.3 |
| DNAK_HALSA | 181 | 26 | 298 | 43.6 | 54 | 8 | 371 | 54.3 |
| DNAK1_STRAW | 181 | 26 | 298 | 43.6 | 61 | 9 | 371 | 54.3 |
| DNAK_HELPG | 181 | 26 | 298 | 43.6 | 63 | 9.2 | 371 | 54.3 |
В данной части, с целью симулировать процесс эволюции, была искусственно произведена мутация белка.
За основу был взят фрагмент из ста нуклеотидов белка 4-альфа-амиломальтазы Corynebacterium glutamicum, уже известной нам по предыдущему практикуму,
(Uniprot ID: Q8NNA7_CORGL) со 110 по 210 аминокислоту.
С помощью команды msbar пакета EMBOSS в последовательность вносились мутации всех типов, кроме блоковых.
Ознакомиться со скриптом можно по данной ссылке.
В модельном эксперименте за единицу времени, 100 лет, в последовательности происходит семь точечных мутаций. Наблюдается 7 поколений предка: p > p1 > p2 > ... > p7.
Для каждого следующег поколения предковой последовательностью считается послежовательность предыдущего поколения, уже претерпевшая какие-либо изменения(если это не изначальная последовательность, конечно).
Таким образом в конце эксперимента мы имеем 8 поколений.
Полученные последовательности были импортированы в программу Jalviewer, после чего выровнены с помощью Tcoffee with Defaults.

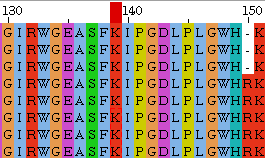
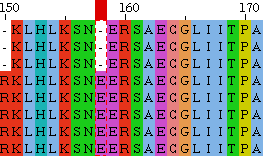
Соответственно, мы мутации на выбранном участке (они представлены в порядке возникновения):
 или или  |  |
| Очевидно, что замены лизина на валин вообще не могло произойти, потому как эти аминокислоты не аналогичны. Значит на этом месте может либо остаться лизин, либо появиться гэп | Возникновение гэпа на месте только что вставленной, то есть эволюционно-выгодной аминокислоты, тоже можно посчитать невозможным. |
Ссылки:
На главную страницуВернуться назад
©Solonovich Vera,2017