После первых неудачных опытов, был проведён поиск белков, сходных со знакомым нам ещё с первого семестра 4-alpha-glucanotransferase(AFH91336.1), белком Corynebacterium pseudotuberculosis 31. Поиск проходил по базе
данных UniProtKB, так как поиск по SwissProt не дал нужных результатов, с базовыми параметрами + E-value = 10, WordSize = 6. Мы всё ещё имеем достаточно неплохой
разброс по E-value. Выборка белков была изменена.
Ниже представлена часть автоматически-скомпелирванной таблицы Hit-table при выдаче в поиске BLAST со столбцами из первой выдачи.
Некоторые колонки было решено опустить, чтобы данные предстали нагляднее. Из последовательностей преставленных на выходе были выбраны следующие.
| Subject IDs | Identity,% | Positives,% | Alignment length | Mismatches | Gap opens | Q.start | Q.end | S.start | S.end | E-value | Bit score |
|---|---|---|---|---|---|---|---|---|---|---|---|
| gi|54037774|sp|P65337.1|MALQ_MYCBO | 44.353 | 58.82 | 726 | 349 | 8 | 7 | 710 | 8 | 700 | 0.0 | 571 |
| gi|1170869|sp|P45176.1|MALQ_HAEIN | 31.944 | 52.26 | 576 | 349 | 14 | 153 | 697 | 110 | 673 | 2.36e-83 | 281 |
| gi|14194917|sp|Q9PKU9.1|MALQ_CHLMU | 24.819 | 46.02 | 415 | 263 | 13 | 189 | 577 | 26 | 417 | 5.17e-28 | 122 |
| gi|1170868|sp|P15977.2|MALQ_ECOLI | 30.796 | 48.62 | 578 | 361 | 12 | 143 | 693 | 94 | 659 | 1.60e-74 | 257 |
| gi|6225648|sp|Q9Z8L2.1|MALQ_CHLPN | 25.000 | 44.17 | 412 | 266 | 14 | 189 | 577 | 22 | 413 | 7.70e-25 | 112 |
| gi|6225649|sp|O34022.1|MALQ_CHLCV | 24.155 | 43.72 | 414 | 265 | 13 | 190 | 577 | 27 | 417 | 2.42e-22 | 104 |
| gi|6225650|sp|O84089.1|MALQ_CHLTR | 24.096 | 44.34 | 415 | 266 | 14 | 189 | 577 | 26 | 417 | 4.41e-22 | 103 |
| gi|332313389|sp|Q8LI30.2|DPE1_ORYSJ | 24.390 | 38.41 | 164 | 117 | 3 | 371 | 529 | 281 | 442 | 0.008 | 43.1 |
| gi|75180614|sp|Q9LV91.1|DPE1_ARATH | 21.362 | 39.01 | 323 | 236 | 10 | 243 | 551 | 128 | 446 | 0.004 | 44.3 |
| gi|75567371|sp|Q82462.1|CAPSD_HASV | 28.889 | 53.33 | 45 | 32 | 0 | 257 | 301 | 482 | 526 | 1.1 | 36.2 |
Таблица 1.Общие данные о последовательностях.
Помимо столбца с идентификаторами белков(ids) и identity, в таблице представлены столбцы: positives - число позиций с положительным весом); mismatches(число замен аминокислот); q. start и q .end/s. start и s. end - координаты выравнивания в изначальном и в сравниваемых белках; E-value; Query cover.
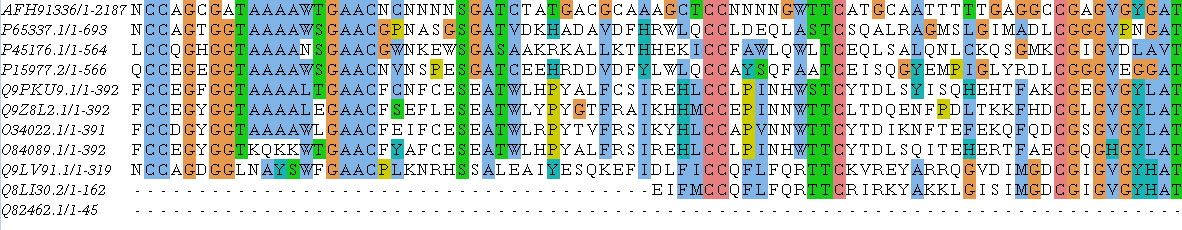
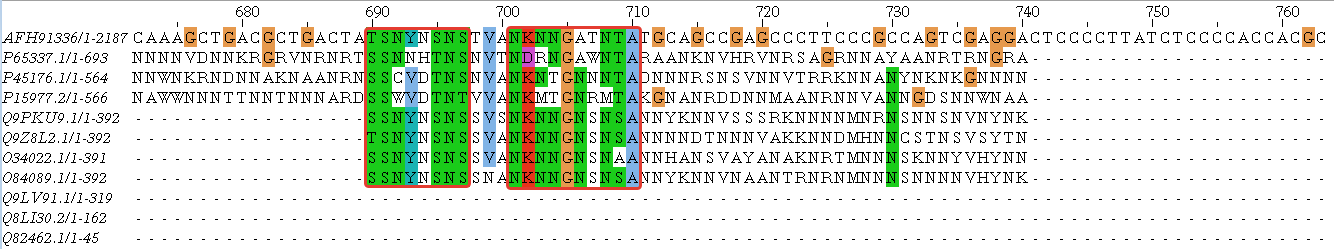
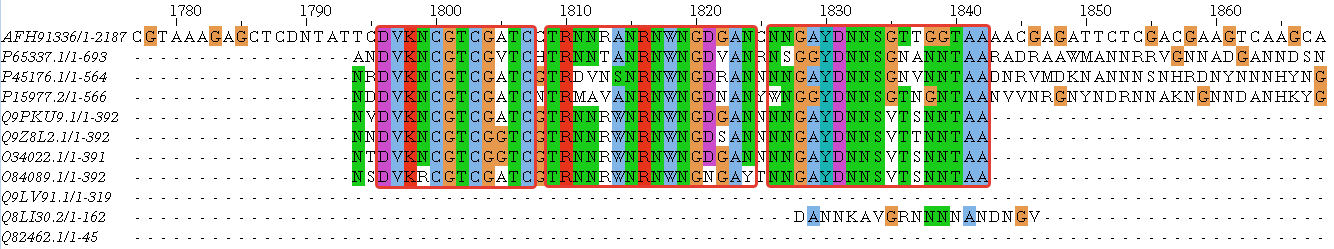
Рисунок 1. Множественное выравнивание.
Было проведено выравнивание с помощью программы JalView. Чтобы ознакомиться с полным выравниванием, воспользуйтесь ссылкой-картинкой или скачайте выравнивание.
Перво-наперво обратимся к значениям E-value и Identity, которые представлены в первой таблице. Очевидно, что чем больший процент консервативных позиций,
тем более схожи последовательности. Стало быть, чем меньше E-value, тем более вероятна гомология.
Поэтому последовательности, с Е-value меньше 1Е-05 скорее всего гомологичны. Тем не менее хорошо бы иметь ещё
параметр Сoverage, который показывает, насколько гомологична вся последовательность, а не отдельный домен. Он состоит из процентного соотношения выравнивания
к длине всей последовательности. И будет приведён в сводной таблице ниже.
Если посмотреть на него, то видно, что лишь у второй последовательности он достаточно высок (то есть можно говорить о гомологии именно последовательности, а не домена).Однако, по одному критерию судить о гомологичности нельзя.
Посмотрим на названия последовательностей, воспользовавшийсь опцией COBALT(Constraint-based Multiple Alignment Tool) представления выравниваний результата
поиска по BLAST. Как видно из Рисунков 2 и 3, выбранные нами белки в большинстве своём, как и изначальный AFH91336.1, представляют 4-альфа-глюканотрансферазы, хотя и
найдены у разных организмов. Исключение составляют последовательности с идентификаторами Q8N2Q7.2(нейролигин-1,
создаёт мостики между молкулами клеток при синапсе двух нейронов) и Q82462.1(белок капсида), они выполняют в орагнизмах совершенно иную функцию, из
чего можно заключить, что они навряд ли гомологи первого нашего белка. К тому же эти белки имеют одни из наибольших E-value в таблице.
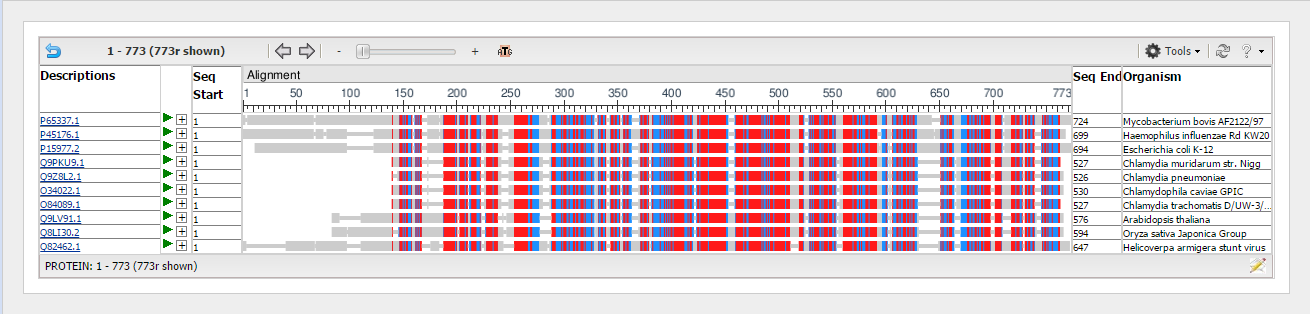
Рисунок 2. Графическое представление.
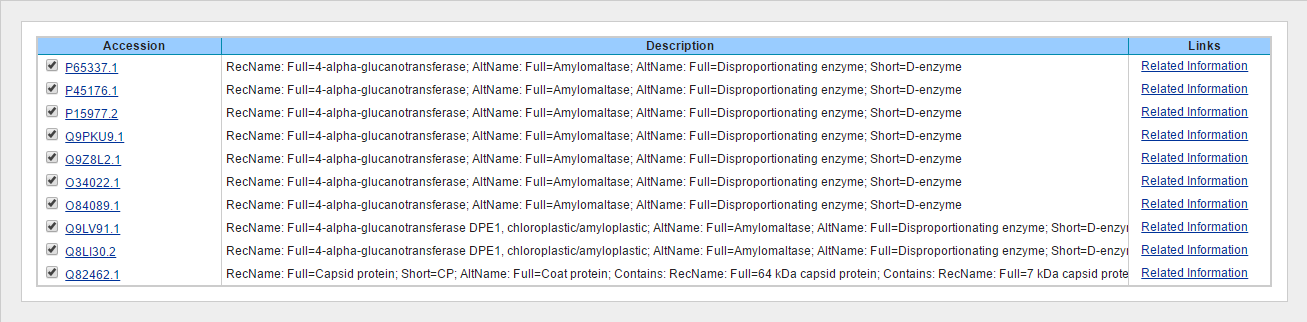
Рисунок 3. Совокупность имён.
Теперь перейдём к оценкам блоков:
Нам нужны отрезки выравнивания нескольких последовательностей с достаточным числом абсолютно консервативных/функционально консервативных позиций.
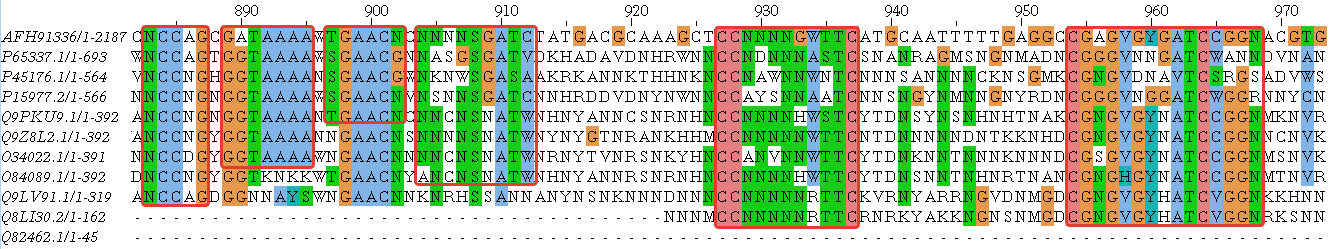
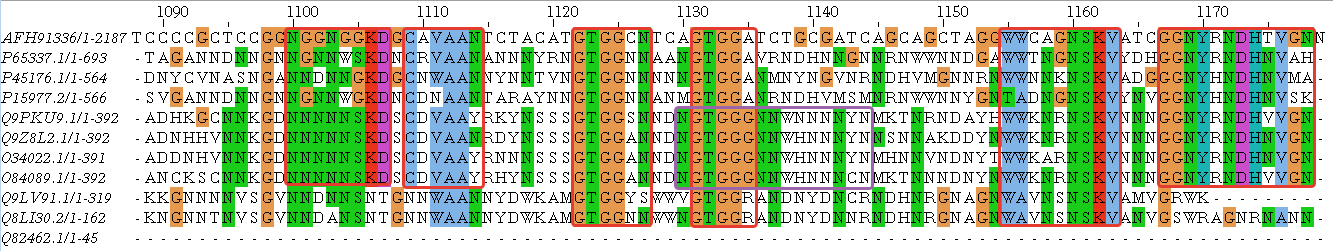
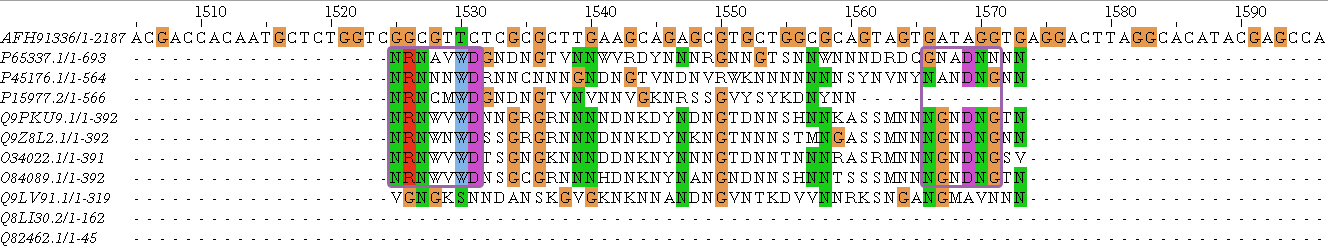
На рисунках ниже блоки выделены рамкой. Красная рамка это гомологияный блок с исследумой последовательностью, когда как фиолетовая - гомология внутри подобранных последоватльностей.
Представленны множественные блоки позволяют пронаблюдать, что выровненные последовательности гомологичны как между собой, так и с первым белком:
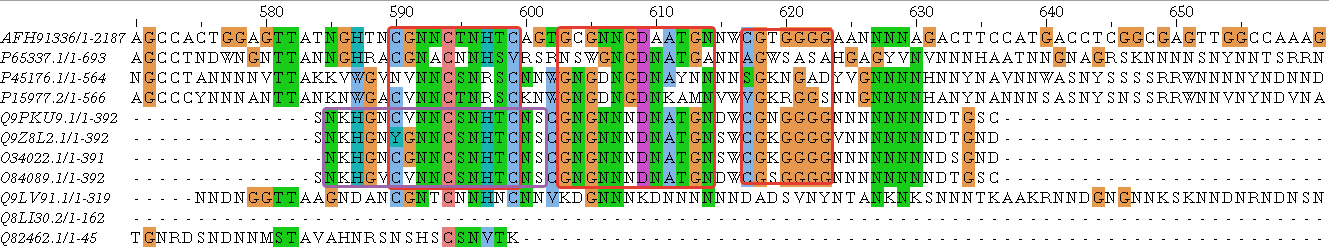 |
| Рисунок 4. |
 |
| Рисунок 5. |
 |
| Рисунок 6. |
 |
| Рисунок 7. |
 |
| Рисунок 8. |
 |
| Рисунок 9. |
Наша главная цель - определить, гомологичны ли белки. Итак, мы нашли блоки более менее для всех последовательнстей, хотя вопросы и вызывают три последние:
Q82462.1|CAPSD_HASV, Q8LI30.2|DPE1_ORYSJ и Q9LV91.1|DPE1_ARATH.
За гомологичность DPE1_ARATH выступает относительно большая длина выравнивания и наличие достаточно крупных блоков для этой последовательности (Рисунки 6,7). Конечно, блоков гомологичности
для неё меньше, однако они присутствуют.
Для DPE1_ORYSJ картина ещё более неутешительная, но всё тот же крупный блок (Рисунок 6) снова включает эту последовательность. Конечно, возможно и совпадение,
однако DPE1_ORYSJ тоже относится к глюко-трансферазам, что скорее говорит о его причастности к этой гомологии.
А вот CAPSD_HASV мало того, что рознится по функциям с остальными белками, так ещё и не участвует в блоках гомологии. Длина выравнивания у него критически мала, попал он в эту выборку явно по ошибке.
| ID/AC | Название белка/Организм | Coverage,% | Identity,% | E-value | Гомологичность | |
|---|---|---|---|---|---|---|
| AlignmentLength/SeqLength | % | |||||
| P65337.1|MALQ_MYCBO | Full=4-alpha-glucanotransferase/Mycobacterium bovis AF2122/97 | 726/724 | 100.15 | 44.353 | 0.0 | Гомологичен |
| P45176.1|MALQ_HAEIN | Full=4-alpha-glucanotransferase/Haemophilus influenzae Rd KW20 | 576/699 | 82.4 | 31.944 | 2.36e-83 | Гомологичен |
| P15977.2|MALQ_ECOLI | Full=4-alpha-glucanotransferase/Escherichia coli K-12 | 578/694 | 83.3 | 30.796 | 1.60e-74 | Гомологичен |
| Q9PKU9.1|MALQ_CHLMU | Full=4-alpha-glucanotransferase/Chlamydia muridarum str.Nigg | 415/527 | 78.7 | 24.819 | 5.17e-28 | Гомологичен |
| Q9Z8L2.1|MALQ_CHLPN | Full=4-alpha-glucanotransferase/Chlamydia pneumoniae | 412/526 | 78.8 | 25.000 | 7.70e-25 | Гомологичен |
| O34022.1|MALQ_CHLCV | Full=4-alpha-glucanotransferase/Chlamydophila caviae GPIC | 414/530 | 78.11 | 24.155 | 2.42e-22 | Гомологичен |
| O84089.1|MALQ_CHLTR | Full=4-alpha-glucanotransferase/Chlamydia trachomatis D/UW-3/CX | 415/527 | 78.7 | 24.096 | 4.41e-22 | Гомологичен |
| Q9LV91.1|DPE1_ARATH | Full=4-alpha-glucanotransferase DPE1, chloroplastic/amyloplastic/Arabidopsis thaliana | 323/576 | 56 | 21.362 | 0.004 | Гомологичен |
| Q8LI30.2|DPE1_ORYSJ | Full=4-alpha-glucanotransferase DPE1, chloroplastic/amyloplastic/Oryza sativa Japonica Group (Japanese rice) | 164/594 | 27.3 | 24.390 | 0.008 | Гомологичен |
| Q82462.1|CAPSD_HASV | Capsid protein/Helicoverpa armigera stunt virus | 45/647 | 728.889 | 1.1 | Негомологичен | |
Таблица 2. Сводная таблица.
Для данного задания мною были выбраны белки Q8N2Q7.2 из прошлого задания и гомологичный ему Q9UKY3.2. Так как случаи гомологий белков в предыдущем задании не слишком интересны, было решено провести исследование, подобное первому заданию, гомолог Q9UKY3.2 был мною выбран как имеющий меньшую длину выравнивания относительно остальных гомологов, ниже представлено выравнивание гомологичного фрагмента с исходным нейролигнином-1, полное выравнивание откроется отдельной страницей при щелчке на картинку, так же ниже прежставлена таблица с основными данными об этих двух белках:
| ID | Название | Длина | Длина выравнивания | Ident | E-value |
|---|---|---|---|---|---|
| Q9UKY3.2 | Putative inactive carboxylesterase 4 | 287 | 230 | 42% | 2e-58 |
| Q8N2Q7.2 | Neuroligin-1 | 840 | - |
Таблица 3. Исследуемые гомологи.
Изменённые параметры выравнивания:

На рисунке достаточно крупные гомологичные участки отмечены латинскими буквами А, B, C. Мелкие связки между участками A и В отмечны отдельными буквами не были Участки А и В это достаточно крупные гомологичные участки примерно равные по длине. Хотелось бы отметить, что гомологичные участки занимают чуть ли не всю длину последовательности Q9UKY3.2, когда как от Q8N2Q7.2 они составляют чуть больше первой трети. Их длины тоже несоразмерны. То есть если участок А лежит у начала обоих последовательностей, то участки В и С для Q9UKY3.2 смещены к концу, что неверно для второй последовательности в силу её длины и того, что участки идут примерно послдовательно. К слову, для Q9UKY3.2 А, В, C и вставки, отмеченные синим, действительно идут последовательно(граничные линии идут очень близко или накладываются друг на драгу), когда как для Q8N2Q7.2 присутствуют многочисленные вставки. Неизменные гомологии должны свидетельствовать о схожих доменах, вставки, очевидно, эволюционно могли образоваться позднее.
На главную страницуВернуться назад
©Solonovich Vera,2017