Cсылка на выравненные последовательности
Cсылка на выравненные последовательности
Cсылка на выравненные последовательности
Для выравнивания были выбраны представители рода Brucella, а именно вторые хромосомы Brucella canis ATCC 23365, Brucella melitensis biovar Abortus 2308 и Brucella pinnipedialis strain 6/566.
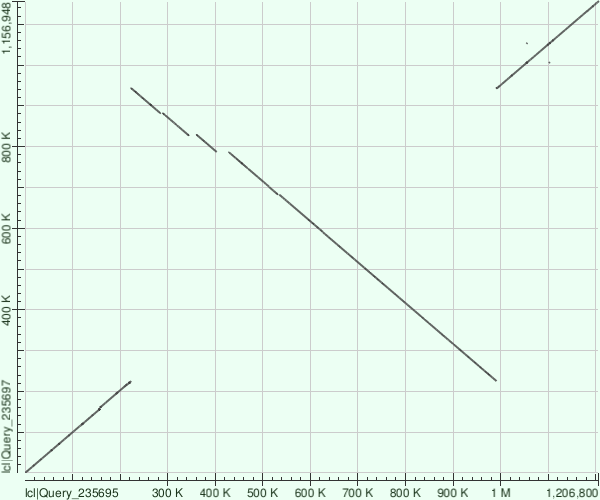
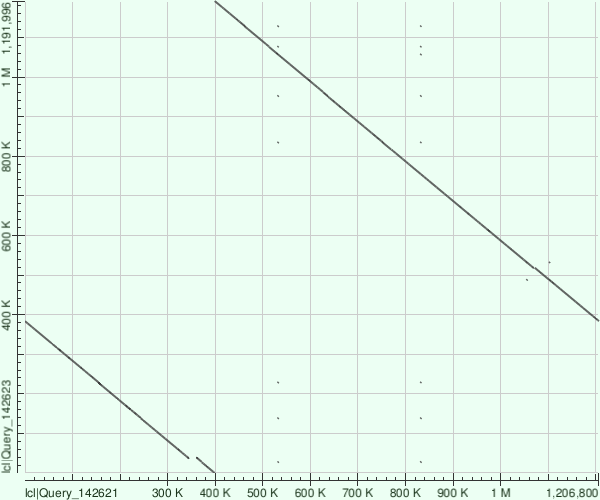
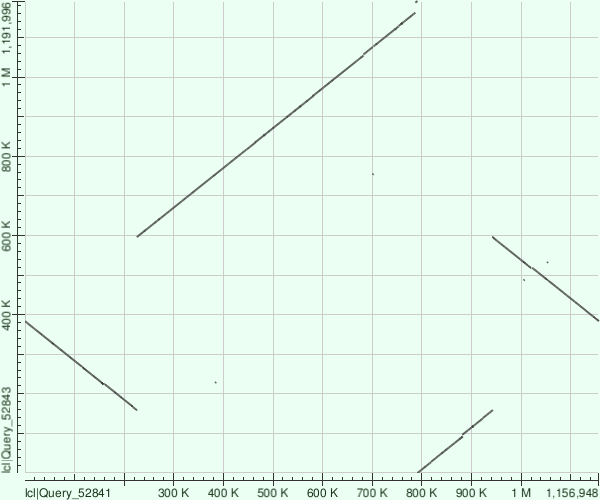
Ниже представлены dot-matrix выравниваний, построенных с помощью blast, с параметрами E-value=0.01, длина слова = 256.
Как вы можете наблюдать, картинки получаются чёткими, характерными, без шумов и с достаточно интересным содержанием, так что качество оправдывает такую большую длину.
По горизонтали на графике представлена Brucella canis, по вертикали - melitensis и pinnipedialis соответственно.Так же было дополнительно построено выравнивание pinnipedialis относительно melitensis(по горизонтали Рисунок 3).
 |  |
| Рисунок 1. Brucella canis/Brucella melitensis
Cсылка на выравненные последовательности |
Рисунок 2. Brucella canis/Brucella pinnipedialis
Cсылка на выравненные последовательности |
 |
|
| Рисунок 3. Brucella meltensis/Brucella pinnipedialis
Cсылка на выравненные последовательности |
|
Идентифицируя карты выравнивния, можно сказать, что:


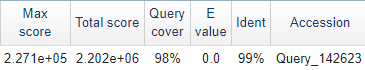
В принипе, в каком-то виде мы получили эти данные раньше, ещё со страницы blast, идентичность и покрытие значатся в колонках Ident и Cover соотвественно:
 |  |
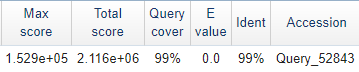 |
| Рисунок 4A. Brucella canis/Brucella melitensis | Рисунок 4B. Brucella canis/Brucella pinnipedialis | Рисунок 4C. Brucella melitensis/Brucella pinnipedialis |
Однако, если проанализировать последовательности в программе NGE, то мы получим более детальную картинку:
Исходный файл с которым работала программа: genomes.tsv
Алгоритм выполнения программ:
| Blocks: | all non-minor
at least 2 fragm. | Exact stem s-blocks | Partial h-blocks | With repeats r-blocks | Rest 1 fragm, not minor | Minor very short m-blocks | Number of fragments | 96 | 61 | 15 | 20 | 8 | 27 | Fragment cover | 3533831/99.38% | 3423033/96.26% | 79819/2.24% | 30979/0.87% | 21176/0.59% | 737/0.02% | Ident(median) | 0.9961 | 0.9968 | 0.9986 | 0.9345 | not shown | 1 | Cover | 1185966/98.19% | 1141866/95.54% | 39940/3.3% | 4160/0.34% | 21176/1.75% | 631/0.05% |
Общая длина послеовательностей:3555744 б.п
Покрытие всех блоков считалось по формуле:
Blocks' lengths: unique + regular = 21647 + 1186126 = 1207773(33.97%)
Таким образом, покрытие блоков не_уникальных, составляет 33.35%
Рассмотрим синтетии, используя данные сконструированных файлов blocks.blocks
и blocks.gbi. Глобальные блоки состоят из последовательно идущих во всех геномах s-блоков, перемежающихся блоками других типов.
NPG насчитал 8 глобальных блоков, схему их расположения можно наблюдать ниже.
| -BRUCA | g3x215380< | i2x4> | g3x462955> | g3x136< | g3x61625> | g3x2143< | i2x4n1< | g3x69512> | g3x175425> | g3x225226< | ||||
| +BRUMM | g3x225226> | i1x3> | g3x462955> | g3x136> | g3x61625> | g3x2143> | i1x4> | g3x69512> | g3x175425> | g3x215380> | ||||
| -BRUPB | g3x175425> | g3x225226< | g3x215380< | i2x4> | g3x462955> | g3x136< | g3x61625> | g3x2143< | i2x4n1< | g3x69512> | ||||
| conservative | g3x462955> | g3x61625> | g3x69512> |
H-блоки или Partial - "полустабильные" блоки собравшие по одному фрагменту из части геномов и u-блоки - уникальные последовательности из одного генома без гомологов среди других геномов, представляют из себя маркёы вставок/делеций, которые мы наблюдали на dot-matrix blast'a.
| BLOCKS | BRUCA | BRUPB | BRUMM |
| h2x25169 | 1 | 1 | 0 |
| h2x5242 | 1 | 1 | 0 |
| h2x2881 | 1 | 0 | 1 |
| h2x2017 | 1 | 1 | 0 |
| h2x886 | 1 | 1 | 0 |
| h2x837 | 1 | 1 | 0 |
| h2x658 | 0 | 1 | 1 |
| h2x414 | 1 | 0 | 1 |
| h2x362 | 0 | 1 | 1 |
| h2x329 | 1 | 0 | 1 |
| h2x285 | 1 | 1 | 0 |
| h2x276 | 1 | 1 | 0 |
| h2x263 | 1 | 0 | 1 |
| h2x189 | 1 | 1 | 0 |
| h2x132 | 1 | 1 | 0 |
| u1x18085 | 1 | 0 | 0 |
| u1x1904 | 0 | 0 | 1 |
| u1x353 | 1 | 0 | 0 |
| u1x310 | 0 | 0 | 1 |
| u1x164 | 0 | 0 | 1 |
| u1x150 | 1 | 0 | 0 |
| u1x108 | 0 | 1 | 0 |
| u1x102 | 0 | 1 | 0 |
Сначала хотелось бы ответить, опираясь на удобные визуальные данные NPG:
В некоторых блоках присутствуют гипотетические белки, хотя аналоги их не определены в других геномах:
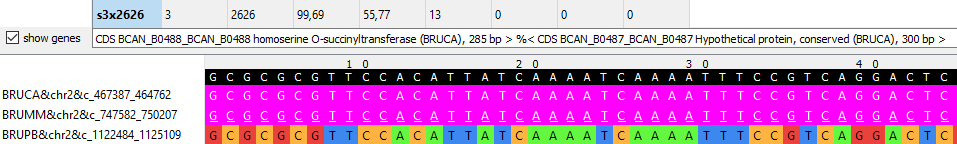 |
| Рисунок 6A О-сукцинил-трансфераза в B.canis, Гликозид-гидролаза в B.mel-s, неопределённый ген/белок в последнем блоке от B.pin-s |
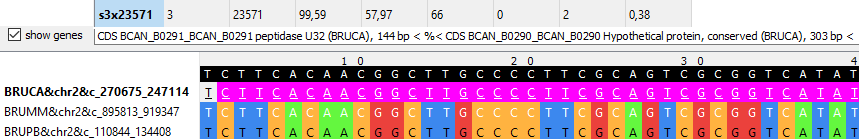 | Рисунок 6B Несоответствия в аннотациях участка BRUCA и BRUMM(B.canis и B.mel-s) |
 | Рисунок 6C Несоответствия в аннотациях на NCBI |
Теперь обратимся к множественным совпадениям - здесь указанный гипотетический белок представляет собой вполне определённые аналоги в других блоках (в большинcтве своём это экстрацеллюлярные связывающие белки, у B.pinnipedialis так же присутствует белок внутренней связывающе-зависимой транспортной системы).
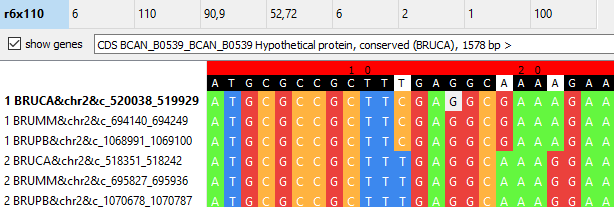 |
| Рисунок 7A,B |
На главную страницуВернуться назад
©Solonovich Vera,2017