Сборка генома de novo.
Чтения
На соответствующем сайте были скачаны чтения в формате fastq проекта по секвенированию бактерии Buchnera aphidicola str. Tuc7 (Acyrthosiphon pisum) с кодом доступа SRR4240379.
Все рабочие файлы хранятся в директории /nfs/srv/databases/ngs/solera/pr14 , архив в том числе был распакован там.
| gunzip SRR4240379.fasrq.gz |
Подготовка чтений
Для сборки генома требовалась предварительная подготовка чтений.
Адапетрные последовательности, дабы удалить их остатки, были скопированы из папки /P/y16/term3/block3/adapters в файл adapters.fasta.
| cat /P/y15/term3/block4/adapters/*.fa > adapters.fasta |
Эти последовательности были удалены из ридов:
|java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240379.fastq SRR4240379_out.fastq ILLUMINACLIP:adapters.fasta:2:7:7
Input Reads: 7400155 Surviving: 7269845 (98,24%) Dropped: 130310 (1,76%) |
А финальным аккордом были удалены плохие буквы с концов чтений, минимальная длина чтений - 30, результат записан в SRR4240379_seqout.fastq.
|java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240379_out.fastq SRR4240379_seqout.fastq TRAILING:20 MINLEN:30
Input Reads: 7269845 Surviving: 6993284 (96,20%) Dropped: 276561 (3,80%) |
Характеристики исходного файла и файлов на выходе вы можете наблюдать ниже:
| Характеристика чтений |
SRR4240379.fastq |
SRR4240379_out.fastq |
SRR4240379_seqout.fastq |
| Число оставшихся чтений |
7400155 |
7269845 |
6993284 |
| Размер файла |
763 862 KB |
750 143 KB |
720 056 KB |
Таблица 1. Сравнение fastq-файлов с чтениями.
Сборка
Первый этап сборки - подготовка k-меров (k=29 и k=25). Она была выполнена с помощью команды velveth пакета velveth, все файлы выхода были помещены в отдельную директорию vel или vel25 соответственно:
| velveth vel 29 -fastq -short SRR4240379_seqout.fastq
| velveth vel25 25 -fastq -short SRR4240379_seqout.fastq |
По итогам работы данной программы в указанных директориях появились файлы Log, Roadmaps и Sequences с логом программы и обработанными последовательностями соответственно.
Затем командой velvetg была произведена сама сборка:
| velvetg vel
Final graph has 2049 nodes and n50 of 47361, max 91528, total 723361, using 0/12549379 reads
| velvetg vel25
Final graph has 4430 nodes and n50 of 5195, max 17284, total 731752, using 0/6993284 reads
|
Эта команда в указанные папки поместила следующие файлы выдачи:
Graph LastGraph PreGraph contigs.fa stats.txt |
Последний содержит информацию о покрытии и длине контигов в k-мерах, причем в столбцах *cov приведено покрытие с учетом частично отличающихся последовательностей, а в столюце *Ocov - только полностью совпадающих. С полной выдачей обоих файлов stats можно ознакомиться, скачав таблицу.
K29 | K25 |
| N50:47361 | N50:5195 |
| Число k-меров:2049 | Число k-меров:4430 |
| Контиги с максимальной длиной |
| ID:5,2,6 | ID:13,18,16 |
| lgth | short1_cov | short1_Ocov | lgth | short1_cov | short1_Ocov |
|---|
| 82103 | 47.938394 | 47.936762 | 17284 | 77.501620 | 77.153321 |
|---|
| 70497 | 49.611544 | 49.607175 | 16308 | 73.563098 | 73.429728 |
| 49941 | 48.604493 | 48.599808 | 13450 | 85.422751 | 85.313606 |
| Контиги с максимальным покрытием |
| ID:50, 68, 813 | ID:2011, 1467, 2485 |
| lgth | short1_cov | short1_Ocov | lgth | short1_cov | short1_Ocov |
|---|
| 64 | 98.015625 | 98.015625 | 3 | 99.333333 | 25.000000 |
| 13 | 94.307692 | 94.307692 | 15 | 99.133333 | 27.000000 |
| 1 | 92.000000 | 45.000000 | 3 | 99.000000 | 24.333333 |
| Контиги с минимальным покрытием |
| ID:925, 928, 648 | ID:3642, 2858, 3610 |
| lgth | short1_cov | short1_Ocov | lgth | short1_cov | short1_Ocov |
|---|
| 6 | 1 | 1 | 10 | 1 | 1 |
| 5 | 1 | 1 | 9
| 1 | 1 |
| 4 | 1 | 1
| 9 | 1 | 1 |
| Медиана значений по контигам |
| 7 | 9,7671315 | 9,026316
| 5 | 5,8 | 5,5 |
| Среднее для значений по контигам |
| 705.9167 | 715,9939837 | 712,0242585
| 165,7047101 | 261,0510877 | 248,1754206 |
Таблица 2. Покрытия, длины контигов
Хотелось бы отметить, что контигов с минимальным покрытием 1 встречается очень много, а в таблице приведены только три первых из них
Анализ
С помощью megablast (порог Evalue 0.01, длина слова 24) были получены выравнивания для трех самых длинных контигов с исследуемой хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253).
Ниже представлены данные по ним - уже знакомым нам из Таблицы 2 контигам 5, 2 и 6, для каждого из которых после иллюстрации бласта следует таблица с описанием:
Контиг 5.
Имеет координаты 451729 - 529004.
Найдено 14 выравниваний, ориентация +. Его расположение в геноме - линейное с несколькими инсерциями.
Один раз опишу это для данного контига, для других аналогично.
На точечной матрице вы можете видеть пробелы в линии - это вставки,
контиг здесь и два других контига в последующих бластах представлены по горизонтальной оси Х,
в местах вставок линия прерывается. Это характерно и для всех остальных контигов:
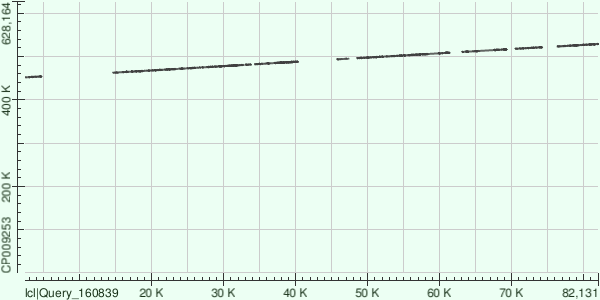 |
| Номер |
E-value |
Начало |
Конец |
Длина |
Совпадения |
Гэпы |
Различия |
| 1 |
0.0 |
467412 |
474667 |
7388 |
5691 (77%) |
208 (3%) |
1489 (20%) |
| 2 |
0.0 |
500370 |
508806 |
8617 |
6516 (76%) |
351 (4%) |
1750 (20%) |
| 3 |
0.0 |
510438 |
516539 |
6234 |
4897 (79%) |
187 (3%) |
1150 (18%) |
| 4 |
0.0 |
523105 |
528679 |
5685 |
4369 (77%) |
207 (3%) |
1109 (20%) |
| 5 |
0.0 |
462496 |
467421 |
5015 |
3861 (77%) |
162 (3%) |
992 (20%) |
| 6 |
0.0 |
481997 |
488106 |
6238 |
4621 (74%) |
308 (5%) |
1309 (21%) |
| 7 |
0.0 |
474844 |
480660 |
5974 |
4431 (74%) |
255 (4%) |
1288 (22%) |
| 8 |
0.0 |
517766 |
521500 |
3783 |
2922 (77%) |
101 (3%) |
760 (20%) |
| 9 |
0.0 |
496111 |
500325 |
4324 |
3255 (75%) |
154 (4%) |
915 (21%) |
| 10 |
0.0 |
451729 |
454069 |
2370 |
1827 (77%) |
55 (2%) |
488 (21%) |
| 11 |
0.0 |
493487 |
494864 |
1384 |
1109 (80%) |
13 (1%) |
262 (19%) |
| 12 |
1e-162 |
480874 |
481545 |
686 |
564 (82%) |
20 (3%) |
102 (15%) |
| 13 |
5e-62 |
528794 |
529004 |
213 |
186 (87%) |
6 (3%) |
21 (10%) |
| 14 |
2e-35 |
495033 |
495148 |
120 |
108 (90%) |
5 (4%) |
7 (6%) |
|
Таблица 3. Информация о бласт и хит-матрица для пятого контига
Контиг 2.
Имеет координаты 528977 - 594099. Ориентация +. Его расположение в геноме - так же линейное с инсерциями, найдено для него 8 выравниваний:
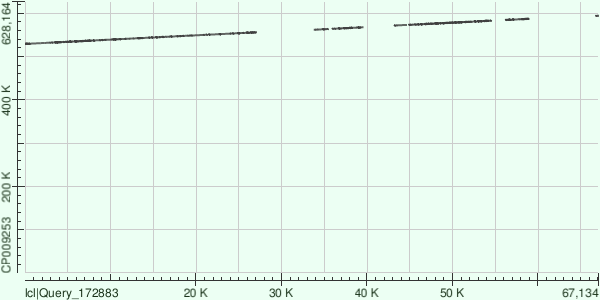 |
| Номер |
E-value |
Начало |
Конец |
Длина |
Совпадения |
Гэпы |
Различия |
| 1 |
0.0 |
528977 |
550219 |
21536 |
17536 (83%) |
539 (3%) |
3461 (16%) |
| 2 |
0.0 |
550361 |
555905 |
5658 |
4575 (81%) |
133 (2%) |
950 (17%) |
| 3 |
0.0 |
573092 |
582686 |
9822 |
7212 (73%) |
461 (5%) |
2149 (22%) |
| 4 |
0.0 |
563837 |
567489 |
3735 |
2912 (78%) |
136 (4%) |
687 (18%) |
| 5 |
0.0 |
571558 |
573007 |
1453 |
1228 (85%) |
6 (0%) |
219 (15%) |
| 6 |
0.0 |
584329 |
587055 |
2777 |
2100 (76%) |
108 (4%) |
569 (20%) |
| 7 |
0.0 |
561741 |
563423 |
1689 |
1333 (79%) |
28 (2%) |
328 (19%) |
| 8 |
4e-72 |
593743 |
594099 |
359 |
289 (81%) |
4 (1%) |
66 (18%) |
|
Таблица 4. Информация о бласт и хит-матрица для второго контига
Контиг 6.
5 выравниваний, так же ориентация +, имеет координаты 127825 - 173180. Его расположение в геноме - линейное с несколькими инсерциями:
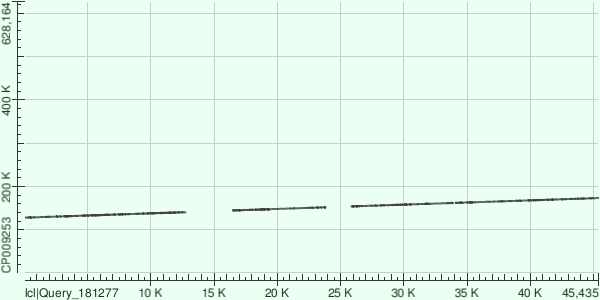 |
| Номер |
E-value |
Начало |
Конец |
Длина |
Совпадения |
Гэпы |
Различия |
| 1 |
0.0 |
127825 |
140555 |
13008 |
9741 (75%) |
544 (4%) |
2723 (21%) |
| 2 |
0.0 |
153752 |
161738 |
8169 |
6347 (78%) |
266 (3%) |
1556 (19%) |
| 3 |
0.0 |
144368 |
151796 |
7536 |
5863 (78%) |
243 (3%) |
1430 (19%) |
| 4 |
0.0 |
161898 |
166752 |
4912 |
3810 (78%) |
108 (2%) |
994 (20%) |
| 5 |
0.0 |
166750 |
173180 |
6517 |
4965 (76%) |
159 (2%) |
1393 (21%) |
|
Таблица 5. Информация о бласт и хит-матрица для шестого контига
Комментарий - координаты контигов считались крайними координатами совпадений в бласте
Сводная таблица:
| ID |
Total score |
Query cover |
E value |
Identity |
Начало |
Конец |
| 5 |
29541 |
69% |
0.0 |
77% |
451729 |
529004 |
| 2 |
31010 |
65% |
0.0 |
81% |
528977 |
594099 |
| 6 |
21303 |
79% |
0.0 |
75% |
127825 |
173180 |
Таблица 6. Сводные данные о результатах megablast.
Вернуться назад
На главную страницу
©Solonovich Vera,2017


